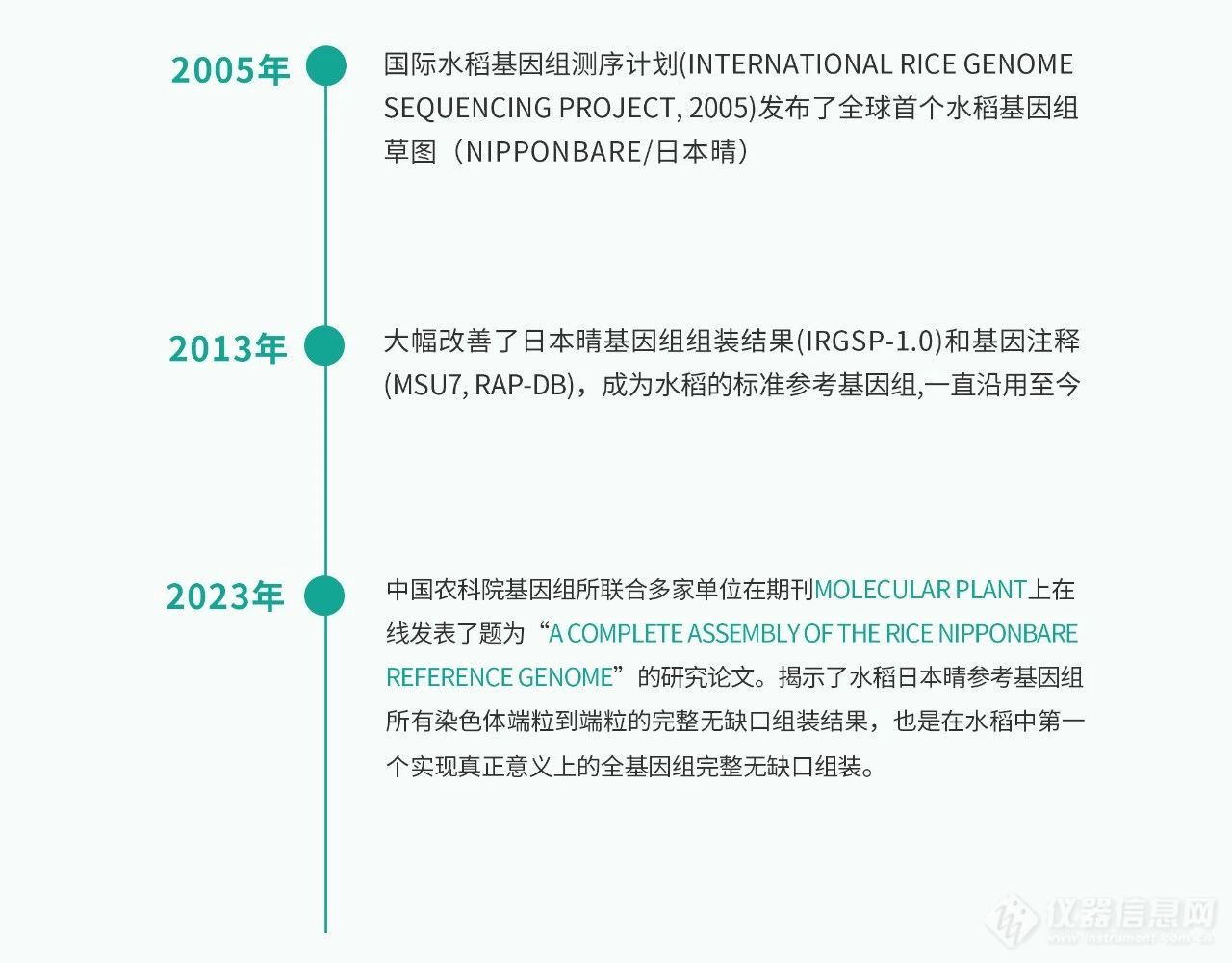
什么是T2T基因组?
T是指端粒,是染色体末端的一段特殊结构,由DNA重复序列和特异结合蛋白所组成的复合体,T2T基因组,指通过ONT ultra-long N50>100Kb结合PacBio HiFi和二代数据进行混合组装,得到的有一条或者多条染色体达到端粒到端粒(Telomere-to-Telomere)的水平基因组,T2T基因组完成图是基因组组装的终极目标。最近几年很多物种的T2T基因组都已发布,比如拟南芥,玉米,西瓜等。
NT ultra-long N50>100Kb结合PacBio HiFi和二代数据进行混合组装,得到的有一条或者多条染色体达到端粒到端粒(Telomere-to-Telomere)的水平基因组,T2T基因组完成图是基因组组装的终极目标。最近几年很多物种的T2T基因组都已发布,比如拟南芥,玉米,西瓜等。
2023年8月,中国农科院深圳农业基因组研究所联合崖州湾国家实验室、中国水稻研究所、中国农科院作物科学研究所和扬州大学等多个单位在国际著名期刊Molecular Plant上在线发表了题为“A complete assembly of the rice Nipponbare reference genome”的研究论文。发表了水稻日本晴参考基因组所有染色体端粒到端粒的完整无缺口组装结果,也是在水稻中第一个实现真正意义上的全基因组完整无缺口组装。

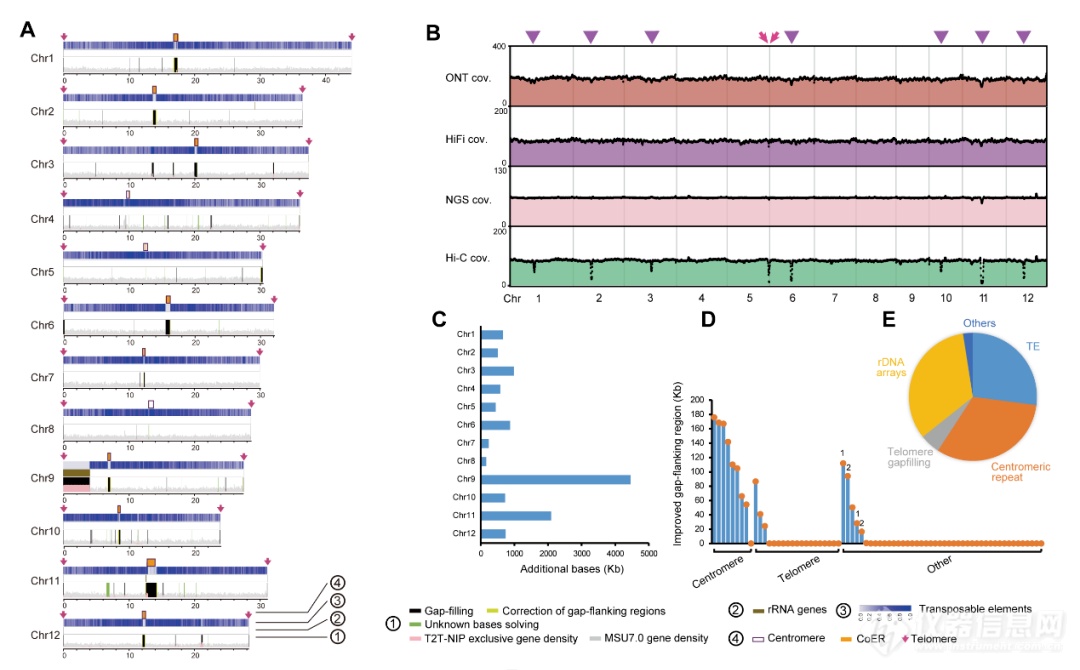
该研究以水稻日本晴为材料,利用约~220X 的超长ONT和~80X 的 Pacbio HiFi 测序数据,结合Hi-C和Illumina测序数据进行辅助调整和矫正,完成了日本晴参考基因组的完整组装(T2T-NIP/AGIS-1.0)。
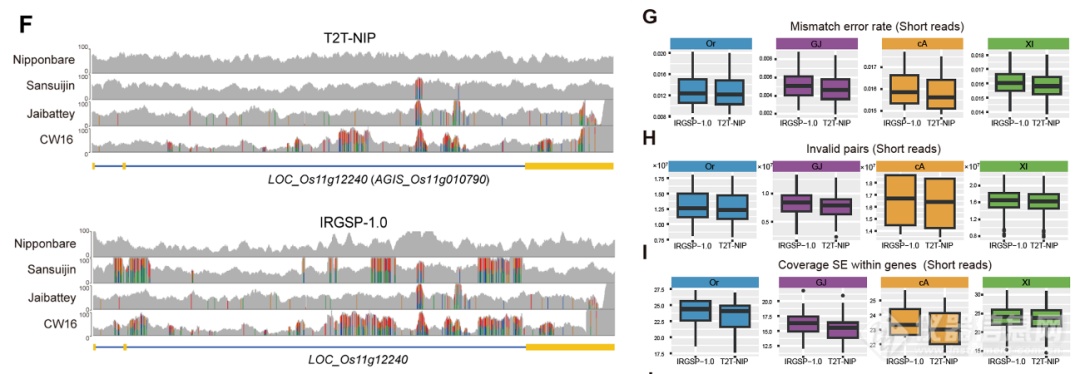
以AGIS-1.0为参考基因组的重测序数据比对
为进一步评估AGIS-1.0的应用潜力,该研究利用水稻多个群体的基因组重测序和ONT数据分别以AGIS-1.0和IRGSP-1.0为参考基因组开展群体变异分析。结果表明,与IRGSP-1.0相比,以AGIS-1.0为参考基因组的reads使用率和比对准确率均有明显提升,尤其是在重复序列导致的多拷贝基因区域的比对质量显著改善,大幅降低了假阳性比对。
该成果是水稻基因组学研究的重大突破,为水稻研究领域提供了新的有力工具和重要理论基础,是水稻基因组组装和基因组学研究的一个重要里程碑。
文章通讯简介:
钱前,中国科学院院士, 博士,研究员。长期从事作物种质遗传资源学研究。在水稻遗传种质资源发掘创新、重要农艺性状解析及分子育种等领域开展了系统深入的研究。先后获国家自然科学一等奖、二等奖、国家科技进步二等奖及省部级科技奖励14项;发表SCI论文321篇,获发明专利44项,主持育成品种10个(6个国审)并在生产上推广应用。
会议邀请丨瀚辰光翼邀您参加第二届精准营养与生命健康创新大会
会议邀请丨瀚辰光翼邀您参加第一届多维组学昆仑论坛
转载|了不起的双流造——打破国外垄断 实现国产化替代
喜讯|瀚辰光翼入选首批“金熊猫未来星”企业
相关产品

关注
拨打电话

留言咨询