Gene-π数字PCR学堂—Drop-off方法检测基因缺失篇
数字 PCR (dPCR)可以提供比传统qPCR更高的精准度和灵敏度,尤其是在肿瘤学应用中,dPCR能精准且可靠地检测到较低浓度样本中的稀有突变等。
目前,使用Drop-off方法可以同时检测基因组内发生的多个序列插入、缺失或碱基突变,最大限度地提高单个检测中样本的数据输出,节省时间和检测成本。本文以naica®微滴芯片数字PCR平台为基础,对使用Drop-off方法检测基因缺失进行阐述。
首先,为大家介绍一下Drop-off方法原理:
☑ Drop-off是指利用探针在一个反应中检测基因组序列的同源基因突变(包括核苷酸的缺失,插入和替换)。
☑ Drop-off方法包括两个TaqMan 探针:一个与野生型序列互补而与突变位点不发生互补的Drop-off探针和一个与突变型和野生型基因都互补的Reference探针(如下图A)。
☑ 当野生型等位基因存在时,Drop-off探针和Reference探针都将与目标序列杂交,产生双重阳性信号(如下图B,蓝色群)。相反,如果存在突变的等位基因,即使一个核苷酸的突变也会阻止Drop-off探针与之杂交,因此只有Reference探针与目标基因结合,从而得到一个阳性信号(如下图B,绿色群)。
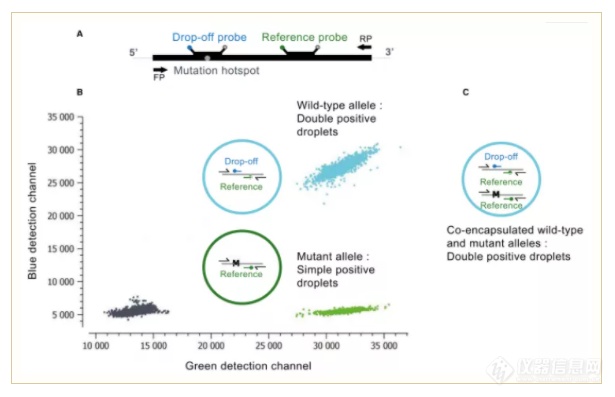
A.Drop-off和Reference探针及引物在目标基因上的示意图(FP:上游引物,RP:下游引物)
B. Crystal Miner 2D图中显示来自双阳性野生型等位基因的荧光簇(天蓝色:蓝色和绿色)和突变等位基因的荧光簇(绿色)及阴性微滴簇(黑色)。Drop-Off探针仅和野生型序列互补,而跳过突变序列(图B,纵坐标的蓝色通道);Reference探针与突变和野生型等位基因进行互补(图B中横坐标的绿色通道)。
野生型浓度CWT可以直接从双阳性微滴中PWT的比例得出。但是,Drop-off突变体浓度Cmut必须从确信包含突变等位基因的微滴比例Pmut中得出。
由于野生型和突变等位基因可能被分配到同一个微滴里面,因此采用双阳性微滴对突变体进行定量是不准确的,只能通过排除双阳性微滴来确定Pmut(将突变阳性群体定义为对Reference探针呈阳性但对Drop-off探针呈阴性的微滴)。
上文已对检测原理和检测方法进行了阐述,那么如何对Drop-off检测结果进行分析呢?
1、使用高灵敏naica®微滴芯片式数字PCR系统的蓝色和绿色两个通道对突变等位基因(MAF)进行绝对定量。
2、使用Crystal Miner分析软件进行量化分析计算。
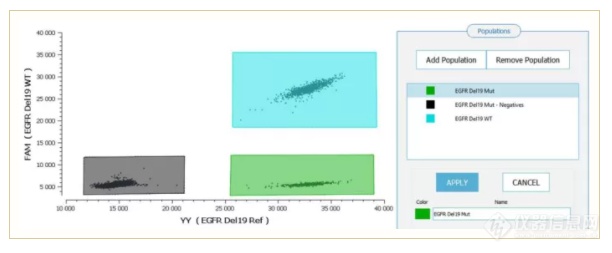
A 、通过自主圈群功能选中三种微滴群体并进行命名-突变型阳性(绿色),双阴性(黑色),野生型双阳性(天蓝色:蓝色和绿色)。
B、通过naica Crystal Miner软件获得Drop-off突变型的浓度Cmut(绿色),以及野生型浓度CWT(天蓝色)。
C、计算MAF Drop-off突变,即MAF Drop-off=Cmut/(Cwt+Cmut)。(如想了解关于Cmut和Cwt详细计算方法,可点击链接:
https://www.gene-pi.com/tutorial/drop-off-assay/)
小提示:
如果不排除双阳性的微滴并将其归为阴性微滴中, Drop-off突变体的MAF则会被低估,对于任何Cmut<100cp/µL,这一系数几乎是恒定的(例如在样品中CWT为500 cp/µL时,低估了25%)。保留双阳性微滴既不会降低测得的Drop-off突变体浓度的相对不确定性,也不会降低LOD。
应用亮点:
☑ 通过使用naica Crystal Miner圈群功能,可以对Drop-off突变进行精准定量。
☑ 排除模棱两可的微滴,在不降低测量精度的前提下避免了低估Drop-off突变体的浓度。
☑ 本次检测使用了naica®微滴芯片数字PCR系统的蓝绿两个通道,第三个检测通道还可以添加一个内部对照或同时量化一个MAF点突变。
如想学习更多Drop-off知识,可以通过以下方式获取Drop-off计算方法:
http://www.cycloudbio.com/product/36748.html
naica®微滴芯片数字PCR系统
法国Stilla Technologies公司的naica®微滴芯片数字PCR系统在进行核酸检测时具有独特的优势。该系统利用cutting-edge微流体创新型芯片—Sapphire芯片(或高通量Opal芯片)作为数字PCR过程的耗材。样品通过毛细通道网格以30,000个微滴的形式进入2D芯片中。3色荧光检测仪器,整个流程只需要2.5小时,并可进行数据的质控和结果追溯分析,获得的数据真实可靠。
naica®六通道微滴芯片数字PCR系统
法国Stilla Technologies公司naica®六通道微滴芯片数字PCR系统,源于Crystal微滴芯片式数字PCR技术,自动化微滴生成和扩增,每个样本孔可实现6荧光通道的检测,智能化识别微滴并进行质控,3小时内即可获得至少6个靶标基因的绝对拷贝数浓度。

来源于:北京深蓝云生物科技有限公司
热门评论
最新资讯
厂商动态
新闻专题
更多推荐