一文知晓CRISPER基因编辑早期脱靶克隆快速筛选的多重naica®数字PCR方法
使用序列特异性的CAS内切酶进行精确性敲除,敲入,替换等已成为一种常用的分子生物学手段。但是,为了识别所需的基因修饰,排除脱靶克隆,仍然需要筛选几百个单克隆细胞系。而大量克隆的维护和筛选是一个费力、耗时、容易出错的过程。
欧洲分子生物学实验室(EMBL)研究人员Moritz Kueblbeck, Andrea Callegari等分享了一种快速验证基因编辑效果的实验方案。该方案使用naica®微滴芯片数字PCR系统(Crystal Digital PCR™)实现了一次三色分析就可完成目标拷贝数的评估和脱靶事件的检测。且基于数字PCR (dPCR)的高敏感性,无需大量的样品,因此,在早期就可以完成筛选试验。Crystal Digital PCR™一次实验3个小时内就可完成,对于编辑错误的克隆当天就可终止培养。那么这个实验具体是怎么设计的?就让我们一起来看看吧。
应用亮点:
▶ naica®微滴芯片数字PCR系统一次三色分析同时完成目标拷贝数的评估和脱靶事件的检测,是非常强大的绝对定量工具。
▶ naica®微滴芯片数字PCR系统对长插入片段(如mEGFP)拷贝数提供稳健的检测方法。
▶ naica®微滴芯片数字PCR系统只需少量生物样本,可以在早期完成检测,快速完成CRISPR筛选,节省时间。
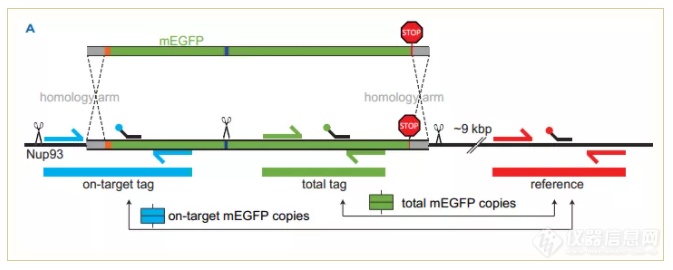
Single-step 3-color Crystal Digital PCR™实验设计:
该实验共设计了三个探针:
1.“total tag”(HEX基团标记)检测mEGFP转基因;
2.“on-target tag”(FAM基团标记)检测内源NUP93编辑位点的最后一个外显子与整合的mEGFP转基因(标签)的5'序列之间的连接;
3.reference(CY5基团标记)作为内参,对“total tag”和“on-target tag”进行归一化。“total tag”归一化获得整合的mEGFP拷贝数和“on-target tag”获得靶标上整合的mEGFP拷贝数。
实验使用U-2 OS细胞作为基因编辑对象,其具有3个NUP93等位基因。
结果分析:
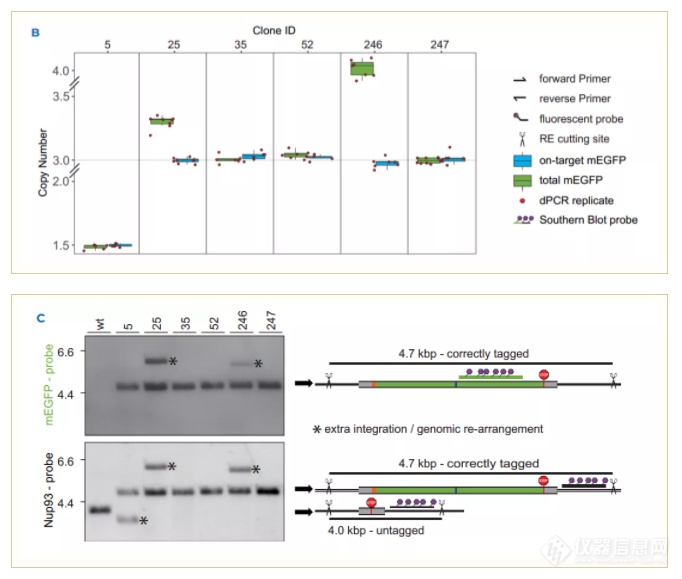
如图B所示,对多个U-2 OS细胞系的基因编辑效果进行分析,naica®微滴芯片式数字PCR系统结果显示克隆35,52和247的U-2 OS细胞mEGFP总拷贝数和靶标拷贝数都为3,这表明这三个细胞系的所有3个NUP93等位基因都成功编辑,没有脱靶,这个结果与检测金标准的Southern blot的结果一致(图C),而克隆5虽然mEGFP总拷贝数和靶标拷贝数一致,但与NUP93位点的预期数量不匹配。这表明其可能发生基因组的重排,而Southern blot结果也验证了这一现象,其出现了一个小于正常NUP93的拷贝条带。对于克隆25和246,虽然其靶标拷贝数符合预期,但是其总拷贝数大于3,这表明其可能存在脱靶整合或者不完全HDR(homology-directed repair)现象,而Southern blot结果显示其存在一个过大的整合条带,表示其可能存在基因重排。
上述这些结果表明基于naica®微滴芯片式数字PCR系统(3-color Crystal Digital PCR™)的基因编辑脱靶检测的三色数字PCR检测方法,是一种快速简便的一步检测方法,其结果与耗时更长的Southern blot技术完全一致,可以用来快速评估基因编辑效果,筛选适合的基因编辑细胞株系。
来源于:北京深蓝云生物科技有限公司
热门评论
最新资讯
厂商动态
新闻专题
更多推荐

















