测序仪笔记分享(万字长文,建议收藏)
导读:本文系统总结常见测序仪的原理和主机结构,并介绍相关测序原理和发展史。测序仪笔记分享,万字长文,建议您阅读并收藏。
一. 测序仪对比
测序技术 |
代表仪器 |
读长 |
通量 |
准确度 |
成本 |
Sanger法 |
ABI 3730xl DNA Analyzer |
500-800bp |
0.096Gbp/天 |
>99.99% |
0.24美分/bp |
Illumina |
HiSeq X Ten System |
150bp |
1800Gbp/运行 |
>99.9% |
0.01美分/bp |
MGISEQ-2000 |
200bp(单端)或2×150bp(双端) |
60Gbp/运行 |
>99.9% |
0.015美元/bp |
|
Roche 454 |
GS FLX+ System |
700bp |
0.7Gbp/运行 |
>99.9% |
0.02美元/bp |
ABI SOLiD |
SOLiD System 5500xl |
75bp |
120Gbp/运行 |
>99.94% |
0.13美分/bp |
PacBio |
Sequel II System |
>10kb |
60Gbp/运行 |
>99% |
0.15美元/bp |
Nanopore |
MinION Device |
>100kb |
30Gbp/运行 |
>90% |
0.02美元/bp |
Helicos |
HeliScope Single Molecule Sequencer |
25-50bp |
28Gbp/运行 |
<80% |
未知 |
1. ABI 3730xl DNA Analyzer

图源自thermofisher官网
1.1. 相关原理
· DNA测序:基于Sanger法的原理,利用DNA聚合酶在体外DNA复制过程中随机掺入带有荧光标记和终止子的双脱氧核苷酸(ddNTPs),从而得到不同长度的DNA片段。这些片段经过电泳分离后,通过激光激发和CCD检测,得到每个碱基发出的荧光信号,从而确定DNA的碱基序列。
· 片段分析:基于荧光检测的原理,利用不同颜色的荧光染料标记不同长度或类型的DNA片段,如微卫星、SNP、AFLP等。这些片段经过电泳分离后,通过激光激发和CCD检测,得到每个片段发出的荧光信号,从而确定片段的大小或等位基因。
1.2. 主要组成
ABI 3730xl DNA Analyzer仪器是一种高通量的DNA测序和片段分析的平台,它可以同时使用48或96根毛细管进行电泳分离和荧光检测。
· 测序仪主机:包含电泳系统、自动进样系统、激光系统、光学系统、温控系统、聚合物输送系统等多个模块,用于控制仪器的运行和数据的采集。
· 计算机工作站:预装用于仪器控制、数据收集和样品文件自动分析的软件,如Data Collection Software、Sequencing Analysis Software、SeqScape Software、GeneMapper Software等。
· 毛细管阵列:提供预组装的48根或96根毛细管阵列,它们与业界标准的96孔和384孔板配合使用。毛细管为内部无涂层毛细管,可提供300次的运行质保。
· DNA测序试剂和耗材:包括BigDye Terminator循环测序试剂盒、GeneScan分子量标准品、片段分析标准品、POP-7聚合物分离胶等。
1.3. 主机模块
· 电泳系统:负责将DNA片段在毛细管中进行电泳分离,根据不同长度的DNA片段在电场中的迁移速度不同,将它们按照从小到大的顺序排列。电泳系统由高压电源、电泳缓冲液、毛细管阵列等组成。
o 高压电源:提供高达30kV的电压,使DNA片段在电场中迁移。
o 电泳缓冲液:提供电导性和pH稳定性,使DNA片段在毛细管中顺利运行。
o 毛细管阵列:提供预组装的48根或96根毛细管,它们与业界标准的96孔和384孔板配合使用。毛细管为内部无涂层毛细管,可提供300次的运行质保。
· 自动进样系统:负责将样品从96孔或384孔板中自动吸取,并注入到毛细管阵列中。自动进样系统由进样针、进样泵、进样阀等组成。
o 进样针:用于从样品板中吸取样品,并通过进样阀将样品注入到毛细管中。
o 进样泵:用于控制进样针的吸取和释放动作,以及进样量的大小。
o 进样阀:用于控制进样针与毛细管之间的连接和断开,以及进样时间的长短。
· 激光系统:负责将激光光束照射到毛细管阵列的出口处,激发荧光信号。激光系统由激光器、光纤、光学开关等组成。
o 激光器:提供单波长、505nm、固态、长寿命的激光光源,用于激发荧光染料。
o 光纤:用于将激光光束从激光器传输到毛细管阵列上。
o 光学开关:用于控制激光光束的开启和关闭,以及激光功率的大小。
· 光学系统:负责将荧光信号收集并转换为电信号。光学系统由滤光片、透镜、CCD相机等组成。
o 滤光片:用于选择不同颜色的荧光信号,并过滤掉背景噪声。
o 透镜:用于聚焦和放大荧光信号,并将其投射到CCD相机上。
o CCD相机:用于将荧光信号转换为数字化的电信号,并传输给计算机工作站进行数据采集和分析。
· 温控系统:负责控制仪器的温度,保证测序的稳定性和可靠性。温控系统由温度传感器、风扇、加热器等组成。
o 温度传感器:用于监测仪器内部和外部的温度,并反馈给温控器进行调节。
o 风扇:用于散热和通风,维持仪器的适宜温度。
o 加热器:用于加热和保温,防止仪器的过冷。
· 聚合物输送系统:负责将聚合物分离胶从储存瓶输送到毛细管阵列中,作为电泳介质。聚合物输送系统由压力罐、气压调节器、流量计等组成。
o 压力罐:用于储存聚合物分离胶,并提供一定的压力,使聚合物分离胶能够流动。
o 气压调节器:用于控制压力罐的气压,以及聚合物分离胶的流速。
o 流量计:用于测量聚合物分离胶的流量,以及毛细管中的胶量。
2. HiSeq X Ten System

图源自Illumina官网
HiSeq X Ten System是Illumina公司的产品。Illumina是一家生物技术公司,它的测序仪是基于桥式PCR和荧光检测的技术,也是目前最流行的二代测序平台之一。它的测序仪有多个系列,如NovaSeq、HiSeq、MiSeq、MiniSeq等,它们的核心技术原理是相同的,但在通量、读长、准确度、成本等方面有所不同。
2.1. 相关原理
· 文库构建:将待测DNA打断成小片段,并在两端加上特殊的接头(Adaptor),这些接头包含与流通池表面探针互补的序列(P5/P7)、用于区分不同文库的索引(Index)、以及用于测序引物结合的序列(Rd1 SP/Rd2 SP)。文库构建后需要进行质量检测和定量。
· 聚集体生成:将文库DNA片段注入到流通池中,并与表面探针杂交结合。然后进行桥式PCR扩增,使每个DNA片段形成一个聚集体。聚集体生成后需要进行温度变化和化学处理,使其单链化并去除P5端的DNA链,只留下P7端的DNA单链。
· 边合成边测序:将带有荧光染料和可逆终止子的四种dNTPs逐一加入到流通池中,并利用DNA聚合酶将它们连接到聚集体的DNA链上。每次只能加入一个碱基,然后用激光激发荧光信号,并用CCD相机记录每个聚集体发出的荧光信号,从而确定碱基序列。然后用化学剂去除荧光染料和可逆终止子,使下一个碱基可以继续加入。重复这个过程,直到完成所有的测序循环。
· 数据分析:将CCD相机收集到的荧光信号转换为原始数据(BCL文件),并进行质量控制和过滤,去除低质量的聚集体和信号。然后根据索引将不同文库的数据分离,并进行碱基识别(Base calling),将荧光信号转换为碱基序列(FASTQ文件)。最后根据不同的测序目的,进行后续的数据分析,如比对、变异检测、表达量计算等。
2.2. 主要组成
· 流通池(Flow cell):是一个微型的玻璃芯片,它的表面覆盖了数亿个固定在不同位置的寡核苷酸探针,这些探针与文库DNA片段的接头互补,可以通过杂交结合。流通池内部有多个通道,每个通道可以进行不同的测序反应。
· 聚集体(Cluster):是指通过桥式PCR在流通池表面扩增形成的由相同DNA片段组成的簇,每个聚集体可以发出荧光信号,从而被检测为一个读长(Read)。聚集体的密度和质量会影响测序的效率和准确度。
· 荧光染料(Fluorescent dye):是指用于标记不同碱基的四种荧光分子,它们分别对应A、T、C、G四种碱基,并发出不同颜色的光。荧光染料还带有可逆终止子,可以控制每次只加入一个碱基。
· 激光器(Laser):是指用于激发荧光染料发光的光源,它可以提供单波长、固态、长寿命的激光光束。
· CCD相机(CCD camera):是指用于捕捉和记录荧光信号的设备,它可以将荧光信号转换为数字化的电信号,并传输给计算机进行数据分析。
· 计算机系统(Computer system):是指用于控制测序仪运行和处理数据的设备,它预装了用于仪器控制、数据收集和样品文件自动分析的软件,如BaseSpace Sequence Hub、Sequencing Analysis Software等。
3. MGISEQ-2000

图源自华大智造官网
MGISEQ-2000测序仪是一种基于荧光检测的第二代测序技术,可以实现高通量、高精度、低成本的基因组测序。
3.1. 相关原理
o DNA测序:基于双端测序的原理,利用DNA聚合酶在体外DNA复制过程中随机掺入带有荧光标记和终止子的双脱氧核苷酸(ddNTPs),从而得到不同长度的DNA片段。这些片段经过桥式扩增后,形成单分子簇,然后通过四色荧光检测,得到每个碱基发出的荧光信号,从而确定DNA的碱基序列。
o 片段分析:基于荧光检测的原理,利用不同颜色的荧光染料标记不同长度或类型的DNA片段,如微卫星、SNP、AFLP等。这些片段经过桥式扩增后,形成单分子簇,然后通过四色荧光检测,得到每个片段发出的荧光信号,从而确定片段的大小或等位基因。
3.2. 主要组成
o 测序仪主机:包含流体控制系统、温控系统、激光系统、光学系统、信号采集系统等多个模块,用于控制仪器的运行和数据的采集。
§ 流体控制系统:负责控制样品和试剂的输送,以及测序反应的进行。流体控制系统由进样针、进样泵、进样阀等组成。
§ 进样针:用于从样品板中吸取样品,并通过进样阀将样品注入到芯片上。
§ 进样泵:用于控制进样针的吸取和释放动作,以及进样量的大小。
§ 进样阀:用于控制进样针与芯片之间的连接和断开,以及进样时间的长短。
§ 温控系统:负责控制仪器和芯片的温度,保证测序的稳定性和可靠性。温控系统由温度传感器、风扇、加热器等组成。
§ 温度传感器:用于监测仪器和芯片内部和外部的温度,并反馈给温控器进行调节。
§ 风扇:用于散热和通风,维持仪器和芯片的适宜温度。
§ 加热器:用于加热和保温,防止仪器和芯片的过冷。
§ 激光系统:负责将激光光束照射到芯片上,激发荧光信号。激光系统由激光器、光纤、光学开关等组成。
§ 激光器:提供单波长、532nm、固态、长寿命的激光光源,用于激发荧光染料。
§ 光纤:用于将激光光束从激光器传输到芯片上。
§ 光学开关:用于控制激光光束的开启和关闭,以及激光功率的大小。
§ 光学系统:负责将荧光信号收集并转换为电信号。光学系统由滤光片、透镜、CCD相机等组成。
§ 滤光片:用于选择不同颜色的荧光信号,并过滤掉背景噪声。
§ 透镜:用于聚焦和放大荧光信号,并将其投射到CCD相机上。
§ CCD相机:用于将荧光信号转换为数字化的电信号,并传输给计算机工作站进行数据采集和分析。
§ 信号采集系统:负责对数字化的电信号进行滤波、校准、分段、碱基识别等处理,最终生成测序结果。信号采集系统由数据采集卡、数据处理软件等组成。
§ 数据采集卡:用于将CCD相机传输的电信号接收并转换为数字信号,以及进行一定的滤波和校准处理。
§ 数据处理软件:用于对数字信号进行进一步的分段、碱基识别、质量评估等处理,以及生成测序结果文件。
o 计算机工作站:预装用于仪器控制、数据收集和样品文件自动分析的软件。
o 芯片:芯片是MGISEQ-2000测序仪的核心部件,它是一种微流控芯片,上面有数百万个微孔,每个微孔都可以进行单分子簇测序,实现高通量的数据产出。芯片有不同的规格和类型,如单端测序芯片、双端测序芯片、片段分析芯片等,可以根据不同的需求选择合适的芯片。
4. GS FLX+ System
图源自罗氏官网
GS FLX+ System测序仪是一种基于焦磷酸测序(Pyrosequencing)技术的二代测序平台,它可以提供高通量、高准确度和超长读长的DNA测序服务。
4.1. 相关原理
· 文库构建:将待测DNA打断成小片段,并在两端加上特殊的接头(Adaptor),这些接头包含与DNA捕获珠表面探针互补的序列(A/B)、以及用于测序引物结合的序列(P1/P2)。文库构建后需要进行质量检测和定量。
· 乳液PCR:将文库DNA片段与DNA捕获珠混合,并加入油相形成乳液滴。每个乳液滴中只包含一个DNA捕获珠和一个文库DNA片段。然后进行PCR扩增,使每个DNA捕获珠上形成一个单分子聚集体。乳液PCR后需要进行破乳液和洗涤处理,去除多余的油相和PCR试剂。
· PTP装载:将经过乳液PCR处理后的DNA捕获珠注入到PTP中,并使每个微孔中只有一个DNA捕获珠。然后进行温度变化和化学处理,使聚集体单链化并去除A端的DNA链,只留下B端的DNA单链。
· 边合成边测序:将带有荧光染料和可逆终止子的四种dNTPs逐一加入到PTP中,并利用DNA聚合酶将它们连接到聚集体的DNA链上。每次只能加入一个碱基,然后用激光激发荧光信号,并用CCD相机记录每个聚集体发出的荧光信号,从而确定碱基序列。然后用化学剂去除荧光染料和可逆终止子,使下一个碱基可以继续加入。重复这个过程,直到完成所有的测序循环。
· 数据分析:将CCD相机收集到的荧光信号转换为原始数据(SFF文件),并进行质量控制和过滤,去除低质量的聚集体和信号。然后进行碱基识别(Base calling),将荧光信号转换为碱基序列(FASTA/FASTQ文件)。最后根据不同的测序目的,进行后续的数据分析,如比对、变异检测、表达量计算等。
4.2. 主要组成
· 测序仪主机:包含电泳系统、自动进样系统、激光系统、光学系统、温控系统、聚合物输送系统等多个模块,用于控制仪器的运行和数据的采集。
· 计算机工作站:预装用于仪器控制、数据收集和样品文件自动分析的软件,如Data Collection Software、Sequencing Analysis Software等。
· PicoTiterPlate(PTP):是一个微型的塑料板,它的表面覆盖了数百万个微孔,每个微孔可以容纳一个DNA捕获珠(DNA Capture Bead),并进行单分子测序反应。
· DNA捕获珠(DNA Capture Bead):是一种直径约28微米的磁性珠子,它的表面覆盖了数千个固定在不同位置的寡核苷酸探针,这些探针与文库DNA片段的接头互补,可以通过乳液PCR(Emulsion PCR)扩增形成单分子聚集体(Single Molecule Cluster)。
· 荧光染料(Fluorescent dye):是指用于标记不同碱基的四种荧光分子,它们分别对应A、T、C、G四种碱基,并发出不同颜色的光。荧光染料还带有可逆终止子,可以控制每次只加入一个碱基。
· 激光器(Laser):是指用于激发荧光染料发光的光源,它可以提供单波长、固态、长寿命的激光光束。
· CCD相机(CCD camera):是指用于捕捉和记录荧光信号的设备,它可以将荧光信号转换为数字化的电信号,并传输给计算机进行数据分析。
4.3. 主机组成
· 电泳系统:是指用于将带有荧光染料和可逆终止子的四种dNTPs逐一加入到PTP中,并利用DNA聚合酶将它们连接到聚集体的DNA链上的系统。每次只能加入一个碱基,然后用激光激发荧光信号,并用CCD相机记录每个聚集体发出的荧光信号,从而确定碱基序列。
· 自动进样系统:是指用于将经过乳液PCR处理后的DNA捕获珠注入到PTP中,并使每个微孔中只有一个DNA捕获珠的系统。然后进行温度变化和化学处理,使聚集体单链化并去除A端的DNA链,只留下B端的DNA单链。
· 激光系统:是指用于激发荧光染料发光的光源,它可以提供单波长、固态、长寿命的激光光束。
· 光学系统:是指用于捕捉和记录荧光信号的设备,它可以将荧光信号转换为数字化的电信号,并传输给计算机进行数据分析。
· 温控系统:是指用于控制PTP板和反应液的温度,以保证测序反应的稳定性和效率的系统。
· 聚合物输送系统:是指用于将不同类型和浓度的聚合物溶液输送到PTP板中,以提供不同阶段所需的反应条件和试剂的系统。
5. SOLiD System 5500xl

图源自thermofisher官网
SOLiD System 5500xl测序仪是一种基于连接法测序(Sequencing by Ligation)技术的二代测序平台,它可以提供高通量、高准确度和中等读长的DNA测序服务。
5.1. 相关原理
· 文库构建:将待测DNA打断成小片段,并在两端加上特殊的接头(Adaptor),这些接头包含与DNA捕获珠表面探针互补的序列(P1/P2)、以及用于测序引物结合的序列(Rd1 SP/Rd2 SP)。文库构建后需要进行质量检测和定量。
· 乳液PCR:将文库DNA片段与DNA捕获珠混合,并加入油相形成乳液滴。每个乳液滴中只包含一个DNA捕获珠和一个文库DNA片段。然后进行PCR扩增,使每个DNA捕获珠上形成一个单分子聚集体。乳液PCR后需要进行破乳液和洗涤处理,去除多余的油相和PCR试剂。
· FlowChip装载:将经过乳液PCR处理后的DNA捕获珠注入到FlowChip中,并使每个微孔中只有一个DNA捕获珠。然后进行温度变化和化学处理,使聚集体单链化并去除P1端的DNA链,只留下P2端的DNA单链。
· 边连接边测序:将带有荧光探针和可逆终止子的四种dNTPs逐一加入到FlowChip中,并利用DNA连接酶将它们连接到聚集体的DNA链上。每次只能加入一个碱基对,然后用激光激发荧光信号,并用CCD相机记录每个聚集体发出的荧光信号,从而确定碱基对序列。然后用化学剂去除荧光探针和可逆终止子,使下一个碱基对可以继续加入。重复这个过程,直到完成所有的测序循环。
· 数据分析:将CCD相机收集到的荧光信号转换为原始数据(BCL文件),并进行质量控制和过滤,去除低质量的聚集体和信号。然后进行碱基识别(Base calling),将荧光信号转换为碱基对序列(FASTQ文件)。最后根据不同的测序目的,进行后续的数据分析,如比对、变异检测、表达量计算等。
5.2. 主要组成
· 测序仪主机:包含电泳系统、自动进样系统、激光系统、光学系统、温控系统、聚合物输送系统等多个模块,用于控制仪器的运行和数据的采集。
· 计算机工作站:预装用于仪器控制、数据收集和样品文件自动分析的软件,如Data Collection Software、Sequencing Analysis Software等。
· FlowChip:是一个微型的玻璃芯片,它的表面覆盖了数百万个微孔,每个微孔可以容纳一个DNA捕获珠(DNA Capture Bead),并进行单分子测序反应。
· DNA捕获珠(DNA Capture Bead):是一种直径约28微米的磁性珠子,它的表面覆盖了数千个固定在不同位置的寡核苷酸探针,这些探针与文库DNA片段的接头互补,可以通过乳液PCR(Emulsion PCR)扩增形成单分子聚集体(Single Molecule Cluster)。
· 荧光探针(Fluorescent probe):是指用于标记不同碱基对的四种荧光分子,它们分别对应A/T、T/A、C/G、G/C四种碱基对,并发出不同颜色的光。荧光探针还带有可逆终止子,可以控制每次只加入一个碱基对。
· 激光器(Laser):是指用于激发荧光探针发光的光源,它可以提供单波长、固态、长寿命的激光光束。
· CCD相机(CCD camera):是指用于捕捉和记录荧光信号的设备,它可以将荧光信号转换为数字化的电信号,并传输给计算机进行数据分析。
5.3. 主机组成
· 电泳系统:是指用于将带有荧光探针和可逆终止子的四种dNTPs逐一加入到FlowChip中,并利用DNA连接酶将它们连接到聚集体的DNA链上的系统。每次只能加入一个碱基对,然后用激光激发荧光信号,并用CCD相机记录每个聚集体发出的荧光信号,从而确定碱基对序列。
· 自动进样系统:是指用于将经过乳液PCR处理后的DNA捕获珠注入到FlowChip中,并使每个微孔中只有一个DNA捕获珠的系统。然后进行温度变化和化学处理,使聚集体单链化并去除P1端的DNA链,只留下P2端的DNA单链。
· 激光系统:是指用于激发荧光探针发光的光源,它可以提供单波长、固态、长寿命的激光光束。
· 光学系统:是指用于捕捉和记录荧光信号的设备,它可以将荧光信号转换为数字化的电信号,并传输给计算机进行数据分析。
· 温控系统:是指用于控制FlowChip板和反应液的温度,以保证测序反应的稳定性和效率的系统。
· 聚合物输送系统:是指用于将不同类型和浓度的聚合物溶液输送到FlowChip板中,以提供不同阶段所需的反应条件和试剂的系统。
6. Sequel II System

图源自PACB官网
Sequel II System测序仪是一种基于单分子实时测序(Single Molecule Real-Time Sequencing,SMRT)技术的三代测序平台,它可以提供高通量、高准确度和超长读长的DNA测序服务。
6.1. 相关原理
· 文库构建:将待测DNA打断成小片段,并在两端加上特殊的接头(Adaptor),这些接头包含用于测序引物结合的序列(P1/P2)。文库构建后需要进行质量检测和定量。
· SMRT Cell装载:将文库DNA片段与DNA聚合酶混合,并注入到SMRT Cell中,并使每个微孔中只有一个DNA聚合酶。然后进行温度变化和化学处理,使文库DNA片段与测序引物结合,并形成环状结构。
· 边合成边测序:将带有荧光核苷酸的四种dNTPs逐一加入到SMRT Cell中,并利用DNA聚合酶将它们连接到环状DNA链上。每次只能加入一个碱基,然后用激光激发荧光信号,并用CCD相机记录每个微孔发出的荧光信号,从而确定碱基序列。然后用化学剂去除环状DNA链上的碱基,使下一个碱基可以继续加入。重复这个过程,直到完成所有的测序循环。
· 数据分析:将CCD相机收集到的荧光信号转换为原始数据(BAM文件),并进行质量控制和过滤,去除低质量的信号。然后进行碱基识别(Base calling),将荧光信号转换为碱基序列(FASTQ文件)。最后根据不同的测序目的,进行后续的数据分析,如比对、变异检测、表达量计算等。
6.2. 主要组成
· 测序仪主机:包含电泳系统、自动进样系统、激光系统、光学系统、温控系统、聚合物输送系统等多个模块,用于控制仪器的运行和数据的采集。
· 计算机工作站:预装用于仪器控制、数据收集和样品文件自动分析的软件,如Data Collection Software、Sequencing Analysis Software等。
· SMRT Cell 8M:是一个微型的玻璃芯片,它的表面覆盖了数百万个微孔,每个微孔可以容纳一个DNA聚合酶(DNA Polymerase),并进行单分子测序反应。
· 荧光核苷酸(Fluorescent nucleotide):是指用于标记不同碱基的四种荧光分子,它们分别对应A、T、C、G四种碱基,并发出不同颜色的光。荧光核苷酸在被DNA聚合酶催化加入到DNA链上时,会释放出荧光信号,并被去除。
· 激光器(Laser):是指用于激发荧光核苷酸发光的光源,它可以提供单波长、固态、长寿命的激光光束。
· CCD相机(CCD camera):是指用于捕捉和记录荧光信号的设备,它可以将荧光信号转换为数字化的电信号,并传输给计算机进行数据分析。
6.3. 主机组成
· 电泳系统:是指用于将带有荧光核苷酸的四种dNTPs逐一加入到SMRT Cell中,并利用DNA聚合酶将它们连接到环状DNA链上的系统。每次只能加入一个碱基,然后用激光激发荧光信号,并用CCD相机记录每个微孔发出的荧光信号,从而确定碱基序列。
· 自动进样系统:是指用于将文库DNA片段与DNA聚合酶混合,并注入到SMRT Cell中,并使每个微孔中只有一个DNA聚合酶的系统。然后进行温度变化和化学处理,使文库DNA片段与测序引物结合,并形成环状结构。
· 激光系统:是指用于激发荧光核苷酸发光的光源,它可以提供单波长、固态、长寿命的激光光束。
· 光学系统:是指用于捕捉和记录荧光信号的设备,它可以将荧光信号转换为数字化的电信号,并传输给计算机进行数据分析。
· 温控系统:是指用于控制SMRT Cell板和反应液的温度,以保证测序反应的稳定性和效率的系统。
· 聚合物输送系统:是指用于将不同类型和浓度的聚合物溶液输送到SMRT Cell板中,以提供不同阶段所需的反应条件和试剂的系统。
7. MinION Device

图源自Oxford官网
MinION Device测序仪是一种基于单分子实时测序(Single Molecule Real-Time Sequencing,SMRT)技术的三代测序平台,它可以提供高通量、高准确度和超长读长的DNA和RNA测序服务。
7.1. 相关原理
· 文库构建:将待测DNA或RNA打断成小片段,并在两端加上特殊的接头(Adaptor),这些接头包含用于测序引物结合的序列(P1/P2)。文库构建后需要进行质量检测和定量。
· SMRT Cell装载:将文库片段与DNA聚合酶混合,并注入到SMRT Cell中,并使每个微孔中只有一个DNA聚合酶。然后进行温度变化和化学处理,使文库片段与测序引物结合,并形成环状结构。
· 边合成边测序:将带有荧光核苷酸的四种dNTPs逐一加入到SMRT Cell中,并利用DNA聚合酶将它们连接到环状DNA链上。每次只能加入一个碱基,然后用激光激发荧光信号,并用CCD相机记录每个微孔发出的荧光信号,从而确定碱基序列。然后用化学剂去除环状DNA链上的碱基,使下一个碱基可以继续加入。重复这个过程,直到完成所有的测序循环。
· 数据分析:将CCD相机收集到的荧光信号转换为原始数据(FAST5文件),并进行质量控制和过滤,去除低质量的信号。然后进行碱基识别(Base calling),将荧光信号转换为碱基序列(FASTQ文件)。最后根据不同的测序目的,进行后续的数据分析,如比对、变异检测、表达量计算等。
7.2. 主要组成
· 测序仪主机:是一个小巧的USB设备,它可以连接到任何电脑或笔记本,并通过软件进行控制和数据传输。
· SMRT Cell:是一个微型的塑料芯片,它的表面覆盖了数千个微孔,每个微孔可以容纳一个DNA聚合酶(DNA Polymerase),并进行单分子测序反应。
· 荧光核苷酸(Fluorescent nucleotide):是指用于标记不同碱基的四种荧光分子,它们分别对应A、T、C、G四种碱基,并发出不同颜色的光。荧光核苷酸在被DNA聚合酶催化加入到DNA链上时,会释放出荧光信号,并被去除。
· 激光器(Laser):是指用于激发荧光核苷酸发光的光源,它可以提供单波长、固态、长寿命的激光光束。
· CCD相机(CCD camera):是指用于捕捉和记录荧光信号的设备,它可以将荧光信号转换为数字化的电信号,并传输给电脑进行数据分析。
7.3. 主机组成
· 电泳系统:是指用于将带有荧光核苷酸的四种dNTPs逐一加入到SMRT Cell中,并利用DNA聚合酶将它们连接到环状DNA链上的系统。每次只能加入一个碱基,然后用激光激发荧光信号,并用CCD相机记录每个微孔发出的荧光信号,从而确定碱基序列。
· 自动进样系统:是指用于将文库片段与DNA聚合酶混合,并注入到SMRT Cell中,并使每个微孔中只有一个DNA聚合酶的系统。然后进行温度变化和化学处理,使文库片段与测序引物结合,并形成环状结构。
· 激光系统:是指用于激发荧光核苷酸发光的光源,它可以提供单波长、固态、长寿命的激光光束。
· 光学系统:是指用于捕捉和记录荧光信号的设备,它可以将荧光信号转换为数字化的电信号,并传输给电脑进行数据分析。
8. HeliScope Single Molecule Sequencer
HeliScope Single Molecule Sequencer测序仪是一种基于荧光测序原理的单分子测序平台,它可以直接对DNA进行测序,无需进行PCR扩增或文库构建。
8.1. 相关原理
· 文库准备:将待测DNA打断成小片段,并在每个小片段(约200bp)的末端加上poly-A尾。
· 芯片装载:将文库DNA片段与固定在芯片上的poly-T引物进行杂交,并精确定位,使每个微孔中只有一个DNA模板。
· 边合成边测序:将带有荧光探针和可逆终止子的四种dNTPs逐一加入到芯片中,并利用DNA聚合酶将它们连接到DNA链上。每次只能加入一个碱基对,然后用激光激发荧光信号,并用CCD相机记录每个微孔发出的荧光信号,从而确定碱基对序列。然后用化学剂去除荧光探针和可逆终止子,使下一个碱基对可以继续加入。重复这个过程,直到完成所有的测序循环。
· 数据分析:将CCD相机收集到的荧光信号转换为原始数据(BCL文件),并进行质量控制和过滤,去除低质量的信号。然后进行碱基识别(Base calling),将荧光信号转换为碱基对序列(FASTQ文件)。最后根据不同的测序目的,进行后续的数据分析,如比对、变异检测、表达量计算等。
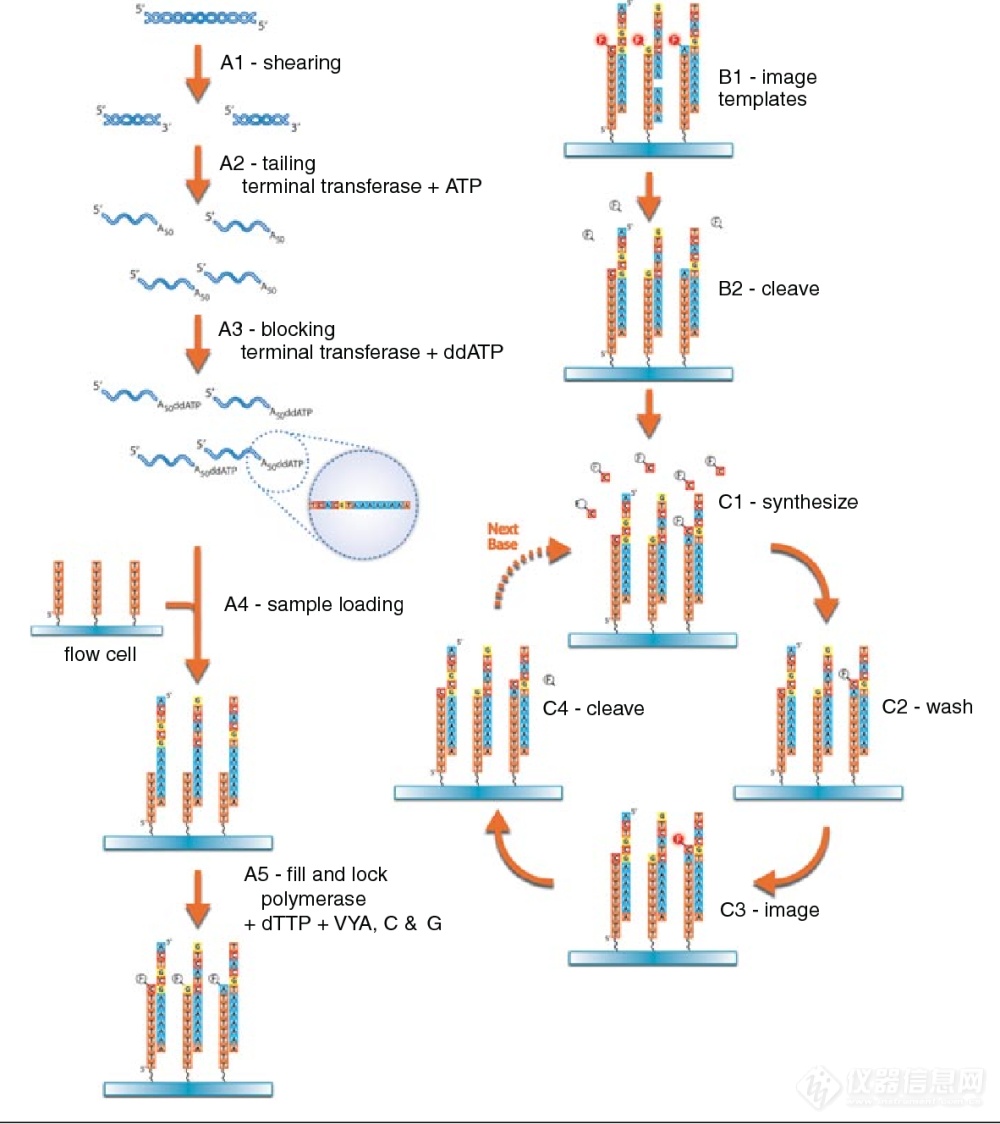
图源自portorford.info|
8.2. 主要组成
· 测序仪主机:是一个大型的设备,它可以连接到电脑或服务器,并通过软件进行控制和数据传输。
· 测序芯片:是一个微型的玻璃芯片,它的表面覆盖了数亿个微孔,每个微孔可以容纳一个DNA模板,并进行单分子测序反应。
· 荧光探针:是指用于标记不同碱基的四种荧光分子,它们分别对应A、T、C、G四种碱基,并发出不同颜色的光。荧光探针在被DNA聚合酶催化加入到DNA链上时,会释放出荧光信号,并被去除。
· 激光器:是指用于激发荧光探针发光的光源,它可以提供单波长、固态、长寿命的激光光束。
· CCD相机:是指用于捕捉和记录荧光信号的设备,它可以将荧光信号转换为数字化的电信号,并传输给电脑进行数据分析。
二. 测序原理和发展
· 测序技术的概念和原理:介绍什么是测序技术,它是如何工作的,以及它的主要分类和特点 。
· 测序技术的发展历史:回顾测序技术的发展过程,从第一代测序技术(如Sanger法)到第二代测序技术(如Illumina法)再到第三代测序技术(如Nanopore法) 。
· 测序技术的应用领域:介绍测序技术在生命科学中的各种应用,如基因组学、转录组学、表观遗传学、微生物组学、个体化医疗等 。
· 测序技术的挑战和未来:分析测序技术面临的主要挑战,如数据量、数据质量、数据分析、数据存储、数据共享等 ,以及展望测序技术的未来发展方向和趋势 。
1. 基本概念
· 读长(read length):读长是指测序得到的DNA片段的长度,一般来说,读长越长,越有利于拼接基因组和发现结构变异。在这方面,三代测序技术(如Nanopore和PacBio)具有明显的优势,它们可以测定长达数十kb甚至Mb级别的读长,而二代测序技术(如Illumina和Roche 454)的读长一般在数百bp到数千bp之间,Helicos的读长则最短,只有25-50bp。
· 准确度(accuracy):准确度是指测序结果与真实DNA序列的一致性,一般用错误率来衡量。
· 通量(throughput):通量是指测序平台每次运行可以产生的数据量,一般用Gbp或Tbp来表示。
· 成本(cost):成本是指进行测序所需的费用,包括仪器、试剂、人工、时间等因素。
2. 技术的选择
“如何选择合适的测序技术”这是一个很重要的问题,因为不同的测序技术有不同的特点和适用范围。一般来说,选择测序技术需要考虑以下几个因素:
· 测序目的:你想要测定什么样的DNA或RNA?是基因组、转录组、表观遗传组、微生物组还是其他?你想要了解什么样的信息?是序列变异、基因表达、基因调控、基因功能还是其他?
· 测序需求:你需要多少数据量来达到你的测序目的?你需要多高的准确度和重复性来保证你的测序质量?你需要多长的读长来覆盖你的目标区域?
· 测序资源:你有多少样品可以进行测序?你的样品质量和数量如何?你有多少时间和预算可以用于测序?
根据这些因素,你可以对比不同的测序技术的优缺点,选择最适合你的测序方案。
· 如果你想要测定全基因组或全转录组,并且对数据量和成本有较高的要求,那么你可以选择Illumina或Roche 454等二代测序技术,它们可以提供高通量和低成本的测序服务。
· 如果你想要测定特定的基因或区域,并且对准确度和重复性有较高的要求,那么你可以选择Sanger法等一代测序技术,它们可以提供高准确度和高重复性的测序服务。
· 如果你想要测定长片段或结构变异,并且对读长和拼接有较高的要求,那么你可以选择Nanopore或PacBio等三代测序技术,它们可以提供超长读长和单分子测序的服务。
3. 概念和原理
测序技术是指获得目的核酸分子(DNA或RNA)碱基排列顺序的技术,它是生命科学研究的基础和核心。测序技术的原理是利用不同的方法对目的核酸分子进行合成、标记、检测和识别,从而确定其碱基序列。
测序技术可以根据其使用的方法和原理分为不同的代数和类型,主要有以下几种:
· 第一代测序技术:是指最早出现的基于荧光测序原理的测序技术,如Sanger法和Maxam-Gilbert法,它们通过使用特殊的链终止核苷酸或化学降解剂来中断DNA合成反应,并通过凝胶电泳和放射自显影来检测荧光信号,从而确定碱基序列。Sanger法是基于DNA聚合酶在体外DNA复制过程中随机掺入终止链延伸的双脱氧核苷酸(ddNTPs)的原理。这些ddNTPs可以用放射性或荧光标记来检测,从而得到DNA的碱基序列。Sanger法由英国生物化学家弗雷德里克·桑格于1977年发明,是第一代DNA测序技术,曾被广泛用于人类基因组计划等大规模的基因组分析。
· 第二代测序技术:是指基于高通量测序原理的测序技术,如Illumina法、Roche 454法、ABI SOLiD法等,它们通过使用特殊的接头、引物、荧光探针等来对目的核酸分子进行扩增、标记和检测,并通过芯片或微珠等平台来实现大规模并行测序,从而大大提高了测序速度和通量。
o Roche 454技术:这是一种基于焦磷酸测序法的技术,它利用喷雾法将DNA打断成小片段,并在两端加上接头。然后将这些片段结合在微珠上,并在乳液PCR中进行扩增。最后将这些微珠放入一个含有许多小孔的反应板中,每个小孔只容纳一个微珠。在测序过程中,每次加入一种dNTP,并检测每个小孔中是否发生了焦磷酸释放反应,从而确定碱基序列。
o Illumina/Solexa技术:这是一种基于桥式PCR和荧光检测的技术,它也是目前最流行的测序技术之一。它将DNA打断成小片段,并在两端加上接头。然后将这些片段吸附在流动槽(flowcell)的表面,并进行桥式PCR扩增,形成聚集体(cluster)。在测序过程中,每次加入四种带有不同荧光标记的dNTP,并利用激光和相机记录每个聚集体发出的荧光信号,从而确定碱基序列。
o ABI SOLiD技术:这是一种基于连接酶和荧光检测的技术,它与Illumina技术类似,也是将DNA打断成小片段,并在两端加上接头。然后将这些片段结合在微珠上,并在乳液PCR中进行扩增。最后将这些微珠固定在玻璃滑片上,形成聚集体。在测序过程中,每次加入四种带有不同荧光标记的二聚体(如AA,AC,AG,AT等),并利用连接酶将它们连接到模板链上。然后利用激光和相机记录每个聚集体发出的荧光信号,从而确定碱基序列。
· 第三代测序技术:是指基于单分子实时测序原理的测序技术,可以直接测定单分子的DNA或RNA的测序方法,不需要进行PCR扩增,从而减少错误和偏差,并且可以获得更长的读长(read length)。如Nanopore法、PacBio法、Helicos法等,它们通过使用特殊的微孔、DNA聚合酶、荧光探针等来对单个核酸分子进行直接测序,无需进行扩增或文库构建,并通过电信号或光信号来检测碱基加入的过程,从而获得超长读长和高准确度的碱基序列。
o PacBio公司的单分子实时测序技术(SMRT),它是基于DNA聚合酶在体外DNA复制过程中随机掺入带有荧光标记的dNTPs的原理。这些dNTPs可以用激光和相机检测,从而得到DNA的碱基序列。PacBio的优点是可以测定长达数十kb的读长,以及检测一些碱基修饰情况,如甲基化等。PacBio的缺点是测序错误率较高(约10-15%),主要为随机的插入和缺失错误,但可以通过多次测序和自身校正来提高准确度。
o Oxford Nanopore公司的单分子纳米孔测序技术(Nanopore),它是基于电信号检测原理,当DNA分子穿过纳米孔时会产生电流信号,一般以5个碱基为一组检测电流信号,对电流信号进行解码。Nanopore的优点是可以测定超长的读长,最长可达Mb级别,以及便携性和实时性。Nanopore的缺点是测序错误率也较高(约10-15%),主要为同聚物和串联重复区域的错误,以及反向重复序列对测序质量的影响。
o Helicos公司的真正单分子测序技术(tSMS),它是基于荧光检测原理,将DNA打断成小片段,在每个小片段的末端加上poly-dA,并于玻璃芯片上随机固定多个poly-dT引物。然后逐一加入带有荧光标记和终止子的dNTPs,并利用显微镜记录每个小片段发出的荧光信号,从而确定碱基序列。Helicos的优点是可以避免PCR扩增带来的偏差,以及对样品量和纯度要求低。Helicos的缺点是测序错误率最高(约20-30%),主要为缺失错误,以及同聚物对测序质量的影响。
4. 发展历史
测序技术的发展历史可以追溯到1975年,当时Frederick Sanger提出了链终止法,并用它成功地测定了噬菌体φX174的基因组序列(5375个碱基),这是人类历史上第一个完整的基因组图谱。
1977年,Walter Gilbert提出了链降解法,并用它成功地测定了噬菌体MS2的基因组序列(3569个碱基)。
1980年,Sanger和Gilbert因为在测序技术方面的贡献而共同获得了诺贝尔化学奖。
1986年,Leroy Hood等人发明了第一台自动化荧光测序仪,并用它成功地完成了人类线粒体DNA(16569个碱基)的全长测序。
1990年,人类基因组计划正式启动,目标是在15年内完成人类全基因组(约30亿个碱基)的测定。
1995年,Craig Venter等人利用全基因组随机打断法(Whole Genome Shotgun Method)首次完成了一种自由生活细菌——溶血性链球菌的全基因组测序(180万个碱基)。
1996年,Roche公司收购了454 Life Sciences公司,并开始开发基于焦磷酸测序法(Pyrosequencing)的高通量测序技术。
1998年,ABI公司推出了第一台基于荧光原位合成法(Fluorescence In Situ Sequencing,FISSEQ)的高通量测序仪——ABI 3700 Genetic Analyzer。
2001年,人类基因组计划和Celera Genomics公司分别公布了人类基因组的初步草图,标志着人类基因组计划的完成。
2005年,Solexa公司推出了第一台基于桥式扩增法(Bridge Amplification)和可逆终止法(Reversible Terminator)的高通量测序仪——Solexa 1G Genome Analyzer。
2006年,Illumina公司收购了Solexa公司,并开始开发基于桥式扩增法和可逆终止法的高通量测序技术。
2007年,Roche 454公司推出了第一台基于乳胶珠扩增法(Emulsion PCR)和焦磷酸测序法的高通量测序仪——Roche 454 GS FLX。
2008年,ABI公司推出了第一台基于乳胶珠扩增法和荧光连接法(Ligation Sequencing)的高通量测序仪——ABI SOLiD System。
2009年,Helicos Biosciences公司推出了第一台基于单分子荧光测序法(Single Molecule Fluorescent Sequencing)的单分子测序仪——HeliScope Single Molecule Sequencer。
2010年,Pacific Biosciences公司推出了第一台基于单分子实时测序法(Single Molecule Real-Time Sequencing,SMRT)的单分子测序仪——PacBio RS。
2011年,Ion Torrent公司推出了第一台基于半导体测序法(Semiconductor Sequencing)的高通量测序仪——Ion Torrent PGM。
2012年,Oxford Nanopore Technologies公司推出了第一台基于纳米孔测序法(Nanopore Sequencing)的单分子测序仪——MinION Device。
2015年,BGI公司推出了第一台基于芯片化荧光测序法(Chip-based Fluorescent Sequencing)的高通量测序仪——BGISeq-500。
2017年,10x Genomics公司推出了第一台基于连线染色体构象捕获技术(Linked-Reads Technology)的高通量测序仪——Chromium Genome System。
至此,从第一代到第三代的各种测序技术已经形成了一个多样化、竞争性和互补性的生态系统,为生命科学研究提供了丰富而强大的工具。
2018年到2023年最近5年测序技术的发展:
· 测序技术的创新和优化:在这段时间内,各种测序技术都在不断地进行创新和优化,以提高测序的速度、准确度、通量、成本效益等方面的性能。例如,Illumina公司推出了NovaSeq系列测序仪,可以实现每天测序6000个人类基因组;PacBio公司推出了Sequel II和Sequel IIe测序仪,可以实现每次测序8Tb的数据和平均读长20kb;Oxford Nanopore公司推出了PromethION和GridION测序仪,可以实现每次测序100Tb的数据和平均读长30kb;Ion Torrent公司推出了Genexus集成化测序系统,可以实现24小时内完成从样本到报告的全流程;10x Genomics公司推出了Chromium X系列测序仪,可以实现每次测序1.2Tb的数据和平均读长150kb;BGI公司推出了DNBSEQ-T7和DNBSEQ-G400测序仪,可以实现每次测序6Tb和400Gb的数据和平均读长100bp。
· 测序技术的多样化和互补性:在这段时间内,各种测序技术都在不断地扩展其应用范围和领域,以满足不同的研究需求和目标。例如,Illumina公司推出了TruSeq Nano DNA Library Prep Kit,可以实现从低至100ng的DNA样本进行全基因组测序;PacBio公司推出了HiFi Reads技术,可以实现单分子测序的高准确度(>99%);Oxford Nanopore公司推出了LamPORE技术,可以实现从RNA直接进行SARS-CoV-2病毒检测;Ion Torrent公司推出了Oncomine Precision Assay,可以实现从肿瘤组织或血液样本进行癌症基因检测;10x Genomics公司推出了Visium Spatial Gene Expression Solution,可以实现从组织切片进行空间转录组测序;华大智造推出DNBelab C系列高通量文库制备试剂盒,自动化文库制备系统,节省人力物力,提高通量,减少操作失误。
· 测序技术的应用和赋能:在这段时间内,各种测序技术都在不断地应用于各个领域和行业,以促进科学发现和社会进步。例如,在基因组学领域,完成了人类基因组计划第二阶段(HGP-write)的启动、人类细胞图谱计划(Human Cell Atlas)的进展、人类变异图谱计划(Human Variome Project)的更新等重大项目;在转录组学领域,完成了人类脑转录组计划(BRAIN Initiative)的初步结果、人类免疫细胞转录组计划(Human Immunome Project)的部分结果、人类肠道菌群转录组计划(Human Gut Microbiome Project)的部分结果等重要研究;在表观遗传学领域,完成了人类表观组计划(Human Epigenome Project)的部分结果、人类表观组图谱计划(Human Epigenome Atlas)的部分结果、人类表观组变异计划(Human Epigenome Variation Project)的部分结果等关键研究;在微生物组学领域,完成了地球微生物组计划(Earth Microbiome Project)的部分结果、人类口腔微生物组计划(Human Oral Microbiome Project)的部分结果、人类皮肤微生物组计划(Human Skin Microbiome Project)的部分结果等重要研究;在个体化医疗领域,完成了百万人基因组计划(Million Genomes Project)的部分结果、百万人精准医疗计划(Million Precision Medicine Project)的部分结果、百万人癌症基因组计划(Million Cancer Genomes Project)的部分结果等重大项目。
5. 应用领域
o 基因组学:是指研究生物体所有遗传信息及其功能、结构、表达、变异、进化等方面的学科,它依赖于测序技术来获取基因组序列和注释,以及进行基因组比较、基因组变异、基因组编辑等研究。
o 转录组学:是指研究生物体在特定条件下所有转录本的类型、数量、结构、功能和相互作用的学科,它依赖于测序技术来获取转录本序列和表达量,以及进行转录本组装、差异表达分析、可变剪接分析、非编码RNA分析等研究。
o 表观遗传学:是指研究生物体在不改变DNA序列的情况下,通过化学修饰或染色质重塑等方式调控基因表达的学科,它依赖于测序技术来获取DNA甲基化、组蛋白修饰、染色质可及性等信息,以及进行表观遗传标记分布、表观遗传变异、表观遗传调控等研究。
o 微生物组学:是指研究特定环境或宿主中所有微生物的种类、数量、功能和相互作用的学科,它依赖于测序技术来获取微生物的16S rRNA或全基因组序列,以及进行微生物分类鉴定、微生物群落结构、微生物功能分析等研究。
o 个体化医疗:是指根据个人的基因组、转录组、蛋白质组等信息,为其提供最适合的预防、诊断和治疗方案的医疗模式,它依赖于测序技术来获取个人的遗传变异和表达谱,以及进行个人风险评估、个人药物反应预测、个人靶向治疗选择等应用。
6. 挑战和未来
测序技术虽然已经取得了巨大的进步和成就,但仍然面临着一些挑战和问题,主要包括:
· 数据量:随着测序技术的发展,测序数据的产生速度远远超过了数据存储和处理的能力,导致数据管理和分析成为一个瓶颈。
· 数据质量:不同的测序技术有着不同的数据质量特征,如读长、准确度、偏好性等,这些特征会影响数据分析的结果和可靠性。
· 数据分析:测序数据的分析涉及到多种复杂的算法和工具,如比对、组装、注释、变异检测等,这些算法和工具需要不断地优化和更新,以适应不同的数据类型和需求。
· 数据存储:测序数据的存储需要占用大量的硬件资源和空间,同时也需要考虑数据的安全性和可访问性。
· 数据共享:测序数据的共享需要解决数据的标准化、元数据、伦理、法律等方面的问题,同时也需要建立有效的数据交换和利用的机制和平台。
测序技术的未来发展方向和趋势主要包括:
· 数据集成:通过将不同来源、不同层次、不同类型的测序数据进行整合和融合,以提高数据的信息量和价值。
· 数据挖掘:通过运用机器学习、人工智能等先进的技术和方法,对测序数据进行深入的分析和挖掘,以发现数据中隐藏的规律和知识。
· 数据应用:通过将测序数据与其他领域的数据进行关联和对比,以拓展测序数据的应用范围和意义。
· 数据创新:通过开发新的测序技术和平台,以提高测序数据的质量和效率,以及实现新的测序功能和目标。
来源于:仪器信息网
热门评论
最新资讯
新闻专题

















