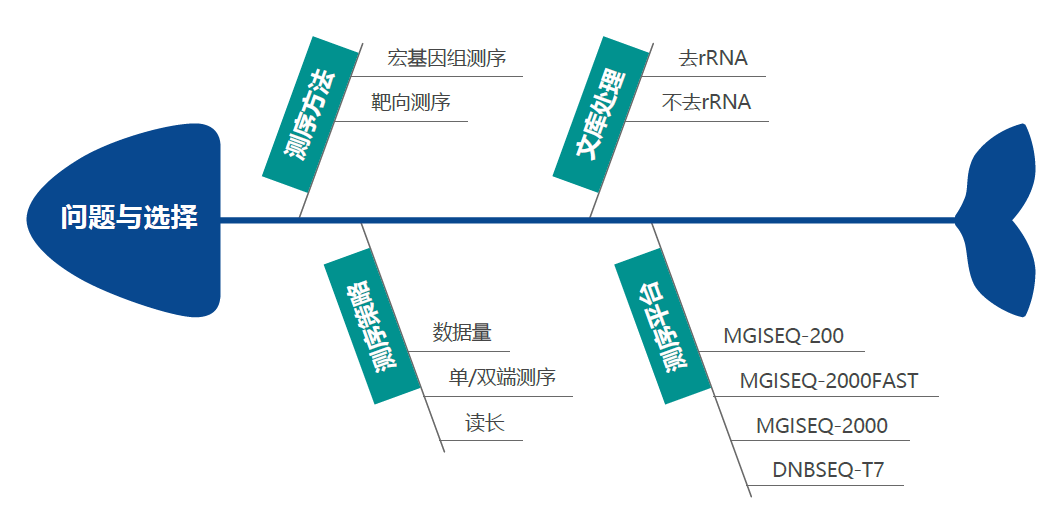
【安捷伦】鉴定新型冠状病毒,宏基因组二代测序(mNGS)技术十分关键!
自 2019 新型冠状病毒(2019-nCoV)肺炎疫情爆发以来,相关科研单位便紧锣密鼓地开展病毒研究工作,并取得了一系列重要的研究成果。2 月 3 日,Nature 在线发布了复旦大学张永振教授团队的一项重要研究成果,该团队对患者支气管肺泡灌洗液进行了宏基因组二代测序(mNGS),鉴定出了一种新型冠状病毒,并发现该病毒基因组与蝙蝠体内发现的 SARS 样冠状病毒基因组有 89.1% 的相似性[1]。张永振教授团队发表的文章截图 | 图源:Nature2 月 20 日,bioRxiv 预印本平台发布了华南农业大学沈永义教授、肖立华教授团队关于新冠病毒中间宿主的研究成果。通过对穿山甲样品进行宏基因组分析,该团队发现穿山甲为新型冠状病毒潜在中间宿主[2]。沈永义教授、肖立华教授团队发表的文章截图 | 图源:bioRxiv可以说,宏基因组测序在新病原体的诊断、监测、跟踪,以及溯源方面具有关键作用,更是新冠病毒研究的一大助力。宏基因组测序:病原体检测的新风口1998 年,威斯康辛大学的 Jo Handelsman 提出宏基因组学(Metagenomics)的概念,并将其定义为:一种以环境样品中的微生物群体基因组为研究对象,以功能基因筛选和测序分析为研究手段,以微生物多样性、种群结构、进化关系、功能活性、相互协作关系、以及与环境之间的关系为研究目的的新的微生物研究方法[3]。包括宏基因组学在内的各类组学研究(右)相较传统遗传学与生物化学方法(左)在全基因水平的研究上效率更高 | 图源:Science2014 年,新英格兰医学杂志发表宏基因组二代测序(mNGS)确诊钩体病的首例临床应用案例[4],打响了病原体 mNGS 的第一枪。新英格兰医学杂志发表宏基因组二代测序(mNGS)确诊钩体病的文章截图 | 图源:NEJM短短 5 年来,mNGS 在新发病原体鉴定、罕见重要病原体诊断和临床大数据研究等方面取得诸多进展。例如在 2018 年,Clinical and Research in Hepatology and Gastroenterology 发布了上海华山医院感染科张文宏教授团队使用 mNGS 协助临床诊断肝结核的案例,mNGS 的适时使用,准确快速地帮助临床明确了患者的发热病因,推动了临床的精准诊断[5]。张文宏教授团队发表文章截图 | 图源:ScienceDirect2019 年,中国临床专家也达成共识,认可了宏基因组分析和诊断技术在急危重症感染领域的临床应用[6]。临床专家共识文章截图 | 图源:万方宏基因组二代测序的流程及原理宏基因组二代测序的检测流程可以大致分为5个步骤:核酸提取、文库构建、上机测序、生物信息学分析与报告解读[7]。具体来看,对于不同的临床样本,核酸提取前需要进行不同的前处理,比如痰液液化、破壁、去宿主等以提高病原体检出率。RNA 病毒需要在文库构建前进行逆转录,生成 cDNA。文库构建的目的在于给未知序列的核酸片段两端加上已知序列信息的接头以便于测序,单样本文库构建完成后需要经历 PCR 扩增、再将多个文库样本混合后进行测序。测序完成后,数据会自动进入搭建好的病原体自动分析流程,该流程包括去除人源宿主序列和低质量序列、以及微生物数据库比对注释等步骤。最后,解读专家根据自动化系统产生的初步结果,再结合部分临床指标、样本类型、病原体种类等因素进行综合分析解读。宏基因组工作流程示意图 | 图源:Nature Biotechnology样本文库质量是宏基因组测序的关键由于环境中的微生物种类五花八门,相对复杂,构建一个高质量的宏基因组文库在整个检测流程中便显得十分重要。故而在取样时,我们要严格遵循取样规则,在取样中应尽量避免对样本的干扰,缩短保存和运输的时间,使样品尽可能代表自然状态下的微生物原貌。并且要采用合适的方法,既要尽可能地完全抽提出环境样品中的 DNA/RNA,又要保持较大的片段以获得完整的目的基因或基因簇。在构建 RNA 文库之前需要对 RNA 样本进行完整性评估[8],只有达标的样本才能进入下一阶段反转录及文库构建。宏基因组文库的质量直接关系到测序的数据量,在影响成本的同时也影响了测序时间,因此,为了提高测序准确性、减少测序过程中的风险,测序前需要测定文库样品的浓度和片段大小分布,确定合适的上机 pooling 方案和测序深度。可见样本的质量控制对于宏基因组测序的重要性。安捷伦自动化样本质控解决方案安捷伦在核酸蛋白质量控制领域拥有愈二十年的经验,针对不同来源样本,分析靶标和通量需求提供全套的自动化解决方案。安捷伦的 2100 生物分析仪是目前最为普遍使用的二代测序质控设备。安捷伦在 2100 生物分析仪上最早开发出了 RNA 完整性参数(RIN),它已成为全世界公认的 RNA 质控指标,它以 0-10 的数值直观反应 RNA 样本的完整性程度,为标准化的实验操作提供了样本质量评估的参考标准。在本次新冠病毒(RNA 病毒)的序列确定中,安捷伦 2100 生物分析仪发挥了重要作用。随着测序样本量的增加,特别是随着后续病毒变异监测以及病毒溯源工作的逐步展开,安捷伦中-高-超高通量自动化核酸质控平台(4200 tapestation 和 AATI FA)将发挥它们的优势。参考文献[1] Yong-Zhen Zhang , Edward C. Holmes,Lin Xu,et al. A new coronavirus associated with human respiratory disease in China[J].nature,2020.[2] Xiao K, Zhai J, Feng Y, et al. Isolation and Characterization of 2019-nCoV-like Coronavirus from Malayan Pangolins[J]. bioRxiv, 2020.[3] Handelsman J, Rondon M R, Brady S F, et al. Molecular biological access to the chemistry of unknown soil microbes: a new frontier for natural products[J]. Chemistry & biology, 1998, 5(10): R245-R249.[4] Wilson M R, Naccache S N, Samayoa E, et al. Actionable diagnosis of neuroleptospirosis by next-generation sequencing[J]. New England Journal of Medicine, 2014, 370(25): 2408-2417.[5] Jing-Wen A , Yang L , Qi C , et al. Diagnosis of local hepatic tuberculosis through next-generation sequencing: Smarter, faster and better[J]. Clinics and Research in Hepatology and Gastroenterology, 2018, 42(3):178-181.[6] 宏基因组分析和诊断技术在急危重症感染应用专家共识组. 宏基因组分析和诊断技术在急危重症感染应用的专家共识[J]. 中华急诊医学杂志, 2019, 28(2):151-155.[7] Quince C, Walker A W, Simpson J T, et al. Shotgun metagenomics, from sampling to analysis[J]. Nature biotechnology, 2017, 35(9): 833.[8] Fan W, Su Z, Bin Yu, et al. A new coronavirus associated with human respiratory disease in China[J]. Nature. 2020 Feb 3. [Epub ahead of print]推荐阅读:1. 抗击新型冠状病毒,安捷伦核酸/蛋白质质量控制产品从这些方面入手!https://www.instrument.com.cn/netshow/SH100320/news_521879.htm2. Agilent 2100 生物分析仪https://www.agilent.com/zh-cn/product/automated-electrophoresis/bioanalyzer-systems/bioanalyzer-instrument/2100-bioanalyzer-instrument-228250关注“安捷伦视界”公众号,获取更多资讯。