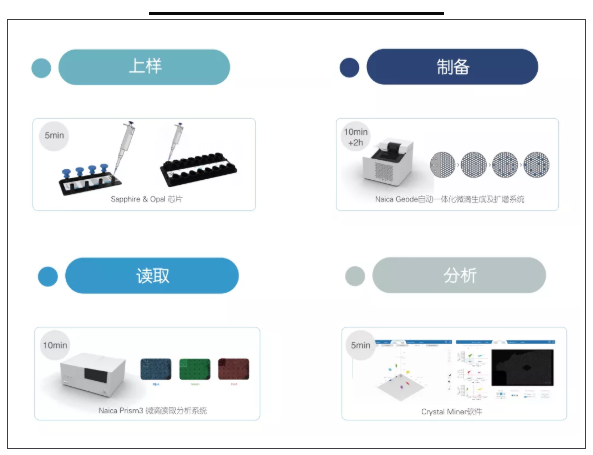
中科院能源所利用单细胞拉曼分选-测序耦合系统 首次精确到一个细菌细胞的环境菌群scRACS-Seq
摘要:2021年5月,中国科学院青岛生物能源与过程研究所荆晓艳博士等人应用星赛生物的RACS-Seq®单细胞拉曼分选-测序耦合系统,以及相应的RAGE芯片和单细胞分析试剂盒(包括环境样品中微生物单细胞提取与制备、稳定同位素饲喂细胞、单细胞核酸裂解与扩增等环节)在美国微生物学会会刊《mSystems》在线发表题为“One-Cell Metabolic Phenotyping and Sequencing of Soil Microbiome by Raman-Activated Gravity-Driven Encapsulation (RAGE)”的文章。单细胞拉曼分选耦合测序(RACS-Seq)是剖析环境菌群功能机制的重要手段,但拉曼分选后单个细菌细胞基因组的覆盖度通常低于10%,极大限制了其应用。近日,中国科学院青岛生物能源与过程研究所单细胞中心基于星赛生物的RACS-Seq®单细胞拉曼分选-测序耦合系统,以及相应的RAGE芯片和单细胞分析试剂盒(包括环境样品中微生物单细胞提取与制备、稳定同位素饲喂细胞、单细胞核酸裂解与扩增等环节),首次实现了精确到一个细菌细胞、全基因组覆盖度达93%的环境菌群scRACS-Seq,为环境微生物组原位代谢功能研究提供了一个强有力的新工具。土壤是地球上最重要的生态系统之一,土壤微生物组的代谢活动支撑着农业与畜牧业,也在地球元素循环、全球气候变化中起着关键性作用。同时,土壤菌群也是地球上最多样与最复杂的微生物组之一,而其中大部分微生物尚难以培养,因此,单个细胞精度的拉曼分析-分选-测序(Single-cell RACS-Seq,简称scRACS-Seq)策略,是剖析土壤等环境菌群之代谢机制的重要手段。然而针对环境菌群的scRACS-Seq一直以来存在两大瓶颈,一是难以无损、快速地获取具有特定拉曼表型的单个细胞;二是难以获得高覆盖度的单细胞基因组数据。这已经成为scRACS-Seq技术体系在复杂菌群中得以广泛应用的关键瓶颈。针对这一业界共性难点问题,单细胞中心荆晓艳、公衍海和徐腾等组成的联合攻关小组,基于前期发明的RAGE-Seq技术(Raman-activated Gravity-driven Encapsulation and Sequencing Xu, et al, Small, 2020,点击查看),从液相拉曼分析稳定同位素底物饲喂的土壤菌群出发,将特定拉曼表型的细菌单细胞精准分离并包裹到皮升级液滴中,进而耦合下游基因组测序。结果表明:(i)土壤菌群中细胞代谢活跃的低丰度物种(如Corynebacterium spp., Clostridium spp., Moraxella spp., Pantoea spp. 和 Pseudomonas spp.等)可经耦合重水饲喂与标记的RAGE-Seq精准地识别和分选,其单细胞基因组覆盖率可高达〜93%;(ii)同样,基于RAGE-Seq,含类胡萝卜素的土壤微生物细胞(如Pantoea spp., Legionella spp., Massilia spp., Pseudomonas spp., 和Pedobacter spp.等)能实现单个细胞分辨率、高基因组覆盖度的代谢重建,从而完整、深入地挖掘其类胡萝卜素合成途径;(iii)这些“原位”合成类胡萝卜素的土壤微生物细胞中,既有代谢活跃的,也相当部分是惰性的,表明基于纯培养的策略势必错失这些代谢惰性的功能微生物,因此“原位”、单细胞精度的功能细胞识别和分离,对于全面、客观的菌群功能剖析和资源挖掘具有重要意义。精确到一个细胞的拉曼分析-分选-测序(scRACS-Seq)此外,该工作还通过组分与状态均精确可控的人工菌群,建立了系统且严格的scRACS-Seq质量评价与控制体系。基于该体系,发现该技术能将不同拉曼表型的细菌单细胞从菌群中快速、精准分离,在保证单细胞拉曼光谱质量的同时,分选准确性达100%。此外,以来自于靶标细胞周围水相的空液滴为阴性对照,发现靶标细胞序列中被菌群中其他细胞DNA污染的概率极低。上述工作定量证明了scRACS-Seq的灵敏度、特异性和可靠性。借助星赛生物的RACS-Seq®单细胞拉曼分选-测序耦合系统,以及相应的RAGE芯片和单细胞分析试剂盒(包括环境样品中微生物单细胞提取与制备、稳定同位素饲喂细胞、单细胞核酸裂解与扩增等环节),scRACS-Seq可以在复杂菌群中以单个微生物细胞的分辨率建立新陈代谢与基因组的联系,从而精确回答“谁在做什么,为什么”。该系统广谱适用于细菌、古菌、真菌和动植物细胞,正服务于涵盖各种复杂生态系统的研究和应用。