naica®微滴芯片数字PCR系统量化造血干细胞移植儿童巨细胞病毒感染的病毒载量
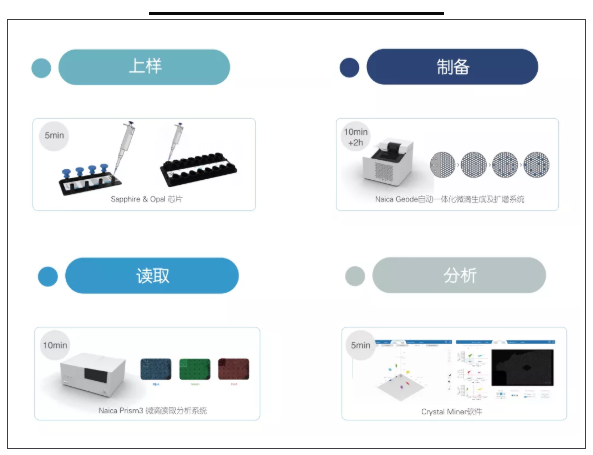
导读中国疾病预防控制中心国家病毒病预防控制研究所和中国首都儿科研究所的科学家在Canadian Journal of Infectious Diseases and Medical Microbiology上发表了题为The Viral Load of Human Cytomegalovirus Infection in Children following Hematopoietic Stem Cell Transplant by Chip Digital PCR的文章。文中应用naica® 微滴芯片数字PCR系统建立了芯片数字PCR(cdPCR)方法,能够精准定量HSCT前后儿童HCMV感染的病毒载量。质粒pUC57-UL83的cdPCR检测限为103拷贝/ml,qPCR检测限为297拷贝/ml。cdPCR检测HCMV AD169毒株的结果为146拷贝/ml,表明cdPCR的灵敏度高于qPCR。人类巨细胞病毒(HCMV)是一种普遍存在的β-疱疹病毒,已感染发展中国家高达90%的人口。作为一种常见病原体,HCMV感染在免疫抑制个体中引起了显著的发病率和死亡率,特别是在接受了造血干细胞移植(HSCT)的患者中,原因是原发感染后潜伏感染的主要靶细胞是造血细胞。对于HSCT后的高危儿童,应在出现临床症状之前检测HCMV感染,因为HCMV病毒的载量及变化与HSCT儿童HCMV感染的发展和严重程度高度相关。因此,HCMV病毒载量的定量检测对患儿的治疗至关重要。应用亮点:▶ 使用naica® 微滴芯片数字PCR系统开发了一种快速、直观、简便和准确的检测HSCT前后儿童HCMV病毒的绝对定量方法。▶ 通过质粒和培养毒株验证naica® 微滴芯片数字PCR系统灵敏度、特异性和重复性。实验方法:作者从首都儿科研究所儿童医院收集了122名异体造血干细胞移植患儿、3名自体造血干细胞移植患儿样本(男/女:73/52),中位年龄7.5岁。该研究通过质粒和培养毒株验证naica® 微滴芯片数字PCR系统灵敏度、特异性和重复性均优于qPCR。在HSCT前后,通过qPCR和cdPCR检测所有供体和受体血清中的HCMV病毒载量。实验结果:作者通过含有pUC57-UL83基因的质粒DNA分别评估cdPCR和qPCR的动态范围。cdPCR的检测限 (LOD) 为103拷贝/ml (2.0拷贝/反应),qPCR的LOD为297拷贝/ml。结果表明,cdPCR的灵敏度高于qPCR。为了评估cdPCR数据的重现性,作者使用质粒建立了HCMV DNA拷贝数的标准曲线。分析cdPCR检测的变异系数(CV、标准差/平均值)。结果表明,cdPCR检测具有良好的重复性(CVHCMV在125个HSCT患儿的检出率为30.40% (38/125),HCMV病毒载量范围为107拷贝/ml-6600拷贝/ml。男性组的检出率为30.14% (22/73),女性组的检出率为30.77% (16/52)。在0-12岁HSCT后HCMV阳性儿童中,HCMV的检出率为89.47% (34/38)。在0-6岁组中,男性的检出率为25.64% (10/39),女性的检出率为22.58% (7/31)。在7-12岁组中,男性的检出率为39.29% (11/28),女性的检出率为40% (6/15)。12岁以上患儿HCMV检出率为33.33% (4/12),男性检出率为16.67% (1/6),女性检出率为50% (3/6)。结果如表3所示。综上所述, cdPCR方法在HCMV检测领域比qPCR更敏感,能快速、直观、简便和准确的检测HSCT患儿HCMV感染率及病毒载量。参考文献:1.P. Griffiffiffiths, I. Baraniak, and M. Reeves, “,e pathogenesis of human cytomegalovirus,” Journal of Pathology, vol. 235, no. 2, pp. 288–297, 2015.2.L. Dupont and M. B. Reeves, “Cytomegalovirus latency and reactivation: recent insights into an age old problem,” Reviews in Medical Virology, vol. 26, no. 2, pp. 75–89, 2016.naica® 六通道微滴芯片数字PCR系统法国Stilla Technologies公司naica® 六通道微滴芯片数字PCR系统,源于Crystal微滴芯片式数字PCR技术,自动化微滴生成和扩增,每个样本孔可实现6荧光通道的检测,智能化识别微滴并进行质控,3小时内即可获得至少6个靶标基因的绝对拷贝数浓度。