专家点评NBT| 陈良怡/李浩宇合作团队发明计算超分辨图像重建算法,稳定提升荧光显微镜2倍分辨率
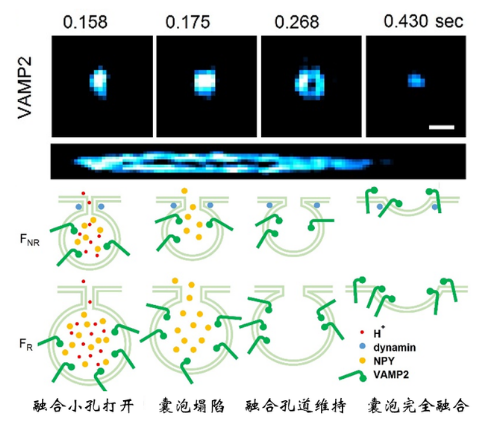
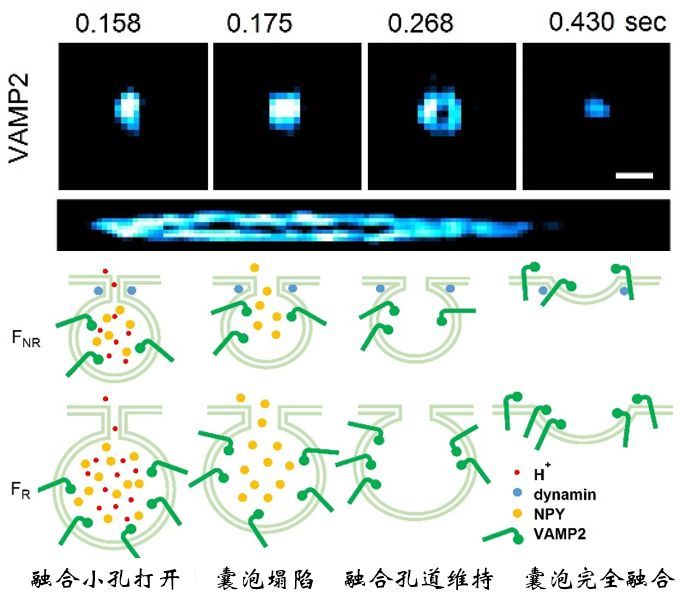
2014年诺贝尔化学奖授予了荧光超分辨显微技术,利用荧光分子的化学开关特性(PALM/FPALM/STORM)或者物理的直接受激辐射现象(STED),实现超越衍射极限的超分辨成像。尽管如此,活细胞中的超分辨率成像仍然存在两个主要瓶颈:(1)超分辨率的光毒性限制了观察活细胞中精细生理过程;(2)受限于荧光分子单位时间内发出的光子数,时间和空间分辨率不可兼得。受限于这个瓶颈,为了在活细胞上达到60 nm空间分辨率极限,现有超分辨率成像手段需要强照明功率(kW~MW/mm2)、特殊荧光探针和长曝光时间( 2 s)。强照明功率引起的强漂白会破坏真实荧光结构的完整性,长曝光时间在图像重构时导致运动伪影,降低有效分辨率。迄今为止,基于光学硬件或者荧光探针的改进无法进一步提升活细胞超分辨率的时空分辨率,实现毫秒尺度60 nm的时空分辨率成像。2021年11月16日,哈尔滨工业大学李浩宇教授团队与北京大学陈良怡教授团队合作在Nature Biotechnology上发表论文Sparse deconvolution improves the resolution of live-cell super-resolution fluorescence microscopy【1】。他们另辟蹊径,发明基于新计算原理的荧光超分辨率显微成像,进一步拓展荧光显微镜的分辨率极限。通过提出“荧光图像的分辨率提高等价于图像的相对稀疏性增加”这个通用先验知识,结合之前提出的信号空时连续性先验知识【2】,他们发明了两步迭代解卷积算法,即稀疏解卷积(Sparse deconvolution)方法,突破现有荧光显微系统的光学硬件限制,首次实现通用计算荧光超分辨率成像。结合自主研发的超分辨率结构光(SIM)系统,实现目前活细胞光学成像中最高空间分辨率(60nm)下,速度最快(564Hz)、成像时间最长(1小时以上)的超分辨成像。结合商业的转盘共聚焦结构光显微镜,实现四色、三维、长时间的活细胞超分辨成像。1、应用举例:DNA折纸标准样本验证为了在已知结构样本中验证分辨率的提升,研究者设计并合成了两个荧光标记位点的DNA折纸样本,每个位点用4~5个Cy5标记。当这些分子间距为60 nm、80 nm和100 nm时,它们在TIRF-SIM下几乎无法区分,但在经过稀疏解卷积重建后(Sparse-SIM,图1)可以很好地区分它们中间的距离。整体结果可以用单分子定位显微镜ROSE【3】交叉验证,与Sparse-SIM得到的DNA折纸的荧光对间距以及不同间距荧光对在玻片上的分布一致。图1:Sparse-SIM解析不同距离DNA折纸样本。(a)在相同视场下,用配对Cy5标记不同距离(60 nm, 80 nm, 100 nm, 120 nm)的DNA折纸样品,用TIRF(左)、TIRF-SIM(中)和Sparse-SIM(右)成像。(b)在TIRF、TIRF-SIM和Sparse-SIM下,黄色(60 nm)、蓝色(80 nm)(80 nm)、绿色(100 nm)和红色(120 nm)框包围的放大区域。比例尺:(a)2 μm;(b)100 nm。2、应用举例:Sparse-SIM超快活细胞成像揭示核孔结构和胰岛素囊泡早期融合孔道在活细胞成像中,稀疏结构光显微镜(Sparse-SIM)可以解析标记不同核孔蛋白(Nup35, Nup93, Nup98,或Nup107)的环状核孔结构,而它们在传统结构光显微镜(2D-SIM)下形状大小与100 nm荧光珠类似(图2c, 2d)。由于相机像素尺寸与孔径直径类似,测量的核孔拟合直径与Sparse-SIM的分辨率相当。校正后Nup35和Nup107孔的直径分别为~66 ± 3 nm和~97 ± 5 nm,而Nup98和Nup93直径大小处于这个范围中(图2e, 2f),结果与以前用其他超分辨成像方法在固定细胞中获得的直径相符【4】。有趣的是,12分钟超分辨成像可以显示活细胞中核孔形状变化,这可能反映了核膜上的单个核孔复合物动态重新定向到焦平面或远离焦平面(图2g),这是其他超分辨方法难以观察到的。图2:Sparse-SIM解析核孔蛋白动态过程。(c)用Sparse-SIM观察活COS-7细胞中以Nup98-GFP标记的动态环状核孔的典型例子,持续时间超过10分钟。上下区域分别显示2D-SIM和Sparse-SIM下的图像。(d)比较(c)中青色框中的核孔结构快照与100 nm荧光珠在不同重建方法(2D-SIM、20次RL解卷积后、50次RL解卷积后、Sparse-SIM)下的结果。(e)由于核孔的大小与Sparse-SIM的分辨率和像素大小相当,按照Supplementary Note 9.1的协议(详情请见文章),分别推导出Nup35-GFP(红色)、Nup98-GFP(黄色)、Nup93-GFP(绿色)和Nup107-GFP(青色)标记的核孔结构的实际直径。(f)Nup35(66 ± 3 nm, n=30)、Nup98(75 ± 6 nm, n=40)、Nup93(79 ± 4 nm, n = 40)、Nup107(97 ± 5nm ,n = 40)的平均直径环。左右两幅蒙太奇分别为传统Wiener重构或稀疏解卷积后的结果。(g)在6个时间点对 (c)中的品红色方框放大并显示。比例尺:(c)500 nm;(d, g, f)100 nm。通过滚动重建,Sparse-SIM的时间分辨率可达564 Hz,识别出来INS-1细胞中VAMP2-pHluorin标记的、更小的胰岛素囊泡融合孔道(如~61 nm孔径)。它们在囊泡融合的早期出现,孔径小(平均直径~87 nm),持续时间短(9.5 ms),不能被之前传统的TIRF-SIM所识别【2】。另一方面,鉴别出来的稳定融合孔在囊泡融合的后期出现,孔径大(平均直径~116 nm),持续时间长(47 ms),是之前看到的结构【2】。值得一提的是,虽然这里发现的囊泡早期融合孔状态很难被其他的超分辨率成像手段所直接验证,但是它们的发生频率与30多年前用快速冷冻蚀刻电子显微镜所观察到的“小的融合孔发生概率远低于大的融合孔”现象相吻合【6】。3、应用举例:稀疏解卷积是提升荧光显微镜分辨率的通用方法与当下热门的深度学习超分辨率显微重建不同,信号的空时连续性、高空间分辨率导致的荧光图像相对稀疏性这两个先验知识,是荧光显微成像的通用先验知识,不依赖于样本的形态以及特定的荧光显微镜种类。因此,稀疏解卷积是通用荧光显微计算超分辨率成像算法,可被广泛应用于提升其他荧光显微模态分辨率,观察不同种类细胞器的精细结构及动态(图3)。图3 | 稀疏解卷积广泛应用于提升不同显微成像模态空间分辨率,揭示各类细胞器精细结构动态。比如稀疏解卷积增强的商业超分辨转盘共焦结构光显微镜(SD-SIM)【7】,可以实现XY方向90纳米,Z方向250 纳米的空间分辨率,清晰记录分裂期7 μm深度内的全细胞内所有线粒体外膜网络(图4)。同样,若稀疏解卷积增强与商业SD-SIM结合,可以很容易实现活细胞上的三维、四色超分辨率成像。稀疏解卷积可以与膨胀显微镜(ExM)【8】结合,解析细胞膨胀后的复杂结构;也可以与宽场、点扫描的共聚焦、受激辐射损耗显微镜(STED)【9】以及微型化双光子显微镜(FHIRM-TPM 2.0)【10】结合,实现近两倍的空间分辨率提升。因此,稀疏解卷积的提出,将帮助使用各种各样荧光显微镜的生物医学研究者更好地分辨细胞中的精细动态结构。图4 | Sparse SD-SIM解析活细胞三维线粒体外膜网络。(k)活体COS-7细胞的线粒体外膜(Tom20-mCherry标记)的三维分布,颜色表征深度。(l)SD-SIM原始数据与Sparse SD-SIM的水平(左)和垂直(右)的白色框区域放大展示。比例尺:(k)5 μm;(l)1 μm。总之,通过稀疏解卷积算法(Sparse deconvolution)来实现计算荧光超分辨率成像,与目前基于特定物理原理或者特殊荧光探针的超分辨率方法都不相同。与超快结构光超分辨显微镜结合形成的Sparse-SIM是目前活细胞光学成像中,分辨率最高(60纳米)、速度最快(564帧/秒)、成像时间最长(1小时以上)的超分辨光学显微成像手段。它也可以与现有的多数商业荧光显微镜结合,有效提升它们的空间分辨率,看到更清楚的精细结构动态。哈尔滨工业大学博士生赵唯淞、北京大学博士后赵士群、李柳菊为共同第一作者,哈尔滨工业大学仪器科学与工程学院李浩宇教授和北京大学未来技术学院陈良怡教授为论文共同通讯作者,共同作者还包括哈尔滨工业大学谭久彬院士、刘俭教授,北京大学毛珩博士,生科院成像平台单春燕博士和华南师范大学刘彦梅教授。参与合作的实验室包括武汉大学宋保亮教授、北京大学陈兴教授、中科院国家纳米科学中心丁宝全教授和生物物理所纪伟教授等。该项工作得到北京大学膜生物学重点实验室、麦戈文脑研究所、北大-清华生命科学联合中心、北京智源人工智能研究院的支持,也是多模态跨尺度国家生物医学成像设施建设过程中的重要成果。专家点评徐平勇(中科院生物物理所)自2014年诺贝尔化学奖授予了超分辨显微技术以来,超分辨成像技术取得了巨大的进步,成像的分辨率得到了进一步的提高。在固定细胞中,以MINFLUX、SIMFLUX以及ROSE等为代表的超分辨成像技术利用调制光照射单分子定位的方法实现了小于10纳米的空间分辨率。然而,在活细胞中进一步提高成像的空间分辨率仍然面临挑战。一个主要原因是活细胞成像的时空分辨率是互相关联的,为了减少活细胞里的运动伪影,需要通过提高采样频率来提高时间分辨率,但是采样频率或者时间分辨率的提高会减少记录的光子数,使得空间分辨率下降。在现有超分辨成像技术中,结构光照明成像SIM技术具有最高的时间分辨率,但是受限于成像原理本身和所采用的维纳反卷积等算法,空间分辨率进一步提高遇到了挑战。陈良怡和李浩宇团队合作发展的稀疏结构光超分辨显微成像技术(Sparse-SIM),保留了陈良怡团队前期发展的海森-SIM的高时间分辨率的优点,并进一步将SIM的空间分辨率提高到60纳米。该技术属于计算超分辨率成像方法,主要包括两步迭代解卷积求解算法。其核心是将Richardson–Lucy反卷积算法应用到SIM成像中,通过前期发展的基于信号的时空连续性的先验知识重建图像的方法减少或者消除Richardson–Lucy反卷积应用中的噪声问题;并利用提出的“荧光图像的分辨率提高等价于图像的相对稀疏性增加”这个先验知识作为约束条件,建立通用的计算框架——稀疏解卷积技术。该工作有几个方面的突破和创新:1)解决了Richardson–Lucy反卷积应用到生物成像中的噪声和先验知识问题,拓展了它在生物成像中的实际应用;2)利用稀疏结构光超分辨成像在活细胞中实现了同时高时空分辨率长时程成像;3)方法具有普适性,可以广泛用于宽场成像和其它超分辨成像技术,提高这些成像方法的分辨率。目前发展的Sparse-SIM主要是基于二维结构光 (2D-SIM) 系统,实现了活细胞中空间分辨率60nm、时间分辨率564Hz、成像时间1小时以上的超分辨成像。这是目前活细胞成像中同时具有的最高时空分辨率。其空间分辨率可与非线性SIM相媲美,但是时间分辨率更高,成像设备上的复杂程度也相对要低一些。将来Sparse-SIM技术也有望能用于三维结构光成像,尽管受限于3D-SIM成像方法本身成像的时间分辨率会有所下降。总之,Sparse-SIM技术同时具有高的时间和空间分辨率,其在活细胞成像中的应用有望带来诸多生物学中的重要发现。尤其重要的是,稀疏解卷积技术框架适用于目前多数荧光显微镜成像方法,并将这些成像的空间分辨率提升了近两倍,将大大促进这些荧光成像方法的发展和它们在生物学中的广泛应用。刘兴国(中科院广州生物医药与健康研究院)以SIM、STORM/PALM、STED为代表的的超分辨成像技术,成功突破了光学衍射极限,极大推动了亚细胞结构和细胞器互作动态等微观结构研究,获得了2014年诺贝尔化学奖。然而超分辨成像技术在时间分辨率和空间分辨率上难于获得同等提高——在超分辨成像技术中,SIM技术具有最好的时间分辨率,然而空间分辨率也是3种主流技术中最低的,缺乏对100nm以下尺度的亚细胞器结构的解析力。在充分利用SIM技术的时间分辨率的基础上,如何提高空间分辨率是一个重要的研究方向。北京大学陈良怡团队与哈尔滨工业大学李浩宇教授在Nature Biotechnology 杂志报道最新开发的Sparse deconvolution算法,并成功结合SIM技术开发出Sparse-SIM,在时空分辨率上成功将SIM技术的空间分辨率从110nm提高到60nm,同时保持毫秒级的时间分辨率。同时,陈良仪团队研究显示,本技术同样可以提高SD-SIM、STED等超分辨技术的轴向分辨率,甚至可以使普通宽场显微镜获得更好的信噪比。这一精彩的工作不但是领域的重要技术进展,而且具有广阔的应用空间。 陈良怡团队之前的工作,在硬件和软件水平挖掘SIM技术的时空分辨率,成功开发了高时空分辨率的Hessian SIM技术;本次研究再次在软件算法上取得突破,进一步推动了SIM技术在活细胞超分辨成像在时空分辨率的极限。应用Sparse-SIM技术,同时检测了核孔复合物结构、网格蛋白(clathrin)动态、溶酶体和内质网相互作用、内质网对线粒体内嵴动态的调控等重要过程,显现出Sparse-SIM强大的应用能力和应用前景。如何易于操作的提高超分辨成像技术的时空分辨率是亚细胞器结构和动态研究方面的一个重要方向,Sparse deconvolution算法或者Sparse-SIM提供了一个重要的生命科学研究工具,去探索更微观的生命科学过程。参考文献[1] Weisong Z, Shiqun Z, Liuju L, et al. Sparse deconvolution improves the resolution of live-cell super-resolution fluorescence microscopy [J]. Nature biotechnology, 2021: DOI: https://doi.org/10.1038/s41587-021-01092-2.[2] Huang X, Fan J, Li L, et al. Fast, long-term, super-resolution imaging with Hessian structured illumination microscopy [J]. Nature biotechnology, 2018, 36(5): 451-459.[3] Gu L, Li Y, Zhang S, et al. Molecular resolution imaging by repetitive optical selective exposure [J]. Nature Methods, 2019, 16(11): 1114-1118.[4] Szymborska A, Marco A d, Daigle N, et al. Nuclear pore scaffold structure analyzed by super-resolution microscopy and particle averaging [J]. Science, 2013, 341(6146): 655-658.[6] Ornberg R L, Reese T S. Beginning of exocytosis captured by rapid-freezing of Limulus amebocytes [J]. The Journal of Cell Biology, 1981, 90: 40 - 54.[7] Schulz O, Pieper C, Clever M, et al. Resolution doubling in fluorescence microscopy with confocal spinning-disk image scanning microscopy [J]. PNAS, 2013, 110(52): 21000-21005.[8] Sun D-E, Fan X, Shi Y, et al. Click-ExM enables expansion microscopy for all biomolecules [J]. Nature Methods, 2021, 18: 107–113.[9] Hell S W, Wichmann J. Breaking the diffraction resolution limit by stimulated emission: stimulated-emission-depletion fluorescence microscopy [J]. Optics Letters, 1994, 19(11): 780-782.[10] Zong W, Wu R, Chen S, et al. Miniature two-photon microscopy for enlarged field-of-view, multi-plane and long-term brain imaging [J]. Nature Methods, 2021, 18(1): 46-49.