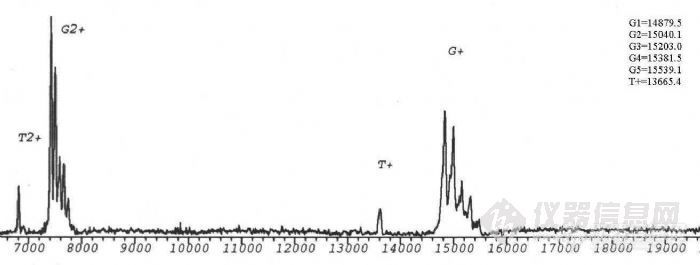
生物质谱在糖蛋白结构分析中的应用项目完成人:桑志红 蔡 耘项目完成单位:国家生物医学分析中心 随着人们对糖蛋白参与生命活动机理的日益深入了解,对天然糖蛋白及重组糖蛋白类药物的分析越来越受到重视。重组糖蛋白类药物的质量控制更是直接关系到药物的疗效及至人类的健康。九十年代以来,随着带有反射功能的基质辅助激光解吸附电离飞行时间质谱(MALDI-TOF-MS)和纳升电喷雾串联质谱(nano-ESI-Q-TOF)等具有软电离方式的现代质谱 技术的发展,质谱以其高灵敏度和强有力的分析混合物的能力,提供了生物大分子的分子量、序列、一级结构信息以及结构转换、修饰等方面的信息,使糖基化分析有了重要的进展。 通常研究糖蛋白的方法是把蛋白链上的寡糖切下来,分别研究蛋白部分和寡糖部分的结构,因此无法研究与两部分共同相关的结构问题,也不能区分不同糖基化位点上切下来的寡糖。自90年代初,国外有人开始用质谱法研究糖蛋白的结构,同时描述了各个位点的不均一性。我们用建立的现代生物质谱技术研究糖蛋白一级结构的方法,将其应用与基因重组糖蛋白的结构分析。为糖蛋白结构分析及基因重组糖蛋白类药物的质量控制提供新的手段。一、 生物质谱研究糖蛋白结构方法的建立实验所用仪器为:1.德国BRUKER 公司的REFLEXIII型基质辅助激光解吸附电离飞行时间质谱仪,N2激光器,波长337nm,线性飞行距离150cm,加速电压2kv。2.英国Micromass 公司Q-TOF型电喷雾串联质谱仪。源温80°C,气体流速40L/h,枪头电压650V,检测频率2.4S,氩气碰撞池压力6*10-5mbar。1. 基质的选择,在MALDI-TOF-MS分析中,基质起着相当重要的作用。不同的基质对不同类的物质响应不同,a-氰基-4-羟基肉桂酸用于测定糖蛋白核糖核酸酶B效果相对较好。2. 糖蛋白分子量的测定,糖蛋白核糖核酸酶B由124个氨基酸组成,在34位Asn处连有一个高甘露糖型N-糖链。由于糖链的微不均一性,与普通蛋白质及核酸不同,其分子离子峰在MALDI-TOF-MS 质谱图上表现为一簇峰,各峰之间约相差一个糖基。正是由于这种微不均一性,使得其分子离子峰变宽,灵敏度降低。糖链分子量越大,峰越宽,灵敏度越低,所以一般只有糖链较短,蛋白的质量不太大的糖蛋白才能测定其平均分子量。用MALDI-TOF可直接测定糖蛋白核糖核酸酶B的平均分子量为 15208.6Da。http://ng1.17img.cn/bbsfiles/images/2011/03/201103211511_284179_1604317_3.jpg3. 糖含量的测定,采用O聚糖酶及内糖苷键酶F分别作用于核糖核酸酶 B,只有内糖苷键酶F能够是其分子量发生变化,表明核糖核酸酶B分子中不存在O-连接糖链存在着N-连接糖链。内糖苷键酶F切断N-糖链五糖核心最内侧的GlcNAc-GlcNAc糖苷键,得到含一个GlcNAc的肽链,减去GlcNAc,可以计算出准确的肽链分子量T=13695.6,与糖蛋白平均分子量之差为糖链的平均分子量G=1513.4,平均糖含量为:(糖链大小/糖蛋白分子量)×100%=9.95%。4. 糖基化位点的确定,研究糖基化类型及糖基化位点的策略:采用蛋白酶酶解与糖苷内切酶酶解相结合的方法,通过酶切前后含糖肽片的位移,结合网上数据库检索,可以确定糖基化类型和糖基化位点。以不同类型的糖苷内切酶作用于糖蛋白(N-糖苷键酶或O-糖苷键酶),在MALDITOF-MS 上观察其质量的变化,可以直接确定糖蛋白中是否含有响应类型的糖链,这是我们确定糖蛋白中糖苷键类型的基础。我们采用先将核糖核酸酶B还原烷基化,加Glu-C酶切,产物再用内糖苷肩酶F酶切,可观察到含糖肽段出现位移,将核糖核酸酶B的肽质量指纹图进行数据库检索,证实发生位移的肽段中含有N-糖链特异连接位点,由此确定34位Asn为糖基化位点。另外我们采用内糖苷键酶F及肽-N-聚糖酶F两种酶进行差位酶切法对含糖肽段进行验证,两种酶酶切后分子离子峰的差值除以GlcNAc的质量,结果就是N-糖基化位点的个数5. 质谱测定氨基酸序列, 我们对核糖核酸酶B肽质量指纹谱中的含糖肽段进行了串联质谱测定,首先在一级质谱图中选择离子4972.23,在串联质谱的碰撞活化室以氩气与其碰撞产生碎片,从碎片的质荷比推算出此肽片中的一段氨基酸序列,检索结果为核糖核酸酶B,从而判断其理论序列是否一致。6. 糖链结构的研究,凝集素对糖肽的亲和提取,进一步分析糖肽序列及糖链结构的关键是含糖肽段的提取。核糖核酸酶B中糖链为高甘露糖型,我们选用对其有特异性吸附的伴刀豆球蛋白对其进行提取利用这种简捷的亲和质谱的方法,对糖肽段进行了分析。建立了亲和质谱分析糖肽类物质的方法,为今后糖肽序列分析及糖链结构分析奠定了基础。二、基因重组糖蛋白人促红细胞生成素(rhEPO)的结构分析。 利用以上建立的方法,我们对样品重组人促红细胞生成素进行了分析,断定此样品为非完全糖基化,样品中只存在N-连接的糖链,无O-糖链。应用酶切法用肽-N-聚糖酶处理后,得到两个含糖肽段,进行数据库检索,测得38位及83位为N-糖基化位点,与文献报道相符,结果可靠。因此,该项课
褚福亮,王福生, 中国人民解放军第302医院全军艾滋病与病毒性肝炎重点实验室 北京市 100039项目负责人 王福生, 100039 ,北京市丰台路26号, 中国人民解放军第302医院全军艾滋病与病毒性肝炎重点实验室. fswang@public.bta.net.cn电话:010-66933332 传真:010-63831870收稿日期 2002-08-15 接受日期 2002-09-03摘要新近广泛应用蛋白质芯片(ProteinChipâ Array)系统成功鉴定出了一些重要疾病(如肿瘤和危害性较大的传染病)新的、特异性的生物标记(biomarkers),后者不仅在生物医学的基础方面具有重要的科学价值,而且在临床疾病的诊断、治疗和预防发挥重要的指导作用,显示了良好的发展前景.本文就表面增强的激光解析电离-飞行时间-质谱(SELDI-TOF-MS)相关的原理、特点、在临床和基础研究中的应用新进展和未来的发展趋势做一综述.此外,我们就蛋白质谱分析技术在病毒性肝炎、肝硬化和肝癌等一系列肝病方面的应用策略和前景进行了分析.褚福亮,王福生. 蛋白质谱分析方法特点及其在蛋白组学研究领域中的应用.世界华人消化杂志 2002 10(12):1431-14350 引言人类基因组计划已经进入后基因组时代-即功能基因组时代[1],作为基因功能的直接体现者-蛋白质,及其之间的相互作用越来越引起基础和临床科学家们的关注[2-6] .因为要彻底了解生命的本质,只把基因测出来还是不够的,还必须要了解其在生物生长、发育、衰老和整个生命过程中的功能、不同蛋白质之间的相互作用以及他们与疾病发生、发展和转化的规律[7-14] .正因为如此,有关上述问题的蛋白质组学研究成了今天生命科学最重要的焦点之一[15] .为了阐明蛋白质在上述生命现象中的作用和相关机制,人们设计了许多新的方法技术,如:二维电泳、质谱分析、微距阵列、酵母双杂交和噬菌体展示等,这些方法在一些特定的情况下,虽然显示出了他们各自不同的优点,但是同样也存在着较大的局限性,难以开展大规模、超微量、高通量、全自动筛选蛋白质等方面的分析,因而设计更全面、同时研究多种蛋白质相互作用的技术,在功能基因组和蛋白组学的研究中建立一个更有效的技术平台,成为本领域中优先关注的问题[16] .近来,美国Ciphergen(赛弗吉)公司研制的ProteinChipâ Array的仪器,并建立了一种新的蛋白质飞行质谱-表面增强的激光解析离子化-飞行时间-质谱(surface-enhanced laser desorption/inionation-time of flight-mass spectra, SELDI-TOF-MS),已取得可喜的进展,筛选出了许多与疾病相关的新型生物标志,不仅为临床疾病的诊断和治疗等提供了新的选择,而且在基础科学、新药研制和疾病预防等方面具有广泛的应用前景[16-18] .本文就SELDI-TOF-MS相关的原理、特点、在临床和基础研究中的应用新进展和未来的发展趋势做一综述.1 ProteinChipâ Array系统和SELDI-TOF-MS的特点1.1 蛋白质芯片系统的组成和原理 蛋白质芯片系统由三部分组成:蛋白质芯片、芯片阅读器和芯片软件.供研究用芯片上有6-10芯池,不同的芯片表面上的化学物质不同,芯片表面分为两大类:一类为化学类表面,包括经典的色谱分析表面,如:结合普通蛋白质的正相表面,用于反相捕获的疏水表面,阴阳离子交换表面和捕获金属结合蛋白的静态金属亲合捕获表面;另一类称为生物类,特定的蛋白质共价结合于预先活化的表面阵列,可以用来研究传统的抗体一抗原反应,DNA和蛋白质作用,受体、配体作用和其他的一些分子之间的相互作用[19] . 根据检测目的不同,可以选用不同的芯片,或者自己设计芯片.将样本和对照点到芯池上以后,经过一段时间的结合反应,用缓冲液或水洗去一些不结合的非特异分子,再加上能量吸收分子(energy absorbing molelule,EAM)溶液,使样本固定在芯片表面.当溶液干燥后,一个含有分析物和大量能量吸收分子“晶体”就形成了.能量吸收分子对于电离来说非常重要.经过以上步骤,就可经把芯片放到芯片阅读器中进行质谱分析. 在阅读器的固定激光束下,芯片上、下移动,使样本上每一个特定点都被“读”到.激光束的每一次闪光释放的能量都聚集在该区一个非常小的点上(focused laser beam,聚焦激光束).这样,每个区都含有丰富的,可寻址(addressable)的位置.蛋白质芯片处理软件精确控制激光寻读过程.当样本受到激发,就开始电离和解除吸附.不同质量的带电离子在电场中飞行的时间长短不同,计算检测到的不同时间,就可以得出质量电荷比,把他输入电脑,形成图像[19].Ball et al [20]采用一种称为人工神经网络(artifical neural network,ANN)的算法处理出现的成千上万的峰,鉴定出三个分子量为13 454、13 457和14 278的生物标记分子,使疾病预测率达到97.1 %.1.2 ProteinChipâ Array芯片和SELDI-TOF-MS的特点 新型蛋白芯片与以往的蛋白芯片不同之处:SELDI-TOF-MS,他是在MALDI(matrix-assisted laser desorption/inionation)[21,22]基础上,改进后实行表面增强的飞行质谱.SELDI-TOF-MS优于MALDI-TOF表现为他不会破坏蛋白质,或使样本与可溶的基质共结晶来产生质谱信号.对SELDI-TOF来说,可以直接将血清、尿液、组织抽取物等不需处理直接点样检测[40] 由于一部分非特异结合的分析物被洗去,因而出现的质峰非常一致,有利于后期分析[23,24] . 与二维电泳相比:二维电泳分析蛋白质的分子量在30 KDa以上时电泳图谱较清楚,对在组织抽提物中占很大比例的低丰度的蛋白质不能被检出;其次,二维电泳胶上的蛋白质斑点很大一部分包含一种以上的蛋白质;而且,二维电泳耗时长,工作量大,对象染色转移等技术要求高,不能完全实现自动化.而SELDI-TOF在200 Da-500 KDa区间都可以给出很好的质谱,对一个样本的分析在几十分钟内就可以完成[19],处理的信息量远远大于二维电泳;对于低丰度物质,即使浓度仅attomole(10-18)的分子,只要与表面探针结合,就可以检测到,这也是二维电泳所不具备的[24,25] . 对于微距阵蛋白芯片来说,需要一种不破坏折叠的蛋白质构象的固定技术,再与另外的蛋白质反应,经检测莹光来观察蛋白质之间的作用[26] .而基于SELDI-TOF-MS的ProteinChip分析蛋白质不需溶解、不需染色、廉价、针对性强. 因而蛋白质芯片仪具有以下优势:(1)可直接使用粗样本,如:血清、尿液、细胞抽提物等[27] .(2)使大规模、超微量、高通量、全自动筛选蛋白质成为可能;(3)他不仅可发现一种蛋白质或生物标记分子,而且还可以发现不同的多种方式的组合蛋白质谱,可能与某种疾病有关[28] (4)推动基因组学发展,验证基因组学方面的变化,基于蛋白质特点发现新的基因.可以推测疾病状态下,基因启动何以与正常状态下不同,受到那些因素的影响,从而跟踪基因的变化[2,14,15] . 其存在的问题:对于不同的样本,根据检测的目标采取或者设计几种芯片,理论上可以把所有的相同性质蛋白质捕获,但是实际上仍有少量的分子没与表面探针结合.使用SELDI-TOF-MS,仅能给出蛋白质的分子量,不能给出C端、N端的序列,也没法知道蛋白质的构型,因此需要将蛋白质充分纯化后,用蛋白酶消化芯片上的蛋白质,分析肽段,再用生物信息学方法鉴定蛋白质序列[18,24] .另外,在国内,该芯片费用较高,分析质谱需要大量后续工作支持.
[摘要] 质谱对于现代蛋白质化学研究是一项重要的技术。也可用于蛋白组分析,在同一时间监控上千种蛋白的表达情况。首先蛋白质混合物可以用双向凝胶电泳分开,再用质谱及随后的蛋白质数据库检索对单个蛋白进行鉴定和分析。最近几年,质谱领域取得了令人鼓舞的进展,使得全自动、高通量的蛋白检测成为可能。质谱也可以用来分析蛋白的转录后调节以及研究蛋白质复合体。本文将介绍目前蛋白组研究中质谱方法的应用并对其优缺点进行讨论。关键词: 质谱;二维电泳(2-DE);蛋白组分析MALDI-TOF1 前 言蛋白组学是研究生物特定组织或细胞内表达的所有蛋白质成分,蛋白质组的一门学科。蛋白组分析一般是用双向凝胶电泳(2-DE),质谱(MS)以及数据检索来完成。2-DE要回顾到20世纪70年代初[1],但是用质谱技术对蛋白质进行鉴定和分析是在近十年内才成为可能。其中最重要的一个原因是新的软离子技术即基质辅助激光解析电离(MALDI)[2]和电喷雾电离(ESI)[3,4]技术的发展和应用,以及样本制备技术和质谱仪的发展等。从数据库中获得成指数上升的序列信息也促进了质谱技术的发展。跟转录组学中的全自动DNA微阵列技术相比,蛋白组分析需要更多的手工操作,尤其在2-DE上会出现很多问题[5,6]。尽管存在很多困难,蛋白组学的重要性还是公认的。DNA微阵列技术是建立在对mRNA稳定水平的检测上,然而蛋白组学研究的对象是细胞中最活跃的因子——蛋白质。最近有研究证明,mRNA的表达和蛋白的水平并非具有相关性[7,8]。蛋白质有转录后修饰过程,并会以不同的形式出现。而这些修饰过程是相应的DNA测序技术所不能预测的。质谱技术在蛋白质修饰分析过程中具有重要的作用。2 质谱对蛋白质的检测质谱仪是由离子源,质量分析,离子检测器,以及数据检索等单元组成。首先,被分析的分子在离子源中被离子化,然后在质量分析器里根据不同的质荷比分开,再对分开的离子进行检测。随着MALDI和EIS的出现,质谱技术已经广泛用来分析蛋白质。质量分析器有很多种,最常用的方法是将飞行时间检测器(TOF)与MALDI或三级四级串联质谱仪联用,或者将四级杆-TOF,离子阱等连接到ESI上。蛋白组分析中,可以用两种不同的方法检测电泳分离后的单个蛋白质。最简单的方法叫做肽指纹谱测定(PMF)[9~13]。这个方法是用特殊的酶对2-DE分离后的蛋白质点进行降解,产生的肽从凝胶中分离出来后再用质谱分析和检测肽的分子量。数据库检测能够产生所有蛋白质理论上的PMF,把它们和质谱分析所获得的肽片进行比较就可以得出结果。第二种方法是在质谱仪中把凝胶分离后的肽分解成片段,产生局部的氨基酸序列(序列标签)。那么就可以用分子量或者序列信息进行数据库检索[14,15]。PMF一般用MALDI-TOF来实现,序列标签可以用串联质谱技术MS-MS进行检测[16~18]。用质谱检测蛋白的灵敏性可以精确到飞摩的水平,甚至已有报道其精确度可以达到阿摩水平[19]。3 用MALDI-TOF进行肽指纹谱分析凝胶分离后的蛋白质鉴定以及肽指纹谱分析是目前质谱实验室常规使用的方法。在进行MALDI分析之前需要最优化的凝胶上消化[20~22]和脱盐过程。胰岛素是最常用的凝胶消化酶,因为它在赖氨酸和精氨酸位点能够特异性切割蛋白质,产生分子量在600~2500Da之间的小分子肽,并能够从凝胶上有效的分离出来。MALDI-TOF是质谱技术中相对简单并很容易使用的一种方式,对样本中的盐和其他污染物有很高的耐受性,这点优于ESI。提取的肽片混合物中较小的片段可以直接沉积在靶板上做PMF分析,剩下的样本可以储存起来作为以后的ESI-MS分析。如果经凝胶分离后所有的肽全用来做MALDI分析,那么样本经脱盐以后将会取得更好的结果。脱盐的过程可以用商品化的试剂盒ZipTip(Millipore),或者在凝胶电泳仪的末端放置一个小的逆向塑料柱来实现[23,24]。PMF中非常重要的一个因素就是质谱测量分子量的准确度[25]。现代的MALDI-TOF仪都具有延迟取样反射器装置,用它们检测的肽段分子量可以精确到10~30ppm。这样的精确度使得4~5个肽段足以清楚的鉴定蛋白质的分子量。MALDI产生的大多数都是单电荷离子,所以它的图谱很容易解释。4 肽质指纹谱分析的自动化为了实现高通量蛋白检测,样本准备,质谱检测,资料分析和数据检索必须实现自动化操作。目前有些实验室用机器人对蛋白质进行自动化取点,凝胶上消化,样本脱盐以及对MALDI平板进行定点测定等。而且,现代化的MALDI-TOF仪完全能够让MALDI检测自动化。资料分析和数据检索过程同样可以自动化。在进行蛋白组分析时,如果在凝胶上不用单个分离就可以对所有蛋白质进行鉴定将是非常具有诱惑力的。为此,研究人员做了很多努力,由Hochestrasser及其同事建立了一套称作分子探测术的方法(molecular scanner)[26,27]。它是将整个2-DE胶上蛋白质样本同时酶解,并将酶解片段一起原位转移至另一张膜上,再直接用MALDI- TOF 质谱对膜上样本进行整体扫描,为实现蛋白质组学研究的自动化、规模化以及临床应用提供了一个崭新的思路和方法。这种思路的优点是显而易见的,不过按照目前的方法,要普及此种技术主要的难点是,质谱扫描一张图谱所需的时间过长,如用0.4mm的精度扫描一张4cm×4cm的膜需55个小时,这就意味着一张16cm×16cm的常用膜的扫描,至少需要36天不间断的工作和大约40G的磁盘空间存储如此庞大的原始数据。5 用ESI-MS/MS的方法创建序列标签只有当被消化的蛋白存在于已有的蛋白或基因组数据库中,并且能从MALDI分析器中得到四五个肽片的数据才能成功进行。如果在EST数据库中只有目的蛋白的部分序列,那么就需要知道肽片的序列信息。创建序列标签的优势在于,用肽的分子量和序列作为数据库检索对象比只用分子量作为检索对象具有更高的特异性。有了序列标签信息后,也可以用EST数据库进行检索[28]。通常来自于一个肽上的序列标签就足以鉴定蛋白质。同MALDI相比,纳升-ESI-MS/MS费时费力,而且在做ESI分析之前必须对样本进行脱盐处理。这些问题可以通过ESI-MS与HPLC联用得到解决。做ESI分析对样本浓度很敏感,用现代化的纳升-HPLC方法可以提供很高的局部肽片凝聚物,分离后的样本可以直接用于质谱分析,而不需要再分解。当分析混合物的时候,在质谱之前做LC分离尤其重要,因为它使得数据依赖的实验(DDE)成为可能,在DDE中,所有的离子都进入检测器进行测量,如果从HPLC到质谱之间出现一个峰的话,软件将自动从质谱转换到MS/MS模式并对产生的离子进行扫描。跟纳升-ESI相比,用这个方法可以从一份标本产生更多的MS/MS数据。Yates等已经详细叙述了如何进行[url=https://insevent.instrument.com.cn/t/Yp][color=#3333ff]LC-MS[/color][/url]/MS全自动蛋白检测的策略[29]。
PS1利用基质辅助激光解吸电离-飞行时间(MALDI-TOF)技术来表征生物分子。样品溶于固定的底物中形成晶体,用激光脉冲使其离子化,离子被加速后通过飞行管时分离,所有离子均可被检测。系统包括三个组成部件:样品点样制备工作站(SymBiot 1)、生物质谱工作站(Voyager-DE PRO)和自动化分析软件(AutoMS-Fit)。SymBiot1 是一个自动样品处理系统,支持亚微升级微量点样,具有快速省时、重现性好的特点;Voyager-DE PRO是为蛋白质组研究专门设计的自动飞行时间质谱分析系统,配有AB公司之专利—延迟检测技术,具有高分辨率、质荷比宽等特点;AutoMS软件可以批处理方式或实时动态方式检索Protein Prospector蛋白数据库或您指定的蛋白数据库,查询参数可以任意设定,检索结果以Microsoft Access格式分类编号及储存。 PS 1技术平台建立伊始便受到了许多蛋白质课题研究组的关注。中国科学院上海生物化学研究所戚正武院士课题组从猪肝中提取某一活性蛋白组分,该组分理化性质不清楚,天然含量十分低,并无相关文献报道。用HPLC分离以后对活性组分的成分不能确定。上海基康生物技术有限公司运用PS 1系统对HPLC分离后的活性组分作了质谱分析,仅在一个工作日内就精确确定该组分由分子量极为相近的几种蛋白质构成,分子量精确度达到10 ppm。后经HPLC再次细分(洗脱梯度增加了2.5倍),证实了质谱的结论。此活性组分曾滤过1kD分子筛,基康的质谱数据纠正了研究人员过去对该活性组分分子量的误判,为研究人员明确实验方向、优化实验步骤提供了强有力的依据。 PS1除了可以进行生物大分子的精确分子量测定,还可用于蛋白的肽指纹图谱分析(peptide mass fingerprint,PMF),提供相关生物信息学服务,并且还可以利用源后衰变(Post Source Decay,PSD)技术来获得样品的MS/MS数据,以得到一级结构信息。PSD方法通常增加了激发激光的功率,使其超过产生一般肽指纹谱图所需功率的阈值,过剩的能量使前体离子在源内离子化之后发生裂解,产生一系列碎片离子,在反射器的作用下,最终可以得到一张连续的碎片离子图谱。经特定的软件分析后,即可在数据库中检索到肽段的氨基酸序列。利用PSD分析技术,还可以对磷酸化,糖基化等翻译后修饰进行定位分析,同样也可以鉴定产生翻译后修饰肽段的蛋白质。Neville et al.(1997)将这一方法成功的用于磷酸肽的序列分析。作为重要的蛋白质鉴定手段之一,PS1的精确度可以达到10 ppm,灵敏度为fmol,分子量检测范围可达到500 kDa,每天可自动分析40-100个样品,适用于大规模“蛋白质组学”研究。
求购 质谱分析的蛋白标准品,MALDI-TOF-MS上用的。 分子量在400--30000da 的或者接近这个质量范围的,如有请联系我。直接给我这个留言就可以,谢谢!
[color=#333333]质谱鉴定样品对蛋白的染色方法要求是什么?[/color]
2003年人类基因组精细图绘制完成,是人类科学史上一个里程碑式的事件。后基因组时代的研究重点自然落在了蛋白质头上。为啥?因为中心法则告诉我们,基因的产物——蛋白质,是生命活动的最终执行者。与基因组类比,研究生物体内全套蛋白质的科学,就是蛋白质组学。基因组计划完成的同年,人类蛋白质组计划启动,令人激动的是,2014年人类蛋白质组的草图也完成了。而蛋白质组学能够飞速发展的最大功臣非质谱莫属。质谱的应用范围非常广泛,但这里只讨论蛋白质组学中的质谱。简单地说,质谱法(mass spectrometry)就是对肽段离子的重量(质荷比,m/z)进行测量的分析方法。样品经质谱仪(mass spectrometer)检测得到质谱图(mass spectrum),通过对质谱图的分析就可以对样品中的蛋白进行鉴定、定量。亲,图1的这种典型的蛋白质组学流程都很熟悉吧。蛋白首先都要被特异性的酶(通常为Trypsin)切割为肽段,再进行后续分析,这在蛋白质组学中被称为“自下而上”的研究策略(Bottom-up proteomics)。我们平时见到的质谱分析基本都是这种类型。提到蛋白质组,即会联想到一系列高大上的名词,iTRAQ、SWATH、SILAC、Shotgun、Label-free等等。很多概念容易弄混淆,下面我们就来理理清楚。图1. 典型的蛋白质组学流程大体上,质谱研究蛋白主要是鉴定和定量。通过二级质谱图(MS2或者MS/MS)进行数据库搜索匹配鉴定蛋白。通过各种标记或非标记的手段对不同样品中的蛋白进行比较就是定量。蛋白定量比较是质谱最重要的用途,图2是对定量方法的一个简单总结。非标定量(Label-free)不需要标记,不同样品分别处理、分别进质谱检测;优点是处理简单、无需标记、价格便宜、可以比较很多组样品,缺点是对操作步骤、LC、质谱稳定性要求严格。SILAC是在细胞培养基中加入稳定同位素标记的氨基酸,在代谢水平标记蛋白,一级质谱图进行定量,可以做到三组样品混合后进行比较,定量准确,但是不能标记组织样本,养细胞成本也较贵。双甲基化标记是通过化学反应的办法在肽段水平进行标记,一级质谱定量,也可以三组对比,标记试剂都比较便宜,而且可以标记任何来源的样品。iTRAQ和TMT是商品化的试剂盒,肽段水平标记,二级质谱定量;分别可以做到最多8组和10组样品间蛋白质组的比较。图2. 质谱定量方法以上这几个是一家的,还有几个名词是属于另外一家,比如Shotgun (DDA)、SWATH/DIA、SRM (MRM)、MRMHR/PRM。质谱进行数据采集的方式大致分为三种:鸟枪法(Shotgun)、选择反应监控(SRM)和全景式的SWATH/DIA。下面对照图3再来简单介绍一下。图3. 质谱扫描方式DDA、IDA、Shotgun和鸟枪法说的是相同的东西,意思是质谱在每个循环的中从一级里挑选丰度高的TopN个肽段去打碎做二级扫描,得到的结果通过与已知数据库中的理论蛋白进行匹配。DDA简单有效,分析流程比较成熟,也是目前质谱分析的主流方式。DDA也有其固有的缺陷,即具有一定的随机性,偏向于检测丰度较高的肽段,而抑制了低丰度肽段的检测。靶向策略被称为质谱领域的Western blot。质谱只去采集目标肽段大小的离子信息,因而提高了灵敏度和特异性。这种方法用来研究感兴趣的特定蛋白,定量准确,但是通量很有限。SWATH/DIA这种全景式的数据采集方式在最近几年突然火了起来,被认为在不远的未来可能会取代DDA的主流位置。该方法采取的策略是将扫描范围内的所有肽段按照质荷比分为若干个窗口,再对每个窗口里所有的肽段一起打碎,采二级,数据分析时通过抽提蛋白的子离子信息进行定量。SWATH/DIA解决了DDA中随机性选择肽段的缺陷,所以重复性更好,定量的准确性基本达到了SRM的水平,而且可以实现大规模定量。借用听来的一个比喻来说明:DDA就像机关枪扫射,数量多、体积大的目标命中的概率要大一些。靶向扫描(SRM或PRM)就像精准狙击,排除干扰,目标明确,每一枪直指目标,但是难以大规模消灭敌人。SWATH/DIA就是地毯式轰炸,只要暴露在我方攻击范围内的敌人,不管三七二十一,全部炸完。图4. 定量方法与采集方式结合如果将上述的定量方法(图2)和质谱数据采集方式(图3)结合起来,就得到了现在基于质谱的蛋白质组学研究的各种策略(图4)。再打个比方,保证吃货们一听就懂:鸡、鱼、肉、蛋、蔬菜要通过炒锅、烤箱、高压锅、微波炉等烹调之后才能变为美食,填饱肚子。同样的,各种定量方法(非标的和标记的)处理的样品,要通过质谱各种采集方式变为电脑中的数据,才能分析并从中得到蛋白的信息。本次的介绍就先到这里了,如果其中有什么问题,欢迎您批评和建议,我们会努力变得更好;如果需要跟我们进行技术交流和讨论,欢迎大家联系武汉金开瑞。后续我们还会继续推出对质谱技术各方面进行解析的文章,敬请期待。ReferencesA draft map of the human proteome. Nature 509: 575–581 (2014)Mass-spectrometry-based draft of the human proteome. Nature 509: 582–587 (2014)A review: Annu. Rev. Biochem. 80: 273–99 (2011)SILAC: Molecular & Cellular Proteomics 1: 376-386 (2002)iTRAQ: Molecular & Cellular Proteomics 343: 91–99 (2010)SRM: Nature Methods 9: 555–566 (2012)SWATH: Molecular & Cellular Proteomics 11: 1–17 (2012)
几种常用的蛋白鉴定方法传统的蛋白鉴定方法,如免疫印迹法、内肽的化学测序、已知或未知蛋白的comigration分析,或者在一个有机体中有意义的基因的过表达通常耗时、耗力,不适合高流通量的筛选。 目前,所选用的技术包括对于蛋白鉴定的图象分析、微量测序、进一步对肽片段进行鉴定的氨基酸组分分析和与质谱相关的技术。1 图象分析技术(Image analysis)“满天星”式的2-DE图谱分析不能依靠本能的直觉,每一个图象上斑点的上调、下调及出现、消失,都可能在生理和病理状态下产生,必须依靠计算机为基础的数据处理,进行定量分析。 在一系列高质量的2-DE凝胶产生(低背景染色,高度的重复性)的前提下,图象分析包括斑点检测、背景消减、斑点配比和数据库构建。 首先,采集图象通常所用的系统是电荷耦合CCD(charge coupled device)照相机;激光密度仪(laser densitometers)和Phospho或Fluoroimagers,对图象进行数字化。 并成为以象素(pixels)为基础的空间和网格。 其次,在图象灰度水平上过滤和变形,进行图象加工,以进行斑点检测。 利用Laplacian,Gaussian,DOG(difference of Gaussians) opreator使有意义的区域与背景分离,精确限定斑点的强度、面积、周长和方向。图象分析检测的斑点须与肉眼观测的斑点一致。 在这一原则下,多数系统以控制斑点的重心或最高峰来分析,边缘检测的软件可精确描述斑点外观,并进行边缘检测和邻近分析,以增加精确度。 通过阈值分析、边缘检测、销蚀和扩大斑点检测的基本工具还可恢复共迁移的斑点边界。 以PC机为基础的软件Phoretix-2D正挑战古老的Unix为基础的2-D分析软件包。 第三,一旦2-DE图象上的斑点被检测,许多图象需要分析比较、增加、消减或均值化。 由于在2-DE中出现100%的重复性是很困难的,由此凝胶间的蛋白质的配比对于图象分析系统是一个挑战。 IPG技术的出现已使斑点配比变得容易。 因此,较大程度的相似性可通过斑点配比向量算法在长度和平行度观测。 用来配比的著名软件系统包括Quest,Lips,Hermes,Gemini等,计算机方法如相似性、聚类分析、等级分类和主要因素分析已被采用,而神经网络、子波变换和实用分析在未来可被采用。 配比通常由一个人操作,其手工设定大约50个突出的斑点作为“路标”,进行交叉配比。 之后,扩展至整个胶。例如:精确的PI和MW(分子量)的估计通过参考图上20个或更多的已知蛋白所组成的标准曲线来计算未知蛋白的PI和MW。 在凝胶图象分析系统依据已知蛋白质的pI值产生PI网络,使得凝胶上其它蛋白的PI按此分配。 所估计的精确度大大依赖于所建网格的结构及标本的类型。 已知的未被修饰的大蛋白应该作为标志,变性的修饰的蛋白的PI估计约在±0。25个单位。 同理,已知蛋白的理论分子量可以从数据库中计算,利用产生的表观分子量的网格来估计蛋白的分子量。 未被修饰的小蛋白的错误率大约30%,而翻译后蛋白的出入更大。 故需联合其他的技术完成鉴定。2 微量测序(microsequencing)蛋白质的微量测序已成为蛋白质分析和鉴定的基石,可以提供足够的信息。 尽管氨基酸组分分析和肽质指纹谱(PMF)可鉴定由2-DE分离的蛋白,但最普通的N-末端Edman降解仍然是进行鉴定的主要技术。 目前已实现蛋白质微量测序的自动化。 首先使经凝胶分离的蛋白质直接印迹在PVDF膜或玻璃纤维膜上,染色、切割,然后直接置于测序仪中,可用于subpicomole水平的蛋白质的鉴定。 但有几点需注意:Edman降解很缓慢,序列以每40 min 1个氨基酸的速率产生;与质谱相比,Edman降解消耗大;试剂昂贵,每个氨基酸花费3~4$。 这都说明泛化的Edman降解蛋白质不适合分析成百上千的蛋白质。 然而,如果在一个凝胶上仅有几个有意义的蛋白质,或者如果其他技术无法测定而克隆其基因是必需的,则需要进行泛化的Edman降解测序。近来,应用自动化的Edman降解可产生短的N-末端序列标签,这是将质谱的序列标签概念用于Edman降解,业已成为一种强有力的蛋白质鉴定。 当对Edman的硬件进行简单改进,以迅速产生N-末端序列标签达10~20个/d,序列检签将适于在较小的蛋白质组中进行鉴定。若联合其他的蛋白质属性,如氨基酸组分分析、肽质质量、表现蛋白质分子量、等电点,可以更加可信地鉴定蛋白质。 选择BLAST程序,可与数据库相配比。 目前,采用一种Tagldent的检索程序,还可以进行种间比较鉴定,又提高了其在蛋白质组研究中的作用。3 与质谱(mass spectrometry)相关的技术质谱已成为连接蛋白质与基因的重要技术,开启了大规模自动化的蛋白质鉴定之门。 用来分析蛋白质或多肽的质谱有两个主要的部分,1)样品入机的离子源,2)测量被介入离子的分子量的装置。 首先是基质辅助激光解吸附电离飞行时间质谱(MALDI-TOF)为一脉冲式的离子化技术。 它从固相标本中产生离子,并在飞行管中测其分子量。 其次是电喷雾质谱(ESI-MS),是一连续离子化的方法,从液相中产生离子,联合四极质谱或在飞行时间检测器中测其分子量。 近年来,质谱的装置和技术有了长足的进展。在MALDI-TOF中,最重要的进步是离子反射器(ion reflectron)和延迟提取(delayed ion extraction),可达相当精确的分子量。 在ESI-MS中,纳米级电雾源(nano-electrospray source)的出现使得微升级的样品在30~40 min内分析成为可能。将反相液相色谱和串联质谱(tandem MS)联用,可在数十个picomole的水平检测;若利用毛细管色谱与串联质谱联用,则可在低picomole到高femtomole水平检测;当利用毛细管电泳与串联质谱连用时,可在小于femtomole的水平检测。 甚至可在attomole水平进行。 目前多为酶解、液相色谱分离、串联质谱及计算机算法的联合应用鉴定蛋白质。 下面以肽质指纹术和肽片段的测序来说明怎样通过质谱来鉴定蛋白质。(1)肽质指纹术(peptide mass fingerprint, PMF)由Henzel等人于1993年提出。 用酶(最常用的是胰酶)对由2-DE分离的蛋白在胶上或在膜上于精氨酸或赖氨酸的C-末端处进行断裂,断裂所产生的精确的分子量通过质谱来测量(MALDI-TOF-MS,或为ESI-MS),这一技术能够完成的肽质量可精确到0。1个分子量单位。 所有的肽质量最后与数据库中理论肽质量相配比(理论肽是由实验所用的酶来“断裂”蛋白所产生的)。 配比的结果是按照数据库中肽片段与未知蛋白共有的肽片段数目作一排行榜,“冠军”肽片段可能代表一个未知蛋白。若冠亚军之间的肽片段存在较大差异,且这个蛋白可与实验所示的肽片段覆盖良好,则说明正确鉴定的可能性较大。(2)肽片段(peptide fragment)的部分测序肽质指纹术对其自身而言,不能揭示所衍生的肽片段或蛋白质。 为进一步鉴定蛋白质,出现了一系列的质谱方法用来描述肽片段。 用酶或化学方法从N-或C-末端按顺序除去氨基酸,形成梯形肽片段(ladder peptide)。 首先以一种可控制的化学模式从N-末端降解,可产生大小不同的一系列的梯形肽片段,所得一定数目的肽质量由MALDI-TOF-MS测量。 另一种方法涉及羧基肽酶的应用,从C-末端除去不同数目的氨基酸形成肽片段。 化学法和酶法可产生相对较长的序列,其分子量精确至以区别赖氨酸(128。09)和谷氨酰胺(128。06)。 或者,在质谱仪内应用源后衰变(post-source decay, PSD)和碰撞诱导解离(collision-induced dissociation, CID),目的是产生包含有仅异于一个氨基酸残基质量的一系列肽峰的质谱。 因此,允许推断肽片段序列。 肽片段PSD的分析在MALDI反应器上能产生部分序列信息。 首先进行肽质指纹鉴定。 之后,一个有意义的肽片段在质谱仪被选作“母离子”,在飞行至离子反应器的过程中降解为“子离子”。 在反应器中,用逐渐降低的电压可测量至检测器的不同大小的片段。 但经常产生不完全的片段。 现在用肽片段来测序的方法始于70年代末的CID,可以一个三联四极质谱ESI-MS或MALDI-TOF-MS联合碰撞器内来完成。 在ESI-MS中,由电雾源产生的肽离子在质谱仪的第一个四极质谱中测量,有意义的肽片段被送至第二个四极质谱中,惰性气体轰击使其成为碎片,所得产物在第三个四极质谱中测量。 与MALDI-PSD相比,CID稳定、强健、普遍,肽离子片段基本沿着酰胺键的主架被轰击产生梯形序列。 连续的片段间差异决定此序列在那一点的氨基酸的质量。 由此,序列可被推测。 由CID图谱还可获得的几个序列的残基,叫做“肽序列标签”。 这样,联合肽片段母离子的分子量和肽片段距N- C端的距离将足以鉴定一个蛋白质。4 氨基酸组分分析1977年首次作为鉴定蛋白质的一种工具,是一种独特的“脚印”技术。 利用蛋白质异质性的氨基酸组分特征,成为一种独立于序列的属性,不同于肽质量或序列标签。 Latter首次表明氨基酸组分的数据能用于从2-DE凝胶上鉴定蛋白质。 通过放射标记的氨基酸来测定蛋白质的组分,或者将蛋白质印迹到PVDF膜上,在15
蛋白质是生物体中含量最高,功能最重要的生物大分子,存在于所有生物细胞,约占细胞干质量的50%以上, 作为生命的物质基础之一,蛋白质在催化生命体内各种反应进行、调节代谢、抵御外来物质入侵及控制遗传信息等方面都起着至关重要的作用,因此蛋白质也是生命科学中极为重要的研究对象。关于蛋白质的分析研究,一直是化学家及生物学家极为关注的问题,其研究的内容主要包括分子量测定,氨基酸鉴定,蛋白质序列分析及立体化学分析等。随着生命科学的发展,仪器分析手段的更新,尤其是质谱分析技术的不断成熟,使这一领域的研究发展迅速。 自约翰.芬恩(JohnB.Fenn)和田中耕一(Koichi.Tanaka)发明了对生物大分子进行确认和结构分析的方法及发明了对生物大分子的质谱分析法以来,随着生命科学及生物技术的迅速发展,生物质谱目前已成为有机质谱中最活跃、最富生命力的前沿研究领域之一[1]。它的发展强有力地推动了人类基因组计划及其后基因组计划的提前完成和有力实施。质谱法已成为研究生物大分子特别是蛋白质研究的主要支撑技术之一,在对蛋白质结构分析的研究中占据了重要地位[2]。 1.质谱分析的特点 质谱分析用于蛋白质等生物活性分子的研究具有如下优点:很高的灵敏度能为亚微克级试样提供信息,能最有效地与色谱联用,适用于复杂体系中痕量物质的鉴定或结构测定,同时具有准确性、易操作性、快速性及很好的普适性。 2.质谱分析的方法 近年来涌现出较成功地用于生物大分子质谱分析的软电离技术主要有下列几种:1)电喷雾电离质谱;2)基质辅助激光解吸电离质谱;3)快原子轰击质谱;4)离子喷雾电离质谱;5)大气压电离质谱。在这些软电离技术中,以前面三种近年来研究得最多,应用得也最广泛[3]。 3.蛋白质的质谱分析 蛋自质是一条或多条肽链以特殊方式组合的生物大分子,复杂结构主要包括以肽链为基础的肽链线型序列[称为一级结构]及由肽链卷曲折叠而形成三维[称为二级,三级或四级]结构。目前质谱主要测定蛋自质一级结构包括分子量、肽链氨基酸排序及多肽或二硫键数目和位置。 3.1蛋白质的质谱分析原理 以往质谱(MS)仅用于小分子挥发物质的分析,由于新的离子化技术的出现,如介质辅助的激光解析/离子化、电喷雾离子化,各种新的质谱技术开始用于生物大分子的分析。其原理是:通过电离源将蛋白质分子转化为[url=https://insevent.instrument.com.cn/t/Mp]气相[/url]离子,然后利用质谱分析仪的电场、磁场将具有特定质量与电荷比值(M/Z值)的蛋白质离子分离开来,经过离子检测器收集分离的离子,确定离子的M/Z值,分析鉴定未知蛋白质。 3.2蛋白质和肽的序列分析 现代研究结果发现越来越多的小肽同蛋白质一样具有生物功能,建立具有特殊、高效的生物功能肽的肽库是现在的研究热点之一。因此需要高效率、高灵敏度的肽和蛋白质序列测定方法支持这些研究的进行。现有的肽和蛋白质测序方法包括N末端序列测定的化学方法Edman法、C末端酶解方法、C末端化学降解法等,这些方法都存在一些缺陷。例如作为肽和蛋白质序列测定标准方法的N末端氨基酸苯异硫氰酸酯(phenylisothiocyanate)PITC分析法(即Edman法,又称PTH法),测序速度较慢(50个氨基酸残基/天);样品用量较大(nmol级或几十pmol级);对样品纯度要求很高;对于修饰氨基酸残基往往会错误识别,而对N末端保护的肽链则无法测序[4]。C末端化学降解测序法则由于无法找到PITC这样理想的化学探针,其发展仍面临着很大的困难。在这种背景下,质谱由于很高的灵敏度、准确性、易操作性、快速性及很好的普适性而倍受科学家的广泛注意。在质谱测序中,灵敏度及准确性随分子量增大有明显降低,所以肽的序列分析比蛋白容易许多,许多研究也都是以肽作为分析对象进行的。近年来随着电喷雾电离质谱(electrospray ionisation,ESI)及基质辅助激光解吸质谱(matrix assisted laser desorption/ionization,MALDI)等质谱软电离技术的发展与完善,极性肽分子的分析成为可能,检测限下降到fmol级别,可测定分子量范围则高达100000Da,目前基质辅助的激光解吸电离飞行时间质谱法(MALDI TOF MS)已成为测定生物大分子尤其是蛋白质、多肽分子量和一级结构的有效工具,也是当今生命科学领域中重大课题——蛋白质组研究所必不可缺的关键技术之一 [5] 。目前在欧洲分子生物实验室(EMBL)及美国、瑞士等国的一些高校已建立了MALDI TOF MS蛋白质一级结构(序列)谱库,能为解析FAST谱图提供极大的帮助,并为确证分析结果提供可靠的依据[6]。
传统的蛋白鉴定方法,如免疫印迹法、内肽的化学测序、已知或未知蛋白的comigration分析,或者在一个有机体中有意义的基因的过表达通常耗时、耗力,不适合高流通量的筛选。 目前,所选用的技术包括对于蛋白鉴定的图象分析、微量测序、进一步对肽片段进行鉴定的氨基酸组分分析和与质谱相关的技术。1 图象分析技术(Image analysis)“满天星”式的2-DE图谱分析不能依靠本能的直觉,每一个图象上斑点的上调、下调及出现、消失,都可能在生理和病理状态下产生,必须依靠计算机为基础的数据处理,进行定量分析。 在一系列高质量的2-DE凝胶产生(低背景染色,高度的重复性)的前提下,图象分析包括斑点检测、背景消减、斑点配比和数据库构建。 首先,采集图象通常所用的系统是电荷耦合CCD(charge coupled device)照相机;激光密度仪(laser densitometers)和Phospho或Fluoroimagers,对图象进行数字化。 并成为以象素(pixels)为基础的空间和网格。 其次,在图象灰度水平上过滤和变形,进行图象加工,以进行斑点检测。 利用Laplacian,Gaussian,DOG(difference of Gaussians) opreator使有意义的区域与背景分离,精确限定斑点的强度、面积、周长和方向。图象分析检测的斑点须与肉眼观测的斑点一致。 在这一原则下,多数系统以控制斑点的重心或最高峰来分析,边缘检测的软件可精确描述斑点外观,并进行边缘检测和邻近分析,以增加精确度。 通过阈值分析、边缘检测、销蚀和扩大斑点检测的基本工具还可恢复共迁移的斑点边界。 以PC机为基础的软件Phoretix-2D正挑战古老的Unix为基础的2-D分析软件包。 第三,一旦2-DE图象上的斑点被检测,许多图象需要分析比较、增加、消减或均值化。 由于在2-DE中出现100%的重复性是很困难的,由此凝胶间的蛋白质的配比对于图象分析系统是一个挑战。 IPG技术的出现已使斑点配比变得容易。 因此,较大程度的相似性可通过斑点配比向量算法在长度和平行度观测。 用来配比的著名软件系统包括Quest,Lips,Hermes,Gemini等,计算机方法如相似性、聚类分析、等级分类和主要因素分析已被采用,而神经网络、子波变换和实用分析在未来可被采用。 配比通常由一个人操作,其手工设定大约50个突出的斑点作为“路标”,进行交叉配比。 之后,扩展至整个胶。例如:精确的PI和MW(分子量)的估计通过参考图上20个或更多的已知蛋白所组成的标准曲线来计算未知蛋白的PI和MW。 在凝胶图象分析系统依据已知蛋白质的pI值产生PI网络,使得凝胶上其它蛋白的PI按此分配。 所估计的精确度大大依赖于所建网格的结构及标本的类型。 已知的未被修饰的大蛋白应该作为标志,变性的修饰的蛋白的PI估计约在±0。25个单位。 同理,已知蛋白的理论分子量可以从数据库中计算,利用产生的表观分子量的网格来估计蛋白的分子量。 未被修饰的小蛋白的错误率大约30%,而翻译后蛋白的出入更大。 故需联合其他的技术完成鉴定。2 微量测序(microsequencing)蛋白质的微量测序已成为蛋白质分析和鉴定的基石,可以提供足够的信息。 尽管氨基酸组分分析和肽质指纹谱(PMF)可鉴定由2-DE分离的蛋白,但最普通的N-末端Edman降解仍然是进行鉴定的主要技术。 目前已实现蛋白质微量测序的自动化。 首先使经凝胶分离的蛋白质直接印迹在PVDF膜或玻璃纤维膜上,染色、切割,然后直接置于测序仪中,可用于subpicomole水平的蛋白质的鉴定。 但有几点需注意:Edman降解很缓慢,序列以每40 min 1个氨基酸的速率产生;与质谱相比,Edman降解消耗大;试剂昂贵,每个氨基酸花费
传统的蛋白鉴定方法,如免疫印迹法、内肽的化学测序、已知或未知蛋白的comigration分析,或者在一个有机体中有意义的基因的过表达通常耗时、耗力,不适合高流通量的筛选。 目前,所选用的技术包括对于蛋白鉴定的图象分析、微量测序、进一步对肽片段进行鉴定的氨基酸组分分析和与质谱相关的技术。1 图象分析技术(Image analysis)“满天星”式的2-DE图谱分析不能依靠本能的直觉,每一个图象上斑点的上调、下调及出现、消失,都可能在生理和病理状态下产生,必须依靠计算机为基础的数据处理,进行定量分析。 在一系列高质量的2-DE凝胶产生(低背景染色,高度的重复性)的前提下,图象分析包括斑点检测、背景消减、斑点配比和数据库构建。 首先,采集图象通常所用的系统是电荷耦合CCD(charge coupled device)照相机;激光密度仪(laser densitometers)和Phospho或Fluoroimagers,对图象进行数字化。 并成为以象素(pixels)为基础的空间和网格。 其次,在图象灰度水平上过滤和变形,进行图象加工,以进行斑点检测。 利用Laplacian,Gaussian,DOG(difference of Gaussians) opreator使有意义的区域与背景分离,精确限定斑点的强度、面积、周长和方向。图象分析检测的斑点须与肉眼观测的斑点一致。 在这一原则下,多数系统以控制斑点的重心或最高峰来分析,边缘检测的软件可精确描述斑点外观,并进行边缘检测和邻近分析,以增加精确度。 通过阈值分析、边缘检测、销蚀和扩大斑点检测的基本工具还可恢复共迁移的斑点边界。 以PC机为基础的软件Phoretix-2D正挑战古老的Unix为基础的2-D分析软件包。 第三,一旦2-DE图象上的斑点被检测,许多图象需要分析比较、增加、消减或均值化。 由于在2-DE中出现100%的重复性是很困难的,由此凝胶间的蛋白质的配比对于图象分析系统是一个挑战。 IPG技术的出现已使斑点配比变得容易。 因此,较大程度的相似性可通过斑点配比向量算法在长度和平行度观测。 用来配比的著名软件系统包括Quest,Lips,Hermes,Gemini等,计算机方法如相似性、聚类分析、等级分类和主要因素分析已被采用,而神经网络、子波变换和实用分析在未来可被采用。 配比通常由一个人操作,其手工设定大约50个突出的斑点作为“路标”,进行交叉配比。 之后,扩展至整个胶。例如:精确的PI和MW(分子量)的估计通过参考图上20个或更多的已知蛋白所组成的标准曲线来计算未知蛋白的PI和MW。 在凝胶图象分析系统依据已知蛋白质的pI值产生PI网络,使得凝胶上其它蛋白的PI按此分配。 所估计的精确度大大依赖于所建网格的结构及标本的类型。 已知的未被修饰的大蛋白应该作为标志,变性的修饰的蛋白的PI估计约在±0。25个单位。 同理,已知蛋白的理论分子量可以从数据库中计算,利用产生的表观分子量的网格来估计蛋白的分子量。 未被修饰的小蛋白的错误率大约30%,而翻译后蛋白的出入更大。 故需联合其他的技术完成鉴定。
6 质谱仪的最新进展用质谱检测蛋白,首先考虑到用PMF与 MALDI-TOF联用,如果无法检测,下一步就用ESI-MS/MS创建序列标签。在PMF分析中,MALDI的平板中只需一小部分样本就足以检测,剩下的样本就可以用来创建序列标签。并且,在MALDI-TOF仪器上,用一种叫做“源后延迟”的方法可以对只有部分序列的肽段进行检测。然而,用这个方法产生的质谱图比较难说明,精确性也很差。最近,用MALDI联合四级杆-时间质谱分析器[30,31]以及原始的MALDI-TOF/TOF[32]方法产生了。因此同一份标本可以首先考虑用PMF检测蛋白[33],如果有必要的话,再用MS/MS创建序列标签。MALDI联合四级杆-TOF检测高通量蛋白是有希望的[31]。7 蛋白质组研究中的转录后修饰分析蛋白组分析很重要的一点就是能对蛋白表达水平以及转录后修饰,如磷酸化和糖基化进行研究。蛋白质的磷酸化是很有趣的,因为在信号转导途径中它扮演了重要的角色。最早检测蛋白质表达水平的方法是进行2-DE之前用35S-Met对样本进行代谢性标记,再在2-DE上进行放射自显影[34-36]。在凝胶中,不同蛋白的磷酸化和糖基化位点通常在凝胶中显示一连串蛋白质点,但是还需要做更详细的分析来确定修饰类型。蛋白质磷酸化的改变既可以用32P标记细胞,也可以用特异性磷酸化抗体做western blotting进行研究。如果用32P标记的方法,仍然需要做2-DE。经过凝胶比较后,把感兴趣的点从胶上切下来,然后用质谱鉴定[36]。Soskic使用的是印迹法,两个2-DE同时进行,一个用于做特异的磷酸化抗体实验,另一个做常规染色。蛋白质磷酸化和糖基化更为详细的特性可以用质谱来检测,但是需要更多的起始材料而不仅仅是二维凝胶上的一个点。另外这些分析不能产生直接的序列信息,技术上也比用质谱检测蛋白要难的多。在蛋白上查找磷酸化位点有很多种方法。为了检测消化后的混和肽中哪一个是磷酸化的,可以用MALDI-MS在磷酸化前后对混合肽做PMF分析:经过磷酸酯酶处理后,磷酸化的肽将会失去一个磷酸基团,分子量将比处理前小80Da.糖基化研究中,多聚糖从蛋白中释放出来,对多聚糖结构的检测就是从这些游离的多聚糖中得到的。一般把MALDI仪和外源性糖苷酶[37-40]联合使用进行检测。如果需要更为详细的信息,可以用ESI-MS联合使用前体离子扫描仪[41-44]。到目前为止,能够用电泳分离后的蛋白进行糖蛋白结构检测的报道很少。其中有一项研究是N端糖蛋白酶切以后用一维SDS-PAGE分离,然后用MALDI-MS以及外源性的糖苷酶进行结构分析[45]。8 用质谱研究蛋白-蛋白之间的作用经典的蛋白组学着重于研究蛋白质在何时何处表达。因为大部分的细胞功能都是由蛋白质复合体而不是由单个蛋白来执行的,所以鉴定蛋白质的成分和相互作用是非常重要的。这个过程可以用生物化学方法纯化蛋白质后用质谱来鉴定不同的成分,如人类剪接体的成分,酵母的核孔复合体[46]以及核蛋白体等都可以用此策略检测出来。一般的蛋白复合体是用亲和层析的方法纯化和分离,如免疫沉淀反应[47,48]。 DNA结合蛋白可以用同它们有特异性亲和力的核酸来分离,然后用质谱来鉴定[49]。Rigaut等人在串联层析的基础上,建立了一种通用的蛋白质复合体纯化方法[50]。在这个方法中,一种TAP标签和靶蛋白融合在一起,然后把蛋白转移到宿主细胞或者组织中,融合蛋白在宿主细胞和组织中能持续表达。TAP标签包括一种A蛋白和一种钙调蛋白,在标签之间有一个TEV蛋白酶切位点。用串联亲和层析法能将融合蛋白及与它相互作用的蛋白成分从细胞提取物中有效的分离,纯化出来。Rappsiber等人分别用亲和层析,交叉耦合以及质谱等方法[51]对酵母的核孔复合物Nup85p的亲和性进行研究。经过层析以后,用一维SDS-PAGE方法就可以分离蛋白复合体中的各个成分,因此二维电泳的不足之处就可以避免。同样也有可能直接用[url=https://insevent.instrument.com.cn/t/Yp][color=#3333ff]LC-MS[/color][/url]方法分析大分子量蛋白质复合体[52]。蛋白组分析的优越之处主要是用于蛋白和蛋白之间的相互作用以及蛋白的转录后修饰的研究,同时也可以用于基因表达水平的研究。质谱对于蛋白组分析来说是一项非常重要的技术,近年来仪器以及数据库软件的发展使得质谱成为能力更强大的工具。参考文献[1] O’Farrell PH. J Biol Chem,1975,250:4007–4021.[2] Karas M, Bachmann D, Bahr U, et al. Int J MassSpecrom Ion Process,1987,78:53–68.[3] Meng CK, Mann M, Fenn JB. et al. Atoms, Mol Clusters,1988,10:361–368.[4] Fenn JB, Mann M, Meng CK, et al.Science,1989,6(246):64–71.[5] Corthals GL, Wasinger VC, Hochstrasser DF, et al.Electrophoresis,2000,21:1104–1115.[6] Celis JE, Kruhoffer M, Gromova I, et al. FEBS Lett,2000,480:2–16.[7] Anderson L, Seilhamer J. Electrophoresis,1997,18:533–537.[8] Gygi SP, Rochon Y, Franza BR, et al. Mol Cell Biol,1999,19:1720–1730.[9] Henzel WJ, Stults JT, Wong SC, et al. ProcNatl Acad Sci USA,1993,90:5011–5015.[10] Mann M, Hojrup P, Roeppstorff P. Biol Mass Spectrom,1993,22:338–345.[11] Pappin DJ, Hojrup P, Bleasby AJ. Curr Biol,1993,3:327–332.[12] James P, Quadroni M, Carafoli E, et al. Biochem BiophysRes Commun,1993,195:58–64.[13] Yates JRD, Speicher S, Griffin PR, et al. AnalBiochem,1993,214:397–408.[14] Mann M, Wilm M. Anal Chem,1994,66:4390–4399.[15] Eng JK, McCormack AL, Yates JR. J Am Soc Mass Spectrom,1994,5:976–989.[16] Wilm M, Mann M. Int J Mass Spectrom Ion Process,1994,136:167–180.[17] Wilm M, Shevchenko A, Houthaeve T, et al. Nature 1996 379:466–469.[18] Wilm M, Mann M. Anal Chem,1996,68:1–8.[19] Morris HR, Paxton T, Panico M, et al. JProtein Chem,1997,16:469–479.[20] Rosenfeld J, Capedeville J, Guillemot JC, et al. AnalBiochem,1992,203:173–179.[21] Shevchenko A, Wilm M, Mann M. Anal Chem,1996,68:850–858.[22] Pandey A, Andersen JS, Mann M. Science’s STKE: www.stke.org/cgi/content/full/OC–sigtrans 2000/37/pl1.[23] Kussmann M, Nordhoff E, Rahbek-Nielsen H, et al. J Mass Spectrom,1997,32:593–601.[24] Gobom J, Nordhoff E, Mirgorodskaya E, et al. J Mass Spectrom,1999,34:105–116.[25] Clauser KR, Baker P, Burlingame AL. Anal Chem,1999,71:2871–2882.[26] Binz PA, Muller M, Walther D, et al. Anal Chem,1999,71:4981–4988.[27] Bienvenut WV, Sanchez JC, Karmime A, et al. Anal Chem,1999,71:4800–4807.[28] Neubauer G, King A, Rappsilber J, et al. Nat Genet,1998,20:46–50.[29] Yates III JR, Carmack E, Hays L, et al. MethodsMol Biol,1999,112:553–569.[30] Loboda AV, Krutchinsky AN, Bromirski M, et al. Rapid Commun Mass Spectrom,2000,14:1047–1057.[31] Shevchenko A, Loboda A, Schevchenko A, et al. Anal Chem,2000,72:2132–2141.[32] Medzihradszky KF, Campbell JM, Baldwin MA, et al. Anal Chem 2000,72:552–558.[33] Krutchinsky AN, Zhang W, Chait BT. J Am Soc Mass Spectrom,2000,11:493–504.[34] Nyman TA, Matikainen S, Sareneva T, et al. Eur J Biochem 2000 267:4011–4019.[35] Celis JE, editor. Cell Biology: A Laboratory Handbook, vol. 4,2nd. Academic Press, 1998,375–385.[36] Gerner C, Frohwein U, Gotzmann J, et al. J Biol Chem,2000,Sep 7 [epubahead of print].[37] Colangelo J, Orlando R. Anal Chem,1999,71:1479–1482.[38] Geyer H, Schmitt S, Wuhrer M, et al. Anal Chem1999 71:476–482.[39] Harvey DJ. Mass Spectrom Rev,1999,18:349–450.[40] Nyman TA, Kalkkinen N, et al. Eur J Biochem,1998,253:485–493.[41] Sheeley DM, Reinhold VN. Anal Chem,1998,70:3053–3059.[42] Reinhold VN, Reinhold BB, Costello CE. Anal Chem,1995,67:1772–1784.[43] Kuster B, Hunter AP, Wheeler SF, et al. Electrophoresis,1998,19:1950–1959.[44] Rout MP, Aitchison JD, Suprapto A, et al. J Cell Biol,2000,148:635–651.[45] Yamaguchi K, Subramanian AR. J Biol Chem ,2000,275:28 466–28 482.[46] Yamaguchi K, von Knoblauch K, Subramanian AR. J BiolChem,2000,275:28 455–28 465.[47] Rotheneder H, Geymayer S, Haidweger E. J Mol Biol,1999,293:1005–1015.[48] Boehning D, Joseph SK. EMBO J,2000,19:5450–5459.[49] Nordhoff E, Krogsdam AM, Jorgensen HF, et al. Nat Biotechnol,1999,17:884–888.[50] Rigaut G, Shevchenko A, Rutz B, et al. Nat Biotechnol,1999,17:1030–1032.[51] Rappsilber J, Siniossoglou S, Hurt EC, et al. Anal Chem2000 72:267–275.[52] Link AJ, Eng J, Schieltz DM, et al. Nat Biotechnol,1999,17:676–682.T.A. Nyman /Biomolecular Engineering, 2001:18 .221–227.
生物质谱技术帮助发现精神分裂症特征蛋白 (2006-12-28 9:34:47,新闻来源:人民网) 日前,四川泸州医学院附属医院硕士研究生姜伟,在导师王开正教授指导下,利用表面增强激光解析电离飞行时间质谱技术,发现了一组对精神分裂症的早期预警、鉴别诊断、治疗观察具有重要意义的蛋白标志物。 目前鉴别诊断精神分裂症缺乏有效的病理学、影像学和实验诊断依据,也缺乏客观的检查指标用于早期诊断,这也是造成对某些精神分裂症的评定产生分歧、鉴定困难的原因之一。 姜伟等人应用获得诺贝尔化学奖、高灵敏度高解析度的表面增强激光解析电离飞行时间质谱技术,检测精神分裂症血清蛋白质指纹图谱,将40名精神分裂症患者血清与44名其他人对照血清随机分为训练集和验证集,将筛选训练集精神分裂症差异蛋白建立人工神经网络诊断模型,再将验证集验证该模型的诊断效率。结果发现精神分裂症患者与对照组血清蛋白质指纹图谱有15个差异表达的蛋白质荷比峰,筛选出其中6个有明显表达差异的标志蛋白,建立的人工神经网络诊断模型对精神分裂症的诊断灵敏度和特异性分别为95.0%和95.8%,阴性预测值和阳性预测值分别为95.8%和95.0%。姜伟等人依据这一结果认为,精神分裂症患者血清具有高表达的特征蛋白,建立的人工神经网络模型为精神分裂症的实验诊断提供了一种蛋白组学的新方法。这种方法检查过程快速、简便,不会破坏所测定的蛋白质,结果可靠,可重复检测。 ————2006.12.27健康报也有报道--------------------------------------------------------------------另:两毫升血5小时鉴别精神分裂症 作者:周丽 文章来源:泸州晚报 点击数:0 更新时间:2006-12-20 只需要从体内抽取两毫升鲜血,经过5个小时的实验室检测,就能诊断是否患有精神分裂症。昨(19)日记者获悉,如此简便的方法已经在我市投入使用,这也是西南地区首次将诺贝尔化学奖应用到精神分裂症的实验室诊断中。 据悉,该技术在泸医附院检验科正式投入使用。泸医附院检验科主任王元正告诉记者,泸医附院引进的表面增强激光解析电离飞行时间质谱技术是获得2002年诺贝尔化学奖的高新技术,通过对患者的抽血检测,发现了一组对精神分裂症的早期预警、鉴别诊断、治疗观察具有重要意义的蛋白标志物。 而泸医附院引进的这种技术,将精神分裂症患者血清蛋白指纹图与健康人做差异蛋白组学分析,利用计算机程序找到了精神分裂症患者特征表达的蛋白质,将6个蛋白的相对含量建立人工神经网络诊断模型并进行盲法验证。结果显示,对精神分裂症的诊断灵敏度为95.0%,特异性为95.8%。整个实验检测过程只需要5个小时。 目前,全国对此技术的利用并不广泛,泸医附院在西南地区率先引进使用。这种技术还用于肿瘤、心血管疾病、风湿免疫性疾病、感染性疾病等疑难疾病的鉴别诊断,特别是对各种肿瘤的早期诊断灵敏度和特异性都在95%以上。
蛋白分析系统在我们选择蛋白分析工具的时候,通常是根据不同的蛋白来选择不同的分析手段,如凝胶电泳、化学荧光染色、质谱等等。但是目前已经研制出的蛋白分析工具的种类繁多,从这一方面也在一定程度上反映了蛋白分析的复杂性。以下是一些近期推出的蛋白分析系统,希望能帮助您轻松完成研究工作。
分子量测定: Q:为什么测定的结果跟理论的结果不一致? A:因为机器测定会有一定的误差,如100000Da的蛋白质最大误差是100Da,如果测定的结果与理论值成倍数关系的可能原因是:0.3倍可能是三电荷峰,0.5倍可能是双电荷峰,原因是样品含碱氨基酸较多;2倍可能是二聚体,3倍可能是三聚体,原因可能是样品中含有过多的二硫键。 Q:为什么我的样品非常纯,但在主峰的附近会有一些小峰?同时在主峰一半或两倍的地方有一个小峰? A:这是因为在激光的扫描的过程中可能会使样品分子损失一些离子,如H2O、NH3等,或者出现样品分子与基质的加合峰,正常的情况下MALDI-TOF单电荷峰占主要,所以有时会在主峰一半或两倍的地方有一个小峰,是双电荷峰和二聚体峰; Q:为什么我的样品纯度、浓度都很好,盐浓度也很低,却得不到结果? A:这只能与蛋白质本身的结构有关,基质不能很好的分离样品,所以希望您能尽可能滇供样品其他的理化质,如酸、碱蛋白、糖蛋白等。的确,有时您的样品的所有质、状态都正常,还是不能得到您想要的结果,这种问题其实已经超出了我们能力的范围,我们仅仅是您整个实验的一个环节,能够做到的是向您提供一个准确的结果。 肽指纹图谱及数据库检索 Q:为什么考马斯染色的结果比银染的结果好。 A:主要是银染的灵敏度较高,得到的蛋白质量较少,再加上在处理样品的过程中银染要脱银,增加洗脱的次数,样品的损失较大,所以噪音和角蛋白污染对目的蛋白的影响较大,对最终的数据库检索也会产生影响 Q:我的PMF的峰值较少,为什么?这些片段数量及峰值是否达到最基本的数据库检索的要求?影响检索的结果吗? A:因为我们用的是胰酶对蛋白质进行降解的,胰酶是一种专一较强的酶,只水解精氨酸、赖氨酸的C端,可能是你的样品的酶解的位点偏多或偏少,偏多酶解为小片段,峰值跟基质在一起难以分辨,偏少肽段的分子量偏大,胶内的样品滞留在胶内,在质谱图上体现不出来,溶液样品可以收集到,但是分子量越大检索的结果越不准确;理论上图谱的峰越多越好,要是都能匹配上那么蛋白的确定程度就更高,相反就越低;一般至少要拿到4个肽段。 Q:如何理解这些片段峰值大小意义,越高(纵坐标值大)越精确吗?以及这些酶解片段的数量及峰值一般应达到什么标准才算比较好?? A:片段峰横坐标的大小的意义是酶解下来的肽段的质量大小,纵坐标表示的收集的该肽段的相对的信号的强度,左边是相对的,右边是绝对的,绝对的越高,抗干扰的能力越好,质谱的精确度由仪器来决定的。我们的仪器的精确度在10ppm以内。 Q:图谱上肽段多于8条,为什么选这8条? A:至于提交检索的肽段的数量是根据肽段的信号强度来选取的,同时在软件处理的过程中会过滤一部分峰, 如胰酶的自解峰等; Q:数据库检索报告中m/zsubmitted,MH+matched两项是什么意思? A:m/z submitted 的意思是您提交给数据库的m/z值(因为质谱仪用m/z来分离样品的), MH+matched指的是与你提交数值相匹配数值,因为在样品在离子化过程中从基质中得到一个质子,所以它会加一个H Q:这两项的分子量和你们的图谱上相应分子量不一致。 A:因为在用图谱数据在数据库检索之前需要通过一个数据的校正和处理,所以图谱上的值与提交检索的值有区别图谱中的峰值是经过一个软件处理后的结果,主要是经同位素峰的合并图谱上的结果会不一至的,例如在你的图上读到的是1519.402,在看图的时候把它给放大处理(只有我这里的图谱处理的软件才能看到),就会发现存在1518.402、1520.402、1521.402、1522.402等相差1的质谱峰,这是由于在天然界中好多物质都存在同位素如O16、O17、O18等,所以在提交给数据库之前会用一个类似于碳水化合物的通式进行校正,所以图谱上读到的数可能会与提交的相差1,还在处理的过程的设置也会影响给出的峰值,但应该在0.1Da以内的;同时在还会进行胰酶自解峰的扣除和空白对照峰的扣除。
自80年代以来一系列新的软电离技术如快原子轰击电离 、基质辅助激光解吸电离 、电喷雾电离等发现后,生物质谱技术迅速发展,已成为现代科学研究前沿的热点之一。而其中又以基质辅助激光解吸质谱(MALD I2MS)和电喷雾电离质谱(ESI2MS)应用最为广泛。基质辅助激光解吸质谱灵敏度高、可操作性强且对生物样品中的无机盐和缓冲溶液具有较好包容性;电喷雾电离质谱选择性好、分析质量范围宽、样品消耗量小、易于与各种色谱联用。在生物样品的处理中常常需要用到非挥发性的盐,用于为细胞营造无毒的环境,稳定溶剂化的样品及维持酶的活性等。此外,许多用于分离生物分子的分离方法也需要高浓度的盐和缓冲溶液 。但是,样品处理及分离过程中所用的NaCl、十二烷基磺酸钠、盐酸胍、尿素、甘油、二甲基亚砜等都会影响后续质谱高灵敏的分析 。因为这些不挥发的低分子量污染物会导致复杂加合物的形成,增加噪音及造成明显的信号抑制 。此外,在复杂的组织或细胞蛋白质组中,与疾病和信号传导相关的蛋白质往往是属于低丰度的蛋白质,这些重要的蛋白质由于本身存在的量极少而很难得以有效鉴定 。因此,对蛋白质/多肽样品的预富集处理将是MALD I2MS或ESI2MS得到高质量质谱图的前提,也是成功鉴定蛋白质的关键。该文献主要侧重于相关工作的概述。
质谱技术在蛋白鉴定等实验中具有不可忽视的作用,应用前景十分广泛。那么,有问题了怎么解决呢?问题1. 一级质谱和二级质谱有什么区别?什么时候做一级,什么时候做二级?答:一级质谱鉴定的方式主要指胎指纹图谱(peptide-mass mapping, PMF),即利用质谱仪精确测量酶解片段的分子量并搜库比较实现蛋白质的鉴定,二级质谱是在一级质谱的基础上再选择部分肽段做进一步的破碎并对碎片进行深入分析和比较,鉴定出该肽段的序列并结合PMF的结果从而实现蛋白质的鉴定。二级质谱能够得到部分肽短的序列,具有更高的可靠性。随着现在杂志对数据的要求越来越严格,二级质谱鉴定是蛋白鉴定的大趋势,而且即使目前做一级质谱鉴定的结果,也需要挑选部分PMF结果做二级质谱验证。问题2. 研究的物种不是模式生物怎么办?答:可以参考亲缘关系最近的模式生物做比对而实现蛋白质的成功鉴定,如果质谱图很好而没有鉴定结果说明这是一个全新的蛋白,可以采用de-novo等技术做深入分析与鉴定。问题3. 该如何评价质谱效果的好坏?答:做PMF一般得分超过60分(P0.05)就算成功鉴定,而串联质谱,得分超过60分或者虽然没有超过60分,但是有最少一条肽段的得分超过30分就算成功鉴定。问题4. 一些特殊的质谱方法有什么用途?答:质谱的其它用途包括修饰位点分析、蛋白测序、混合蛋白鉴定以及分子量精确测定、二硫键位置分析等等。问题5. 用什么染色方法比较好?答:最好用考马斯亮蓝法进行染色,银染也可以,但鉴定成功率稍低,并且推荐用串联质谱对银染蛋白进行鉴定可以大大提高鉴定成功率。问题6 质谱鉴定取胶点有什么注意事项?答:离心管最好用进口离心管,以免塑料污染;水最好用去离子水或者双蒸水;取点的时候带好口罩与手套,以免角蛋白污染。问题7. 凝胶是否可以长期保存?该如何保存?如何运送?答:蛋白凝胶可以长期保存而不影响质谱鉴定效果,一般而言,如果在一两个星期以内,最好用保鲜膜包好,放到4度冰箱保存,如果保存时间超过一个月,可以把蛋白点取下后放入-20度或者-80度冰箱,不会影响后续质谱鉴定效果。运送胶点的时候采用常温运送即可,三五天内都不会有太大的影响。问题8. 质谱鉴定大致是什么流程?质谱鉴定主要包括蛋白酶解、质谱数据获取、对库检索三个步骤。问题9. 串联质谱鉴定和质谱测序有什么区别?质谱鉴定往往采用软件对已有数据库进行自动检索匹配得到结果,而质谱测序则需要根据质谱峰图中相邻或者相近峰的分子量差异直接推算出肽段的序列。问题10. 未知蛋白质是通过质谱还是蛋白质测序仪来测序的呢?现在比较简单的方法是质谱法。如果质谱法不能确定的话还可以结合N端测序,可测10几个氨基酸。两个数据结合分析基本可以确定目的蛋白了,现在的蛋白质组数据库相当强大了。不知道看完这10个FAQ,各位有没有些许受益,如果还有好的想法或者意见,欢迎给小编留言!(来源:互联网)
如果目前分离蛋白质组的最好技术是2-DE,那么随之而来的挑战是数百数千个蛋白如何被鉴定。在这里,我们不考虑传统的蛋白鉴定方法,如免疫印迹法、内肽的化学测序、已知或未知蛋白的comigration分析,或者在一个有机体中有意义的基因的过表达。并不是因为这些方法无效,而是因为它们通常耗时、耗力,不适合高流通量的筛选。目前,所选用的技术包括对于蛋白鉴定的图象分析、微量测序;进一步对肽片段进行鉴定的氨基酸组分分析和与质谱相关的技术。(1) 图象分析技术(Image analysis)。“满天星”式的2-DE图谱分析不能依靠本能的直觉,每一个图象上斑点的上调、下调及出现、消失,都可能在生理和病理状态下产生,必须依靠计算机为基础的数据处理,进行定量分析。在一系列高质量的2-DE凝胶产生(低背景染色,高度的重复性)的前提下,图象分析包括斑点检测、背景消减、斑点配比和数据库构建。首先,采集图象通常所用的系统是电荷耦合CCD(charge coupled device)照相机;激光密度仪(laser densitometers)和Phospho或Fluoro?imagers,对图象进行数字化。并成为以象素(pixels)为基础的空间和网格。其次,在图象灰度水平上过滤和变形,进行图象加工,以进行斑点检测。利用Laplacian,Gaussian,DOG(difference of Gaussians) opreator使有意义的区域与背景分离,精确限定斑点的强度、面积、周长和方向。图象分析检测的斑点须与肉眼观测的斑点一致。在这一原则下,多数系统以控制斑点的重心或最高峰来分析,边缘检测的软件可精确描述斑点外观,并进行边缘检测和邻近分析,以增加精确度。通过阈值分析、边缘检测、销蚀和扩大斑点检测的基本工具还可恢复共迁移的斑点边界。以PC机为基础的软件Phoretix-2D正挑战古老的Unix为基础的2-D分析软件包。第三,一旦2-DE图象上的斑点被检测,许多图象需要分析比较、增加、消减或均值化。由于在2-DE中出现100%的重复性是很困难的,由此凝胶间的蛋白质的配比对于图象分析系统是一个挑战。IPG技术的出现已使斑点配比变得容易。因此,较大程度的相似性可通过斑点配比向量算法在长度和平行度观测。用来配比的著名软件系统包括Quest,Lips,Hermes,Gemini等,计算机方法如相似性、聚类分析、等级分类和主要因素分析已被采用,而神经网络、子波变换和实用分析在未来可被采用。配比通常由一个人操作,其手工设定大约50个突出的斑点作为“路标”,进行交叉配比。之后,扩展至整个胶。例如:精确的PI和MW(分子量)的估计通过参考图上20个或更多的已知蛋白所组成的标准曲线来计算未知蛋白的PI和MW。在凝胶图象分析系统依据已知蛋白质的pI值产生PI网络,使得凝胶上其它蛋白的PI按此分配。所估计的精确度大大依赖于所建网格的结构及标本的类型。已知的未被修饰的大蛋白应该作为标志,变性的修饰的蛋白的PI估计约在±0.25个单位。同理,已知蛋白的理论分子量可以从数据库中计算,利用产生的表观分子量的网格来估计蛋白的分子量。未被修饰的小蛋白的错误率大约30%,而翻译后蛋白的出入更大。故需联合其他的技术完成鉴定?(2) 微量测序(microsequencing)。蛋白质的微量测序已成为蛋白质分析和鉴定的基石,可以提供足够的信息。尽管氨基酸组分分析和肽质指纹谱(PMF)可鉴定由2-DE分离的蛋白,但最普通的N-末端Edman降解仍然是进行鉴定的主要技术。目前已实现蛋白质微量测序的自动化。首先使经凝胶分离的蛋白质直接印迹在PVDF膜或玻璃纤维膜上,染色、切割,然后直接置于测序仪中,可用于subpicomole水平的蛋白质的鉴定。但有几点需注意:Edman降解很缓慢,序列以每40 min 1个氨基酸的速率产生;与质谱相比,Edman降解消耗大;试剂昂贵,每个氨基酸花费3~4$。这都说明泛化的Edman降解蛋白质不适合分析成百上千的蛋白质。然而,如果在一个凝胶上仅有几个有意义的蛋白质,或者如果其他技术无法测定而克隆其基因是必需的,则需要进行泛化的Edman降解测序。近来,应用自动化的Edman降解可产生短的N-末端序列标签,这是将质谱的序列标签概念用于Edman降解,业已成为一种强有力的蛋白质鉴定。当对Edman的硬件进行简单改进,以迅速产生N-末端序列标签达10~20个/d,序列检签将适于在较小的蛋白质组中进行鉴定.若联合其他的蛋白质属性,如氨基酸组分分析、肽质质量、表现蛋白质分子量、等电点,可以更加可信地鉴定蛋白质。选择BLAST程序,可与数据库相配比。目前,采用一种Tagldent的检索程序,还可以进行种间比较鉴定,又提高了其在蛋白质组研究中的作用。(3) 与质谱(mass spectrometry)相关的技术。质谱已成为连接蛋白质与基因的重要技术,开启了大规模自动化的蛋白质鉴定之门。用来分析蛋白质或多肽的质谱有两个主要的部分,1)样品入机的离子源,2)测量被介入离子的分子量的装置。首先是基质辅助激光解吸附电离飞行时间质谱(MALDI-TOF)为一脉冲式的离子化技术。它从固相标本中产生离子,并在飞行管中测其分子量。其次是电喷雾质谱(ESI-MS),是一连续离子化的方法,从液相中产生离子,联合四极质谱或在飞行时间检测器中测其分子量。近年来,质谱的装置和技术有了长足的进展。在MALDI-TOF中,最重要的进步是离子反射器(ion reflectron)和延迟提取(delayed ion extraction),可达相当精确的分子量。在ESI-MS中,纳米级电雾源(nano-electrospray source)的出现使得微升级的样品在30~40 min内分析成为可能。将反相液相色谱和串联质谱(tandem MS)联用,可在数十个picomole的水平检测;若利用毛细管色谱与串联质谱联用,则可在低picomole到高femtomole水平检测;当利用毛细管电泳与串联质谱连用时,可在小于femtomole的水平检测。甚至可在attomole水平进行。目前多为酶解、液相色谱分离、串联质谱及计算机算法的联合应用鉴定蛋白质。下面以肽质指纹术和肽片段的测序来说明怎样通过质谱来鉴定蛋白质。1)肽质指纹术(peptide mass fingerprint, PMF)是由Henzel等人于1993年提出。用酶(最常用的是胰酶)对由2-DE分离的蛋白在胶上或在膜上于精氨酸或赖氨酸的C-末端处进行断裂,断裂所产生的精确的分子量通过质谱来测量(MALDI-TOF-MS,或为ESI-MS),这一技术能够完成的肽质量可精确到0.1个分子量单位。所有的肽质量最后与数据库中理论肽质量相配比(理论肽是由实验所用的酶来“断裂”蛋白所产生的)。配比的结果是按照数据库中肽片段与未知蛋白共有的肽片段数目作一排行榜,“冠军”肽片段可能代表一个未知蛋白.若冠亚军之间的肽片段存在较大差异,且这个蛋白可与实验所示的肽片段覆盖良好,则说明正确鉴定的可能性较大。2)肽片段(peptide fragment)的部分测序。肽质指纹术对其自身而言,不能揭示所衍生的肽片段或蛋白质。为进一步鉴定蛋白质,出现了一系列的质谱方法用来描述肽片段。用酶或化学方法从N-或C-末端按顺序除去氨基酸,形成梯形肽片段(ladder peptide)。首先以一种可控制的化学模式从N-末端降解,可产生大小不同的一系列的梯形肽片段,所得一定数目的肽质量由MALDI-TOF-MS测量。另一种方法涉及羧基肽酶的应用,从C-末端除去不同数目的氨基酸形成肽片段。化学法和酶法可产生相对较长的序列,其分子量精确至以区别赖氨酸(128.09)和谷氨酰胺(128.06)。或者,在质谱仪内应用源后衰变(post-source decay, PSD)和碰撞诱导解离(collision-induced dissociation, CID),目的是产生包含有仅异于一个氨基酸残基质量的一系列肽峰的质谱。因此,允许推断肽片段序列。肽片段PSD的分析在MALDI反应器上能产生部分序列信息。首先进行肽质指纹鉴定。之后,一个有意义的肽片段在质谱仪被选作“母离子”,在飞行至离子反应器的过程中降解为“子离子”。在反应器中,用逐渐降低的电压可测量至检测器的不同大小的片段。但经常产生不完全的片段。现在用肽片段来测序的方法始于70年代末的CID,可以一个三联四极质谱ESI-MS或MALDI-TOF-MS联合碰撞器内来完成。在ESI-MS中,由电雾源产生的肽离子在质谱仪的第一个四极质谱中测量,有意义的肽片段被送至第二个四极质谱中,惰性气体轰击使其成为碎片,所得产物在第三个四极质谱中测量。与MALDI-PSD相比,CID稳定、强健、普遍,肽离子片段基本沿着酰胺键的主架被轰击产生梯形序列。连续的片段间差异决定此序列在那一点的氨基酸的质量。由此,序列可被推测。由CID图谱还可获得的几个序列的残基,叫做“肽序列标签”。这样,联合肽片段母离子的分子量和肽片段距N-、C?端的距离将足以鉴定一个蛋白质。(4) 氨基酸组分分析。1977年首次作为鉴定蛋白质的一种工具,是一种独特的“脚印”技术。利用蛋白质异质性的氨基酸组分特征,成为一种独立于序列的属性,不同于肽质量或序列标签。Latter首次表明氨基酸组分的数据能用于从2-DE凝胶上鉴定蛋白质。通过放射标记的氨基酸来测定蛋白质的组分,或者将蛋白质印迹到PVDF膜上,在155℃进行酸性水解1 h,通过这
我是新手,还没入门,想请教下大家有没有过质谱测蛋白的?大家都是用哪种质谱来测得蛋白,有哪些网站或者书籍之类的推荐学习吗?
听说蛋白酶解肽段二级质谱图可以通过de novo方法测序,请问,除了手工解析,用哪些软件可以快速鉴定辅助解析
1994年,Marc Wilkins在Siena双向凝胶电泳(two-dimensional electrophoresis,2-DE)会议上最早提出了蛋白质组(proteome)概念,并于1995年7月的Electrophoresis杂志上发表。随着高通量、高灵敏度、高分辨率生物质谱技术的出现,蛋白质组学技术取得飞速发展,人们不再满足于对一个细胞或组织的蛋白质进行定性研究,而是着眼于蛋白质量的研究,于是蛋白质组学概念就被提出,并得到了广泛的应用。蛋白质组学(Proteomics)是蛋白质(protein)与 基因组学(genomics)两个词的组合体,表示“一种基因组所表达的全套蛋白质”,即包括一种细胞乃至一种生物所表达的全部蛋白质。蛋白质组学研究,就是要把一个基因组表达的绝大多数蛋白质或一个复杂的混合体系中绝大多数蛋白质进行精确的定量和鉴定。蛋白质组本质上指的是在大规模水平上研究蛋白质的特征,包括蛋白质的表达水平,翻译后的修饰,蛋白与蛋白相互作用等,由此获得蛋白质水平上的关于疾病发生,细胞代谢等过程的整体而全面的认识。蛋白质组学是一门以全面的蛋白质性质研究为基础,在蛋白质水平对疾病机理、细胞模式、功能联系等方面进行探索的科学。目前最新的iTRAQ蛋白定量分析技术在此基础上被提出,并被得到广泛应用。仅仅知道蛋白质的身份并不足以对蛋白质给出最终定论,因为蛋白质的浓度对于实现其在细胞中的功能来说极其重要,一种特殊蛋白质在浓度上的变化,就能预示细胞的突变过程。因此,科学家能够对蛋白质的相对和绝对浓度进行测量,是很重要的事情。过去,科学家通常先进行二维(2D)凝较电泳,切断条带,再用质谱方法测量条带中的蛋白质。可是,这种方法不是很理想:既不是非常敏感,也不是非常精确。新泽西医学及牙科大学的蛋白组学研究中心主任Hong Li说:“当我们开始蛋白组学研究时,就采用2D凝胶技术,但得出的信息量却让大伙很失望,因为许多蛋白质已经改变了自身的代谢过程,如热休克蛋白或者是管家蛋白。”蛋白组学中的方法一直在不断提高。基于高度敏感性和精确性的串联质谱方法,不需要凝胶,就可以获得相对和绝对定量的蛋白质结果。iTRAQ和iCAT是这些新进展中的两大主力,但是,新技术也有不尽如人意的地方,需要不断改进。iTRAQ技术是由美国应用生物系统公司(Applied Biosystems Incorporation,ABI)2004年开发的同位素标记相对和绝对定量(isobaric tags for relative and absolute quantitation,iTRAQ)技术,是一种新的、功能强大的可同时对四种样品进行绝对和相对定量研究的方法,这种方法是建立在iTRAQ试剂的基础上。上海舜百生物公司目前所采用的就是这种iTRAQ技术。该技术的主要特点在于:1. 分离能力强、分析范围广;2. 定性分析结果可靠,可以同时给出每一个组分的分子量和丰富的结构信息;3. MS具备高灵敏度、检测限低;4. 分析时间快,分离效果好;5. 自动化程度高;6. iTRAQ技术不仅可发现胞浆蛋白,还有膜蛋白、核蛋白、胞外蛋白。iTRAQ技术可检测出低丰度蛋白、强碱性蛋白、小于10KD或大于200KD的蛋白。上海舜百生物使用的液相色谱仪的型号是日本岛津公司2D-nano-HPLC,质谱仪型号是美国ABI公司的MALDI-TOF-TOF 4700,标记试剂盒是美国ABI公司的ITRAQ标记试剂盒。舜田生物所采用的iTRAQ试剂是一种小分子同重元素化学物质,包括三部分:一端是报告部分(reporter group ),另一端是肽反应部分(peptide reactive group),中间部分是平衡部分(balance group)。 其中,reporter group:质量为114Da、115 Da、116 Da、117 Da,因此iTRAQ试剂共四种。 peptide reactive group:将reporter group与肽N端及赖氨酸侧链连接,几乎可以标记样本中所有蛋白质。 balance group:质量为31 Da、30 Da、29 Da、28 Da,使得四种iTRAQ试剂分子量均为145 Da,保证iTRAQ标记的同一肽段m/z相同。舜百生物公司iTRAQ的操作程序如下:将蛋白质裂解为肽段,然后用iTRAQ试剂进行差异标记。再将标记的样本相混合,这样就可以对其进行比较。与样本结合后,通常用MudPIT多维蛋白质鉴定技术进行下一步的操作,用2D液相色谱串联质谱进行分析。在质谱分析鉴定特殊肽离子片断结构的基础上,采用美国应用生物系统公司的软件包MASCOT和Protein Pilot对每一个肽段进行鉴定。其具体操作如下图所示: http://img.dxycdn.com/trademd/upload/userfiles/image/2013/11/A1383890263png_small.jpgiTRAQ技术对检测标本也有一定要求。舜百生物要求检测蛋白量最低不少于50 ug,浓度最低不少于5 ug/uL,否则同位素无法标记。而对蛋白提取试剂也要求使用普通的组织、细胞裂解液即可,切忌不要使用二维电泳试剂提取。iTRAQ技术区别与以往二维电泳技术具有更明显的优势,两者比较如下:1. 二维电泳所检测的发生表达变化的蛋白都位于细胞浆内,而iTRAQ可检测到蛋白有胞浆蛋白外,还有线粒体蛋白、膜蛋白和核蛋白。2. 二维电泳观察到的蛋白变化在2倍以上,而用iTRAQ计算出的蛋白变化在1.3-1.6倍之间。3. iTRAQ技术在鉴定大分子和小分子蛋白方面也有优势。4. 二维电泳是通过切断条带,再用质谱方法测量条带中的蛋白质,但该方法既不是非常敏感,也不是非常精确,获取的信息量很少。 而itraq技术是基于高度敏感性和精确性的串联质谱方法,不需要凝胶,就可以获得相对和绝对定量的蛋白质结果。5. iTRAQ可以对任何类型的蛋白质进行鉴定,包括高分子量蛋白质、酸性蛋白质和碱性蛋白质,而二维电泳对这些蛋白质都束手无策。由此,舜百生物得出结论:iTRAQ技术比二维电泳技术能发现更多数量和种类的蛋白,但在比较胞浆蛋白及蛋白量的变化方面,二维电泳技术有一定长处,对iTRAQ的实验结果有互补作用。
把蛋白完全水解成氨基酸,可以在pmol到nmol水平上分离和定量。虽然氨基酸的序列信息已经无法得到,但是组成蛋白的氨基酸种类及含量信息可以得到,利用氨基酸的指纹信息可以鉴定蛋白。
[font='times new roman'][size=14px]药物靶蛋白鉴定方法[/size][/font][font='times new roman'][size=14px]非蛋白质组学鉴定方法[/size][/font][font='times new roman'][size=14px]非蛋白质组学的传统药物靶蛋白鉴定方法,如免疫印迹法、内肽的化学测序、已知或未知蛋白的迁徙分析等方法,通常耗时、耗力且不适合进行高流通量的筛选。目前,所使用的技术包括:第一,蛋白鉴定的图象分析,利用产生的表观分子量的网格来估计蛋白的分子量,未被修饰的小蛋白错误率大约[/size][/font][font='times new roman'][size=14px] [/size][/font][font='times new roman'][size=14px]30%[/size][/font][font='times new roman'][size=14px],而翻译后修饰蛋白错误率更高,故需联合其他技术完成鉴定;第二,微量测序,首先使经凝胶分离的蛋白直接印迹在[/size][/font][font='times new roman'][size=14px] [/size][/font][font='times new roman'][size=14px]PVDF [/size][/font][font='times new roman'][size=14px]膜,经过一系列操作后将其置于测序仪中进行蛋白质鉴定,但该方法仍然存在一些缺点,如由于酸性水解或者部分降解而产生氨基酸的变异,故应联合其他的蛋白质属性进行鉴定。[/size][/font][font='times new roman'][size=14px]化学蛋白质组学方法[/size][/font][font='times new roman'][size=14px]化学蛋白质组学方法一般先将小分子化合物通过与蛋白质溶液反应,使化学探针或小分子化合物与固相联接,得到被修饰的固相微球,然后利用合适的分离技术将这些蛋白质纯化,结合[/size][/font][font='times new roman'][size=14px] [/size][/font][font='times new roman'][size=14px][url=https://insevent.instrument.com.cn/t/Yp][color=#3333ff]LC-MS[/color][/url] [/size][/font][font='times new roman'][size=14px]分析,得到靶蛋白的信息。[/size][/font][font='宋体']亲和色谱法[/font][font='times new roman'][size=14px]亲和色谱法是化学蛋白质组学策略中较为经典的方法之一,它主要应用于研究蛋[/size][/font][font='times new roman'][size=14px]白质与生物活性小分子或蛋白质与蛋白质的相互作用[/size][/font][font='times new roman'][sup][size=14px][17][/size][/sup][/font][font='times new roman'][size=14px]。该方法通过官能团将配体结合在固相基质中,然后与蛋白质孵育,此时与配体结合的蛋白会留在基质上,最后通[/size][/font][font='times new roman'][size=14px]过变性或与自由配体竞争将结合蛋白洗脱下来,再通过凝胶电泳或者[/size][/font][font='times new roman'][size=14px] [/size][/font][font='times new roman'][size=14px][url=https://insevent.instrument.com.cn/t/Yp][color=#3333ff]LC-MS[/color][/url][/size][/font][font='times new roman'][size=14px] [/size][/font][font='times new roman'][size=14px]进行分[/size][/font][font='times new roman'][size=14px]析。该方法的缺陷在于所研究分子衍生物活性不确定、材料配体结合力差异性以及非特异性吸附都将会干扰研究结果。[/size][/font][font='宋体']基于活性的化学蛋白质组学技术[/font][font='times new roman'][size=14px]基于活性的化学蛋白质组学技术([/size][/font][font='times new roman'][size=14px]ABPP[/size][/font][font='times new roman'][size=14px])是广泛使用的技术之一。[/size][/font][font='times new roman'][size=14px]ABPP [/size][/font][font='times new roman'][size=14px]是由美国的[/size][/font][font='times new roman'][size=14px] [/size][/font][font='times new roman'][size=14px]Cravatt[/size][/font][font='times new roman'][size=14px] [/size][/font][font='times new roman'][size=14px]课题组在[/size][/font][font='times new roman'][size=14px] [/size][/font][font='times new roman'][size=14px]2002 [/size][/font][font='times new roman'][size=14px]年首次提出,最早用于酶谱分析,之后被应用于药物靶蛋白筛选。[/size][/font][font='times new roman'][size=14px]ABPP [/size][/font][font='times new roman'][size=14px]技术的关键是合成同时带有反应基团和标记物的[/size][/font][font='times new roman'][size=14px] [/size][/font][font='times new roman'][size=14px]ABPP [/size][/font][font='times new roman'][size=14px]探针,可进一步与待测的蛋白质发生相互反应。药物靶点筛选领域设计的[/size][/font][font='times new roman'][size=14px] [/size][/font][font='times new roman'][size=14px]ABPP [/size][/font][font='times new roman'][size=14px]探针通常包括三[/size][/font][align=center][font='times new roman'][size=14px]3[/size][/font][/align][align=center][/align][font='times new roman'][size=14px]个功能部分:反应基团、连接基团和报告基团。与二维凝胶电泳法([/size][/font][font='times new roman'][size=14px]2-DE[/size][/font][font='times new roman'][size=14px])、同位素编码亲和标签([/size][/font][font='times new roman'][size=14px]ICAT[/size][/font][font='times new roman'][size=14px])等技术相比,[/size][/font][font='times new roman'][size=14px]ABPP [/size][/font][font='times new roman'][size=14px]技术着重研究蛋白质的表达和功能,[/size][/font][font='times new roman'][size=14px] [/size][/font][font='times new roman'][size=14px]可从天然蛋白质组样品中直接筛选出与小分子特异性结合的蛋白质,从而能更直接快速地明确小分子和蛋白质之间的相互作用,确定小分子的作用靶点,这对于筛选具有低亲和力的靶蛋白极为有利。另外,根据富集和鉴定策略的不同,[/size][/font][font='times new roman'][size=14px]ABPP [/size][/font][font='times new roman'][size=14px]技术可分为竞争性标记方法和生物正交的探针模拟物标记方法。[/size][/font][font='times new roman'][size=14px]1.%2.%3 [/size][/font][font='times new roman'][size=14px]非化学修饰的蛋白质组学方法[/size][/font][font='times new roman'][size=14px]1.1.%3.%4 [/size][/font][font='times new roman'][size=14px]细胞热位移测定[/size][/font][font='times new roman'][size=14px]在过去[/size][/font][font='times new roman'][size=14px] [/size][/font][font='times new roman'][size=14px]20 [/size][/font][font='times new roman'][size=14px]年中,热位移分析([/size][/font][font='times new roman'][size=14px]TSA[/size][/font][font='times new roman'][size=14px])已成为最广泛使用的无修饰药物靶点发现方法之一。这种方法简单直接,但探针无法区分不同的蛋白质,因此[/size][/font][font='times new roman'][size=14px] [/size][/font][font='times new roman'][size=14px]TSA [/size][/font][font='times new roman'][size=14px]仅适用于纯化蛋白质的实验。为了规避这个问题,[/size][/font][font='times new roman'][size=14px]Molina [/size][/font][font='times new roman'][size=14px]等人开发了一种概念上相似的技术,称为[/size][/font][font='times new roman'][size=14px]细胞热位移测定[/size][/font][font='times new roman'][size=14px]([/size][/font][font='times new roman'][size=14px]C[/size][/font][font='times new roman'][size=14px]ETSA[/size][/font][font='times new roman'][size=14px])[/size][/font][font='times new roman'][size=14px],用于直接研究细胞环境中的药物[/size][/font][font='times new roman'][size=14px]-[/size][/font][font='times new roman'][size=14px]靶标相互作用。如图[/size][/font][font='times new roman'][size=14px] [/size][/font][font='times new roman'][size=14px]([/size][/font][font='times new roman'][size=14px]A[/size][/font][font='times new roman'][size=14px])所示,细胞裂解物或完整细胞的多个等分试样首先用药物或载体处理,加热到不同的温度并冷却,然后通过离心分离出可溶性部分。随着温度的升高,蛋白质逐渐展开以暴露疏水核,导致蛋白质在高温下沉淀。蛋白质越稳定,蛋白质对热诱导沉淀的抵抗力越高,因此,可以测定可溶性蛋白质随温度变化的稳定性曲线。例如,该[/size][/font][url=https://www.sciencedirect.com/topics/chemistry/antifolate][font='times new roman'][size=14px]方法通过叶酸[/size][/font][/url][font='times new roman'][size=14px]抗癌药物[/size][/font][url=https://www.sciencedirect.com/topics/chemistry/methotrexate][font='times new roman'][size=14px]甲氨蝶呤[/size][/font][/url][font='times new roman'][size=14px]与[/size][/font][url=https://www.sciencedirect.com/topics/chemistry/dihydrofolates][font='times new roman'][size=14px]二氢叶酸[/size][/font][/url][font='times new roman'][size=14px]还原酶的结合、雷替曲塞与胸苷酸合成酶的结合进行了药物靶标的验证。[/size][/font][font='times new roman'][size=14px]CETSA[/size][/font][font='times new roman'][size=14px] [/size][/font][font='times new roman'][size=14px]是一种允许研究活细胞中药物靶点的方法。[/size][/font][font='times new roman'][size=14px]CETSA[/size][/font][font='times new roman'][size=14px] [/size][/font][font='times new roman'][size=14px]结合小分子库可用于筛选潜在抑制剂、评估靶标参与效率和监测靶标特异性。此外,[/size][/font][font='times new roman'][size=14px]CETSA[/size][/font][font='times new roman'][size=14px] [/size][/font][font='times new roman'][size=14px]还可用于筛选[/size][/font][font='times new roman'][size=14px]新药和表型化合物的靶点,解决脱靶蛋白、结合机制、药物疗效和完整细胞耐药性等[/size][/font][font='times new roman'][size=14px]问题。[/size][/font][font='times new roman'][size=14px]1.2.%3.%4 [/size][/font][font='times new roman'][size=14px]热蛋白组学分析[/size][/font][font='times new roman'][size=14px]热蛋白组学分析([/size][/font][font='times new roman'][size=14px]TPP[/size][/font][font='times new roman'][size=14px])首先将蛋白在有或无活性小分子情况下孵育,并加热到不同的温度以诱导蛋白变性,剩余的可溶性蛋白用缓冲液提取。如图所示,在每个温度下,可溶性蛋白通过高分辨质谱进行量化,画出变性曲线,进一步测[/size][/font][align=center][font='times new roman'][size=14px]4[/size][/font][/align][align=center][/align][font='times new roman'][size=14px]定热稳定性和识别配体引起的变化。其中,[/size][/font][font='times new roman'][size=14px]50% [/size][/font][font='times new roman'][size=14px]蛋白发生聚沉时的温度为[/size][/font][font='times new roman'][size=14px] [/size][/font][font='times new roman'][size=14px]Tm[/size][/font][font='times new roman'][size=14px]([/size][/font][font='times new roman'][size=14px]melting temp[/size][/font][font='times new roman'][size=14px]e[/size][/font][font='times new roman'][size=14px]r[/size][/font][font='times new roman'][size=14px]a[/size][/font][font='times new roman'][size=14px]tur[/size][/font][font='times new roman'][size=14px]e[/size][/font][font='times new roman'][size=14px])[/size][/font][font='times new roman'][size=14px],通过对比加药前后[/size][/font][font='times new roman'][size=14px] [/size][/font][font='times new roman'][size=14px]T[/size][/font][font='times new roman'][size=14px]m[/size][/font][font='times new roman'][size=14px] [/size][/font][font='times new roman'][size=14px]的变化,确定活性分子的靶蛋白。[/size][/font][font='times new roman'][size=14px]TPP[/size][/font][font='times new roman'][size=14px] [/size][/font][font='times new roman'][size=14px]可以[/size][/font][font='times new roman'][size=14px]通过定量[/size][/font][font='times new roman'][size=14px] [/size][/font][font='times new roman'][size=14px]MS [/size][/font][font='times new roman'][size=14px]分析,在蛋白质组水平评估活细胞中活性分子与蛋白结合的情况。[/size][/font][font='times new roman'][size=14px]图[/size][/font][font='times new roman'][size=14px] [/size][/font][font='times new roman'][size=14px] [/size][/font][font='times new roman'][size=14px]([/size][/font][font='times new roman'][size=14px]A[/size][/font][font='times new roman'][size=14px])[/size][/font][font='times new roman'][size=14px]细胞热位移测定和[/size][/font][font='times new roman'][size=14px]([/size][/font][font='times new roman'][size=14px]B[/size][/font][font='times new roman'][size=14px])[/size][/font][font='times new roman'][size=14px]热蛋白组学分析简要工作流程[/size][/font][font='times new roman'][size=14px][14][/size][/font][font='times new roman'][size=14px]药物亲和反应的靶点稳定性技术[/size][/font][font='times new roman'][size=14px]药物亲和反应的靶点稳定性技术([/size][/font][font='times new roman'][size=14px]DARTS[/size][/font][font='times new roman'][size=14px])是一种鉴定药物靶标的新方法。药物[/size][/font][font='times new roman'][size=14px]与靶蛋白结合后,靶蛋白对蛋白酶的敏感性降低,与对照组相比,药物结合蛋白更不[/size][/font][url=https://www.sciencedirect.com/topics/chemistry/protein-hydrolysis][font='times new roman'][size=14px]易水解[/size][/font][/url][font='times new roman'][size=14px]。这种差异可通过蛋白凝胶电泳和质谱等技术对差异蛋白进行鉴定,可以确定[/size][/font][font='times new roman'][size=14px]药物直接作用的靶点蛋白[/size][/font][font='times new roman'][size=14px],最大优势是不需要对药物进行任何化学修饰,即可以确定药物的直接结合蛋白。[/size][/font][font='times new roman'][size=14px]DARTS[/size][/font][font='times new roman'][size=14px] [/size][/font][font='times new roman'][size=14px]在天然小分子靶点的鉴定中发挥了重要的作用,例如[/size][/font][font='times new roman'][size=14px]对[/size][/font][font='times new roman'][size=14px] [/size][/font][font='times new roman'][size=14px]ecumicin[/size][/font][font='times new roman'][size=14px] [/size][/font][font='times new roman'][size=14px]、白藜芦醇[/size][/font][font='times new roman'][size=14px]([/size][/font][font='times new roman'][size=14px] [/size][/font][font='times new roman'][size=14px]resveratrol [/size][/font][font='times new roman'][size=14px])[/size][/font][font='times new roman'][size=14px] [/size][/font][font='times new roman'][size=14px]等多种天然产物蛋白靶点的鉴定[/size][/font][font='times new roman'][sup][size=14px]。但[/size][/sup][/font][font='times new roman'][size=14px]DARTS[/size][/font][font='times new roman'][size=14px] [/size][/font][font='times new roman'][size=14px]也存在局限性,例如细胞裂解液中的低丰度蛋白的鉴定和非特异性结合会导致[/size][/font][font='times new roman'][size=14px]蛋白对蛋白酶的敏感性升高增加。利用这一特性,研究者还开发了药物亲和力响应靶稳定性的方法用于药物靶点筛选。[/size][/font]

http://ng1.17img.cn/bbsfiles/images/2017/01/201701191653_634410_1632253_3.gif【论坛技术讲座第44期】生物质谱深入解析蛋白质--鉴定、确证与量化分析的完整解决方案主讲人:刘麟 AB SCIEX中国公司产品经理 活动时间:2010年12月13日 下午 3:00刘麟 简介国内最早从事生物质谱应用研究的专家之一。从事生物质谱仪应用开发10年以上,既具有很深的生物质谱理论知识和专业技能,又有从事分子生物学和生物化学研究经验,目前就职于美国ABSCIEX公司生物质谱产品经理。http://ng1.17img.cn/bbsfiles/images/2017/01/201701191653_634410_1632253_3.gif1、报名条件:只要您是仪器网注册用户均可参加。2、报名参加人数:无人数限制(先到先得!报名后临时不能参加请回帖说明,无故未参加者将扣20积分)3、观看方法:点击http://ng1.17img.cn/bbsfiles/images/2010/12/201012141607_266956_244_3.jpg4、参与互动:本次讲座采取网络讲堂直播模式,欢迎大家积极发言提问。 5、环境配置:只要您有电脑、外加一个耳麦就能参加。6、参加奖励:报名且参与讲座的人将每人奖励5--50分不等的奖励。 7、提问时间:现在就可以在此帖提问啦,截至2010年12月13日 8、会议进入:2010年12月13日14:00点就可以进入会议室9、开课时间:2010年12月13日15:0010、特别说明:注意!只有先申请报名,待审核完才能进入会议室。报名并通过审核将会收到1 封电子邮件通知函(您已注册培训课程),请注意查收,并按提示进入会议室!注意:为了使您的报名申请顺利通过,请填写完整而正确的信息哦~非网站用户将不予以通过哦!快来参加吧:我要报名》》》快来提问吧:我要提问》》》
http://img.dxycdn.com/trademd/upload/asset/meeting/2013/09/06/A1378379551.jpg 氨基酸是蛋白质的基础组成单位,通过研究蛋白质中氨基酸的性质和组成来预测蛋白质的结构和功能,蛋白质氨基酸残基组成分析主要是通过氨基酸分析仪来完成的,本文推荐了2个基于氨基酸组成进行蛋白质预测软件。基于氨基酸组成的蛋白质预测软件根据组成蛋白质的20种氨基酸的物理和化学性质可以辨析电泳等实验中的未知蛋白质,也可以分析已知蛋白质的物化性质。ExPASy工具包包涵的程序:AACompIdent:与把氨基酸序列在SWISS-PROT库中搜索不同,AACompIdent工具利用未知蛋白的氨基酸组成去确认具有相同组成的已知蛋白。该程序分析时需提交的相关信息包括:蛋白质的氨基酸组成、等电点pI和分子量(如果知道)、正确的物种分类及特别的关键词。此外,用户还需在六种氨基酸“组合”中作出选择,这影响到分析如何进行。例如,某种“组合”会把残基Asp/Asn(D/N)和Gln/Glu(Q/E)组合成 Asx(B)和Glx(Z);或者某种残基会在分析中被完全除去。对数据库中的每一个蛋白序列,算法会对其氨基酸组成与所查询的氨基酸组成的差异打分。由电子邮件返回的结果被组织成三级列表:第一张列表中的蛋白都基于特定的物种分类而不考虑pI和分子量;第二张列表包含了不考虑物种分类、pI和分子量的全体蛋白;第三张列表中的蛋白不但基于特定物种分类,并且将 pI和分子量也考虑在内。虽然计算所得结果各不相同,但零分表明了该序列与提出的组成完全相符。AACompSim:AACompIdent的一个变种,AACompSim提供类似的分析,但与前者以实验所得的氨基酸组成为依据进行搜索不同,后者使用SWISS-PROT中的序列为依据。有报道称,氨基酸组成在物种之间是十分保守的(Cordwell等,1995),并且通过分析氨基酸的组成,研究者能从低于25%序列相似性的蛋白之间发现弱相似性(Hobohm和Sander,1995)。因此,在“传统的”数据库搜索基础上辅以组成分析,能为蛋白质之间关系提供更多见解。PROSEARCH:PROPSEARCH也提供基于氨基酸组成的蛋白质辨识功能。用144种不同的物化性质来分析蛋白质,包括分子量、巨大残基的含量、平均疏水性、平均电荷等,把查询序列的这些属性构成的“查询向量”与SWISS-PROT和PIR中预先计算好的各个已知蛋白质的属性向量进行比较。这个工具能有效的发现同一蛋白质家族的成员。可以通过Web使用这个工具,用户只需输入查询序列本身。分子量搜索(MOWSE)分子量搜索(MolecularWeightSearch,MOWSE)算法利用了通过质谱(MS)技术获得的信息。利用完整蛋白质的分子量及其被特定蛋白酶消化后产物的分子量,一种未知蛋白质能被准确无误地确认,给出由若干实验才能决定的结果。由于未知蛋白无需再全部或部分测序,这一方法显著地减少了实验时间。MOWSE的输入是一个纯文本文件,包含一张实验测定的肽段列表,分子量范围在0.7到4.0Kda之间。计算过程基于在OWL非冗余蛋白质序列库中包含的信息。打分基于在一定分子量范围内蛋白中一个片段分子量出现的次数。输出的结果是得分最佳的30个蛋白的列表,包括它们在OWL中的条目名称、相符肽段序列、和其它统计信息。模拟研究得出在使用5个或更少输入肽段分子量时,准确率为99%。该搜索服务可通过向mowse@daresburg.ac.uk发送电子邮件实现。为获得更多关于查询格式的细节信息,可以相该地址发送电子邮件,并在消息正文中写上“help”这个词。蛋白质氨基酸组成分析用盐酸在110 ℃将蛋白或多肽水解成游离的氨基酸,用氨基酸分析仪测定各氨基酸的含量。采用经典的阳离子交换色谱分离、茚三酮柱后衍生法,对蛋白质水解液及各种游离氨基酸的组分含量进行分析。仪器基本结构同普通HPLC相似,但针对氨基酸分析进行了细节优化(例如氮气保护、惰性管路、在线脱气、洗脱梯度及柱温梯度控制等等)通常细分为两种系统:蛋白水解分析系统(钠盐系统)和游离氨基酸分析系统(锂盐系统),利用不同浓度和pH值的柠檬酸钠或柠檬酸锂进行梯度洗脱。其中钠盐系统一次最多分析约25种氨基酸,速度较快,基线平直度好;锂盐系统一次最多分析约50种氨基酸,速度较慢,基线一般不如钠盐系统好。分析效果:从目前已知的氨基酸分析方法比较来看,除灵敏度(即最低检测限)比HPLC柱前衍生方法稍低以外(HPLC:0.5 pmol;氨基酸分析仪:10 pmol),其他如分离度、重现性、操作简便性、运行成本等方面,都优于其他分析方法。蛋白质氨基酸残基组成分析的主要步骤包括:首先是蛋白被水解为氨基酸,其次是采用离子色谱等方法进行游离的氨基酸含量和组成的分析。总之利用蛋白可以分析氨基酸,利用氨基酸也可以研究蛋白质。
蛋白质组学研究--生物质谱技术 对分离的蛋白质进行鉴定是蛋白质组研究的重要内容,蛋白质微量测序、氨基酸组成分析等传统的蛋白质鉴定技术不能满足高通量和高效率的要求,生物质谱技术是蛋白质组学的另一支撑技术。 生物质谱技术在离子化方法上主要有两种软电离技术,即基质辅助激光解吸电离(matrix—assisted laser desorption/ionization,MALDl)和电喷雾电离(electrospray ionization,ESl)。MALDI是在激光脉冲的激发下,使样品从基质晶体中挥发并离子化。ESI使分析物从溶液相中电离,适合与液相分离手段(如液相色谱和毛细管电泳)联用。MALDI适于分析简单的肽混合物,而液相色谱与ESI—MS的联用(LC—MS)适合复杂样品的分析。 软电离技术的出现拓展了质谱的应用空间,而质量分析器的改善也推动了质谱仪技术的发展。生物质谱的质量分析器主要有4种:离子阱(iontrap,IT)、飞行时间(TOF)、四极杆(quadrupole)和傅立叶变换离子回旋共振(Fourier transform ion cyclotron resonance,FTICR)。它们的结构和性能各不相同,每一种都有自己的长处与不足。它们可以单独使用,也可以互相组合形成功能更强大的仪器。 离子阱质谱灵敏度较高,性能稳定,具备多级质谱能力,因此被广泛应用于蛋白质组学研究,不足之处是质量精度较低。与离子阱相似,傅立叶变换离子回旋共振(FTICR)质谱也是一种可以“捕获”离子的仪器,但是其腔体内部为高真空和高磁场环境,具有高灵敏度、宽动态范围、高分辨率和质量精度(质量准确度可很容易地小于1mg/L),这使得它可以在一次分析中对数百个完整蛋白质分子进行质量测定和定量。FTICR—MS的一个重要功能是多元串级质谱,与通常的只能选一个母离子的串级质谱方式不同,FTICR—MS可以同时选择几个母离子进行解离,这无疑可以大大增加蛋白质鉴定工作的通量。但是它的缺点也很明显,操作复杂、肽段断裂效率低、价格昂贵等,这些缺点限制了它在蛋白质组学中的广泛应用。MALDI通常与TOF质量分析器联用分析肽段的精确质量,而ESI常与离子阱或三级四极杆质谱联用,通过碰撞诱导解离(collision—induceddissociation,CID)获取肽段的碎片信息。 1. MALDI—TOF—MS (1)MALDI—TOF—MS的技术特点:①具有分离、鉴定双重功能,可用于混合物的分析;②测量范围宽,相对分子质量可达300 000;③精度高,蛋白质相对分子质量测定精度可达0.01%;④灵敏度高,所需样品量少,可达fmol级;⑤分析时间短,5~10rain可完成一次分析;⑥样品制备简便,操作易自动化;⑦对样品要求低,能忍耐较高浓度的盐、缓冲剂和非挥发性杂质,所以特别适合于鉴定二维凝胶电泳分离的蛋白质。 (2)MALDI—TOF—MS的技术改进:近年来MALDI—TOF—MS又有许多新的技术改进,以提高其检测灵敏性和准确度,增强其蛋白质鉴定的功能。如将MALDI离子源与四极杆—飞行时间—串联质谱对接,实现了同一样品在同一质谱仪上的肽指纹图谱与肽序列标签分析同时进行,提高于蛋白质鉴定的速度和准确性。还有MALDI—TOF—TOF—MS等。但这些技术共同的缺点是仪器非常昂贵。 (3)MALDI—TOF—MS的发展方向:①寻求新的基质(如混合基质、室温离子化液体等)和新的制样方法(如超微量进样,n1级);②将新技术应用在分析中(如酶切技术、毫微升溶剂提取技术等);③对质谱仪进行改进或与其他分析仪器联用,如MALDI与傅立叶转换离子回旋共振质谱(MALDI—FTMS)联用能得到更多的蛋白结构信息;④小型化、智能化、简易化及自动化已成为趋势。 2.ESI (1)ESI的优势:①检测范围宽,生物大分子经电喷雾后质荷比大大下降,因而可测相对分子质量高达十几万甚至更高的生物样品;②分辨率和灵敏度高,可达10-15—10-12mol;③不需要特定基质,避免子基质峰的干扰;④适用于结构分析,可分析生物大分子的构象及非共价相互作用,与串联质谱结合可分析蛋白质、多肽的一级结构和共价修饰位点等;⑤自动化程度高,可与液相色谱、毛细管电泳等高效分离手段在线联用;⑥MALDI是脉冲式离子化技术,而ESI是连续离子化技术,检测所需时间更短;⑦ESI常和四极杆质谱联用,仪器价格相对便宜。 (2)ESI的局限性:①对样品中的盐类耐受性差;②对混合物图谱的解析较复杂;③受溶剂的影响和限制很大。目前ESI主要用于亲水生物大分子的分析,较少用于疏水生物样品的分析。总体来说,MALDI和ESI各有长处,有各自的适用范围,是两种互补的技术。 (3)ESI的技术发展:近年来,ESI也有许多新的技术发展。液相色谱与电喷雾质谱连用(LC—ESI—
[font=宋体][font=宋体]泛素化是一种细胞内的蛋白质标记系统,蛋白质泛素化是指将小的蛋白质泛素共价地连接到其他蛋白质分子上的过程。泛素([/font][font=Calibri]ubiquitin[/font][font=宋体])是一种高度保守的蛋白质,其结构由[/font][font=Calibri]76[/font][font=宋体]个氨基酸残基组成。泛素连接到目标蛋白质上的过程,经历了泛素激活、泛素转移和靶蛋白接受三个主要步骤。[/font][/font][font=宋体] [/font][font=宋体]蛋白质泛素化具有多种特点,例如它是高度选择性的,不同蛋白质泛素化的位置和数量可以影响其功能;它是可逆的,通过去泛素化反应可以调控蛋白质的泛素化状态;它还是动态调控的,受到多种因素的调控,如细胞信号通路和环境刺激。[/font][b][font=宋体]泛素化蛋白大小:[/font][/b][font=宋体] [/font][font=宋体][font=宋体]蛋白泛素化是指将小蛋白颗粒泛素([/font][font=Calibri]Ubiquitin[/font][font=宋体])与其他蛋白质共价结合的修饰过程。 泛素化修饰通常会导致泛素共价连接在蛋白质的赖氨酸残基上形成多重泛素链。 这种蛋白质泛素化增加了蛋白质的分子量,因为每个泛素分子的质量大约为[/font][b][font=Calibri]8.5[/font][font=宋体]千达尔顿([/font][font=Calibri]kDa[/font][/b][font=宋体][b])[/b]。[/font][/font][font=宋体] [/font][font=宋体] [/font][b][font=宋体]泛素化蛋白质组学在许多领域有重要的应用,主要包括:[/font][/b][font=宋体] [/font][font=宋体]①疾病机制研究:泛素化是一种广泛存在于细胞中的蛋白质修饰方式,参与了细胞的生长、分化、修复和调控等多个生命活动。泛素化蛋白质组学的研究可以帮助我们了解泛素化修饰的生物学功能和调控机制,为疾病发生机制和治疗策略的研究提供重要线索。例如,在癌症、代谢综合征、神经退行性疾病等疾病中,则会出现异常泛素化。[/font][font=宋体]②药物研发:通过分析药物对泛素化蛋白质的影响,可以评估药物的效力和选择性,为药物研发提供指导。[/font][font=宋体]③临床诊断:泛素化蛋白质组学鉴定与定量分析技术可以揭示细胞调控的机制,通过分析泛素化蛋白质的组学数据,可以确定泛素化修饰在细胞信号转导、蛋白质降解和细胞周期调控等过程中的重要作用。此外,通过比较病态和正常样品中泛素化蛋白质的差异,可以鉴定与疾病发生发展相关的泛素化修饰靶点,并进一步理解疾病的分子机制。因此,这些技术也可用于临床诊断。[/font][font=宋体]④蛋白质降解调控:在癌症、神经退行性疾病和免疫相关疾病等病症中,蛋白质降解调控出现异常。而泛素化蛋白组在调控蛋白质降解中发挥重要作用。通过与泛素连接,目标蛋白质被送入蛋白酶体或蛋白酶体样体中进行降解。这个过程是细胞清除异常、老化或受损蛋白质的重要途径。[/font][font=宋体]⑤高通量技术应用:高通量泛素化蛋白质组学鉴定与定量分析技术的发展包括质谱鉴定和抗体鉴定两种方法。质谱鉴定技术利用质谱仪的高灵敏度和分辨率,能够鉴定泛素化修饰的蛋白质及其泛素化位点。抗体鉴定技术则通过特异性抗体的使用,可以富集和鉴定泛素化修饰的蛋白质。这些技术为全面了解泛素化在细胞中的作用机制和调控网络提供了可能。[/font][font=宋体]总的来说,泛素化蛋白质组学在多个领域都有重要的应用价值,推动了我们对生命过程的深入理解以及疾病治疗的创新发展。[/font][font=宋体] [/font][font=宋体]更多详情关于[url=https://cn.sinobiological.com/resource/protein-review][b]蛋白资源[/b][/url]详情可以参看:[/font][url=https://cn.sinobiological.com/resource/protein-review][u][font=宋体][color=#0000ff][font=Calibri]https://cn.sinobiological.com/resource/protein-review[/font][/color][/font][/u][/url][b][font=宋体] [/font][font=宋体][font=宋体]义翘神州:蛋白与抗体的专业引领者,欢迎通过百度搜索[/font][font=宋体]“义翘神州”与我们取得联系。[/font][/font][/b]
有没有人做过质谱绝对定量蛋白的?
一、 前言基因工程已令人难以置信的扩展了我们关于有机体DNA序列的认识。但是仍有许多新识别的基因的功能还不知道,也不知道基因产物是如何相互作用从而产生活的有机体的。功能基因组试图通过大规模实验方法来回答这些问题。但由于仅从DNA序列尚不能回答某基因的表达时间、表达量、蛋白质翻译后加工和修饰的情况、以及它们的亚细胞分布等等,因此在整体水平上研究蛋白质表达及其功能变得日益显得重要。这些在基因组中不能解决的问题可望在蛋白质组研究中找到答案。蛋白质组研究的数据与基因组数据的整合,将会在后基因组研究中发挥重要作用。目前蛋白质组研究采用的主要技术是双向凝胶电泳和质谱方法。双向凝胶电泳的基本原理是蛋白质首先根据其等电点,第一向在pH梯度胶内等电聚焦,然后转90度按他们的分子量大小进行第二向的SDS-PAGE分离。质谱在90年代得到了长足的发展,生物质谱当上了主角,蛋白质组学又为生物质谱提供了一个大舞台。他们中首选的是MALDI-TOF,其分析容量大,单电荷为主的测定分子量高达30万,干扰因素少,适合蛋白质组的大规模分析。其次ESI为主的LC-MS联机适于精细的研究。本文将简介几种常用的生物质谱技术,并着重介绍生物质谱技术在蛋白质组学各领域的应用。二、 生物质谱技术1.电喷雾质谱技术(ESI)电喷雾质谱技术( Electrospray Ionization Mass Spectrometry , ESI - MS) 是在毛细管的出口处施加一高电压,所产生的高电场使从毛细管流出的液体雾化成细小的带电液滴,随着溶剂蒸发,液滴表面的电荷强度逐渐增大,最后液滴崩解为大量带一个或多个电荷的离子,致使分析物以单电荷或多电荷离子的形式进入气相。电喷雾离子化的特点是产生高电荷离子而不是碎片离子, 使质量电荷比(m/ z) 降低到多数质量分析仪器都可以检测的范围,因而大大扩展了分子量的分析范围,离子的真实分子质量也可以根据质荷比及电荷数算出。2.基质辅助激光解吸附质谱技术(MOLDI)基质辅助激光解析电离(MOLDI)是由德国科学家Karas和Hillenkamp发现的。将微量蛋白质与过量的小分子基体的混合液体点到样品靶上,经加热或风吹烘干形成共结晶,放入离子源内。当激光照射到靶点上时,基体吸收了激光的能力跃迁到激发态,导致蛋白质电离和汽化,电离的结果通常是基体的质子转移到蛋白质上。然后由高电压将电离的蛋白质从离子源转送到质量分析器内,再经离子检测器和数据处理得到质谱图。TOF质量分析器被认为是与MALDI的最佳搭配,因为二者都是脉冲工作方式,在质量分析过程中离子损失很少,可以获得很高的灵敏度。TOF质量分析器结果简单,容易换算,蛋白质离子在飞行管内的飞行速度仅与他的(m/z)-1/2成正比,因此容易通过计算蛋白质离子在飞行管内的飞行时间推算出蛋白质离子的m/z值。与传统质量分析器相比,更易得到高分辨率和高测量精度;速度快,离子飞行时间仅为几个μs和约100μs之间;质量范围宽,可以直接检测到几十万道尔顿的单电荷离子。飞行时间质量分析器被认为是21世纪最有应用前景的质量分析器。3.傅立叶变换-离子回旋共振质谱(FT-ICR MS)傅立叶变换-离子回旋共振质谱法(FT-ICR MS)是离子回旋共振波谱法与现代计算机技术相结合的产物。傅立叶变换-离子回旋共振质谱法是基于离子在均匀磁场中的回旋运动, 离子的回旋频率、半径、速度和能量是离子质量和离子电荷及磁场强度的函数, 当对离子施加与其回旋频率相同的射频场作用时, 离子将同相位加速到一较大的半径回旋, 从而产生可被接受的类似电流的信号。傅立叶变换-离子回旋共振质谱法所采用的射频范围覆盖了欲测定的质量范围,所有离子同时被激发, 所检测的信号经过傅立叶变换, 转换为质谱图。其主要优点有:容易获得高分辨;便于实现串极质谱分析;便于使用外电离源并与色谱仪器联用。此外,他还有灵敏度高,质量范围宽,速度快,性能可靠等优点。4.快原子轰击质谱技术(FABMS)快原子轰击质谱技术( Fast Atom Bomebardment Mass Spectrometry , FABMS) 是一种软电离技术,是用快速惰性原子射击存在于底物中的样品,使样品离子溅出进入分析器,这种软电离技术适于极性强、热不稳定的化合物的分析,特别适用于多肽和蛋白质等的分析研究。FABMS能提供有关离子的精确质量,从而可以确定样品的元素组成和分子式。而FABMS -MS 串联技术的应用可以提供样品较为详细的分子结构信息,从而使其在生物医学分析中迅速发展起来。三、蛋白质的分析鉴定随着质谱技术的发展,分子量的测定已从传统的有机小分子扩展到了生物大分子。MALDI-MS技术以其极高的灵敏度、精确度在蛋白质分析中得到了广泛的应用。该技术不仅可测定各种疏水性、亲水性和糖蛋白的分子量,还可直接测定蛋白质混合物的分子量。这可认为是蛋白质分析领域的一项重大突破。蛋白质组的研究是从整体水平上研究细胞或有机体内蛋白质的组成及其活动规律。质谱技术作为蛋白质组研究的三大支撑技术之一,除了用于多肽,蛋白质的分子量测定外,还广泛的应用于肽指纹图谱测定及氨基酸序列测定。肽指纹图谱(Peptide Mass Fingerprinting, PMF)测定是对蛋白酶解或降解后所得多肽混合物进行质谱分析的方法。质谱分析所得肽断与多肽蛋白数据库中蛋白质的理论肽断进行比较,判断出所测蛋白是已知还是未知。由于不同的蛋白质具有不同的氨基酸序列,不同蛋白质所得肽断具有指纹特征。采用肽指纹谱的方法已对酵母、大肠杆菌、人心肌等多种蛋白质组进行了研究。对肽序列的测定往往要应用串连质谱技术,采用不同的技术选择特定质核比的离子,并对其进行碰撞诱导解离,通过分析肽段的断裂情况推导出肽序列。四、后转录修饰的蛋白质的检测和识别在蛋白质组的研究中,蛋白质和多肽的序列分析已不局限于阐明蛋白质的一级结构,对翻译后的修饰的进一步分析也是蛋白质化学的一项重要任务。这种修饰对于蛋白质的功能非常重要,如:细胞识别中的蛋白质相互作用,信号传导和蛋白质定位。1. 蛋白质的糖基化糖蛋白在细胞内部,细胞膜和细胞外均有发现,实际上大部分蛋白质是糖蛋白。对糖蛋白的检测和分析发现,糖蛋白中糖组分的结构和功能具有多样性。糖蛋白中的糖通常是不同种类的,而且是由一些可控数量的单糖组成。糖基化的多样性与细胞周期,细胞分化和发展的状态有关。在蛋白组时代中,蛋白质的修饰会引起其理化性质的改变,因此是不容忽视的。从1D或2D凝胶得到的糖基化蛋白的识别,一般是进行MALDI-MS指纹分析, 或是对MALDI-PAD或ESI-MS/MS得到的碎片谱进行分析。对完整的糖蛋白的研究是非常困难的,所有已知的离子化技术都有其局限性。目前,人们主要研究糖肽,其好处之一就是质量减小了,这就会得到更好的分辨率,而且糖肽仍保留了糖基化位点。将分离的糖蛋白用不同的蛋白酶消化后就可进行糖肽的研究。一旦糖肽被识别出,就可以用串连质谱(ESI-MS/MS)来阐明肽序列。当蛋白的序列已知时,计算质量差就可推出其上附着的寡糖的质量。要将糖部分从糖蛋白中释放出来,可用化学切割或酶切割(流程图见图1)。目前,连有结构专一性糖苷酶的质谱在提供序列,分支和链接数据方面是最有力的技术。对于N糖基化常用的糖苷内切酶有PNGase-F, PNGase-A, EndoF和EndoH。化学切割也可以用来释放O-连接和N-连接的多糖,但经常出现的缺点是他会完全破坏所有的肽键,因而丢失了关于糖附着位点的信息。而且这些切割不能从糖肽中连续释放单糖。用肼的化学切割可以除去两种类型的糖基化。在60℃可专一性的释放O-连接的糖,而在95℃能释放N-连接的糖。释放O-原子更常用的方法是用碱进行β消除。通常,糖基中加入金属离子在MALDI和ESI中离子化。用MALDI-MS分析糖类的一个好的选择是将之与其他一些化合物混合,这样可以进一步提高灵敏度和分辨率。不同的质谱方法可以产生多糖的源后裂解(PSD)和碰撞诱导解离 (CID)谱,这可以给出有关糖的序列,分支及糖间的连接等信息。2. 蛋白质的磷酸化蛋白质中氨基酸的磷酸化在生命系统中起重要的作用。磷酸化经常作为分子开关控制不同过程蛋白质的活性,如新陈代谢,信号传导,细胞分裂等过程。因此,蛋白质中磷酰氨基酸的识别在蛋白质分析中是一项重要的工作。已知的磷酰氨基酸的类型有四种:1.O-磷酸盐,通过羟氨酸的磷酸化形成的,如丝氨酸,苏氨酸,酪氨酸。2.N-磷酸盐,通过精氨酸,赖氨酸或组氨酸中的氨基的磷酸化形成的。3.乙酰磷酸盐,通过天冬氨酸或谷氨酸的磷酸化形成的。4.S-磷酸酯,通过半胱氨酸的磷酸化形成的。
20世纪基因组学研究取得的巨大成就为蛋白质组学的发展奠定了基础。蛋白质组学是从整体水平上分析生命体、组织或细胞的蛋白质组成及其活动规律的科学,以基因表达产物为研究对象,延伸了基因组学研究深度,更深层次地揭示了生命活动规律。蛋白质组学的研究内容主要包括蛋白质表达存在方式(修饰形式)的鉴定、结构与功能分析、蛋白质定位、蛋白质差异表达以及蛋白质间相互作用分析等[1]。目前蛋白质组学研究技术主要包括:二维电泳技术、蛋白质芯片技术、质谱技术等[2]。其中,二维电泳技术是早期蛋白质组学的重要技术之一,但是由于实验步骤多,耗时长,重复性差等特点,已经逐步被新型技术所取代。蛋白质芯片技术是将多种蛋白质纯品点于芯片表面,形成蛋白质矩阵进行免疫等标记反应,主要受限于很多蛋白质无法获得纯品而不能用于芯片制备。质谱技术由于灵敏度高、特异性强、分析范围宽等优点逐渐成为蛋白质组学的主要研究手段,可以对特定生命过程中的功能性蛋白质分子进行定性和定量检测,因此在基础科研和临床研究中得到了广泛的应用[3,4]。一、基于质谱的蛋白质组学技术1.基于质谱的蛋白质组学定性技术:蛋白质定性鉴定的基本原理在于:蛋白质组的基本序列已经通过基因组学信息获得,可以用来鉴定多肽的氨基酸序列,并且获得多肽与蛋白质的对应关系[1],即质谱提供的多肽碎片数据可以与蛋白质数据库自动匹配来确定多肽序列与蛋白质归属。基本技术策略分为:(1)自上而下(Top–down)策略[5],即完整蛋白质在质谱中进行分析,可以提供完整蛋白质的质量数,但是由于质谱仪受到质量分析范围的限制,此方法在常规实验室不易实现。(2)自下而上(Bottom–up)策略[6],即蛋白质被蛋白酶水解成多肽,然后对多肽进行质谱分析和碎裂。基于这条策略的大致步骤为:蛋白质样品首先经过酶解降解为多肽,然后对多肽进行色谱–质谱分离与鉴定,最后通过搜索引擎(MASCOT:http://www.matrixscience.com/server.html, SEQUEST:http://fields.scripps.edu/sequest等)在公共蛋白质组学数据库(SWISS–PORT: http://web.expasy.org/groups/swissprot, NCBI:http://www.ncbi.nlm.nih.gov/pubmed等)中自动完成质谱数据的解析,确定多肽序列与蛋白质种类。该技术灵敏度高,特异性好,仪器自动化程度高,可以鉴定出生物样品中成千上万种蛋白质,被认为是大规模、高通量蛋白质定性检测的首选方法。2.基于质谱的蛋白质组学相对定量技术:对于大多数生命科学和医学研究来说,仅完成样品中蛋白质组的定性研究是远远不够的,还需要对蛋白质组进行定量分析。由于组学的研究对象是多个蛋白质,单次检测很难实现所有蛋白质的绝对定量,因此蛋白质组学定量多为相对定量检测。蛋白质组学定量的质谱技术包括谱图计数、质谱峰强度定量、同位素定量技术等。其中使用同位素作为内标定量的方法是目前质谱定量的最佳手段,即对整体蛋白质组进行同位素标记,并使用每一种天然蛋白质与同位素蛋白质的比值进行相对定量分析。主要分为细胞层面标记和蛋白质层面标记两种技术路线:(1)细胞层面标记的细胞培养氨基酸稳定同位素标记(stable isotope labeling with amino acids in cell culture,SILAC)方法[7]:即在两种细胞样品中分别加入轻重同位素标记的培养基,经过传代培养后,两种细胞样品中的全部蛋白质中分别嵌合了轻重同位素,可以在质谱上根据同位素的不同质荷比直接判断样品来源并进行定量比对。(2)蛋白质层面标记:使用含有同位素的小分子与样品全部蛋白质直接标记,如同位素标记相对和绝对定量技术(isobaric tags for relative and absolute quantification,iTRAQ)[8]、同位素编码亲和标记(isotope–coded affinity tag,iCAT) [9]、18O标记[10]等方法,此类方法使用带有稳定同位素的小分子与特定氨基酸侧链反应,使得多个样品可以分别连接含有不同同位素个数(多至8个)的小分子,从而产生一级数据相同但是二级数据不同的质谱谱图,通过二级谱图强度比对进行多个样品的定量分析。3.基于质谱的目标蛋白质绝对定量技术:质谱技术对目标蛋白质的绝对定量检测主要通过质谱多反应监控技术与同位素多肽内标技术联用来实现[11]。该方法首先选定目标蛋白质的一个或多个多肽,合成序列相同但含有稳定同位素的多肽作为内标,定量加入样品中,通过监测特定多肽及其同位素多肽的质谱峰强度进行比对和计算获得目标蛋白质的定量值。质谱多反应监控技术通过进行母离子筛选与子离子筛选等二次选择过程,筛选出目标蛋白质,而非目标蛋白质由于无法通过筛选达到检测器,极大降低了噪音干扰。因此,此方法针对性强,本底噪音低,是目前质谱技术中定量能力最好的一种,可以控制变异系数小于15%,检测限低至纳克每毫升,适合血液、组织等临床样品的定量检测[11]。二、质谱技术发现肿瘤蛋白质标志物质谱技术作为一项强有力的研究工具在科学研究中发挥着巨大的作用,特别在肿瘤相关研究中,目前已经获得美国食品药品监督管理局(Food and Drug Administration,FDA)批准的肿瘤标志物包括多种蛋白质前列腺特异性抗原(prostate–specific antigen, PSA), 癌胚抗原(carcinoembryonic antigen CEA), 人类表皮生长因子受体2(human epidermal growth factor receptor 2,Her–2), 人绒毛膜促性腺激素(human chorionic gonadotropin, HCG), 糖类抗原CA125等,均揭示了蛋白质与肿瘤发生发展密切相关。这些已有成果极大促进了质谱技术在肿瘤蛋白质标志物研究中的应用,并取得了标志性进展。例如:美国约翰霍普金斯大学的Chan课题组发现了新型卵巢癌蛋白质标志物,他们使用表面增强激光解析电离质谱技术(surface enhanced laser desorption and ionization time–of–flight mass spectrometry, SELDI–TOF MS)技术对503个妇女的血清进行了蛋白质组学的分析[12],在随后的大量临床验证中最终确定CA125、β2微球蛋白,转铁蛋白,甲状腺运载蛋白和载脂蛋白A1的联合检测可以作为卵巢癌的新型临床诊断指标。2009年9月该试剂盒OVA1(商品名称:http://ova–1.com)获得了美国FDA的认证,进入临床使用,被认为是国际肿瘤蛋白质标志物研究的重要标志性成果。同时,肿瘤仍然是国际上致死率最高的疾病之一,缺乏早期检测技术和有效治疗方案,临床中还存在着大量问题需要解决,新型标志物的研发迫在眉睫。由于肿瘤蛋白质标志物研究的难度大,风险高,因此近十年来仅有几例试剂盒获得了美国FDA批准,进入临床使用。大量标志物研究还停留在论文研究水平,其中临床问题、研究思路和技术方案的选择直接关系到研究的成功与否。1.临床问题选择:在肿瘤蛋白质标志物研究中,临床问题的选择是研究核心。在肿瘤研究中,需要解决的临床问题往往包括肿瘤早期检测、肿瘤分期检测、治疗方案与药物选择、疗效评估等多个方面。研究者需要根据不同肿瘤的临床情况,具体分析并凝练不同肿瘤的主要临床问题。例如,对于病程发展快、五年存活率低、没有有效手术或化疗手段的肿瘤,早期诊断是研究重点,如胰腺癌、卵巢癌、肺癌等;对于病程发展慢、手术效果明显的肿瘤,肿瘤的愈后与复发是需要关注的问题,如前列腺癌、肠癌等;还有一些肿瘤有特殊的检测需求,如乳腺癌虽然有临床有效的雌激素受体(estrogen receptor,ER),孕激素受体(progesterone receptor,PR),HER2等基因标志物,可以进行药物靶点治疗,但是三阴性乳腺癌的检测还缺乏有效的标志物与治疗方案。因此,在肿瘤蛋白质标志物研究实验开展之前,明确临床问题,并以此确定临床样品入组标准,是研究成功的核心基础。2.研究思路设计:不同于基础科学实验,临床实验需要在大量样本中进行实验结果的验证,因此肿瘤蛋白质标志物研究往往包括新型标志物发现和验证两部分。标志物发现实验是在疾病组和对照组之间进行蛋白质组学分析,鉴定样本中的未知蛋白质组并进行相对定量比较,分析数据选择出在两组样本中差异最大的一个或几个蛋白质作为新型标志物的候选物。随后,标志物验证实验在大量未知样本中进行蛋白质候选物的定量检测,使用发现实验中建立的区分标准进行判读,计算检测灵敏性(sensitivity)和特异性(specificity)。有效的蛋白质标志物研究往往需要发现与验证的两步设计思路来相互保证。3.技术方案选择:根据蛋白质标志物研究的两步设计思路,发现实验中使用基于质谱的蛋白质组学定性技术与相对定量技术对样本中的大量未知蛋白质进行分析,获得标志物候选物名单。验证实验中根据已有名单,进行目标蛋白质(非蛋白质组学)的精确定量检测。这几种质谱技术的配合使用,可以满足不同实验情况和目的,最终实现新型蛋白质标志物的成功研发。三、展望质谱技术是现阶段蛋白质组学研究的核心技术,具有灵敏度高、特异性强、分析通量大等优势,特别是其与同位素内标的联合使用,大大提高了质谱定量能力,因此在多种肿瘤标志物研究中取得了突破性进展并被广泛应用。目前,大量肿瘤蛋白质标志物候选物已经通过使用质谱技术被从血液、组织、体液中筛选出来,预计在完成大规模临床验证后可以作为新型标志物在临床使用,促进肿瘤检测水平的发展。同时值得注意的是,质谱技术还不具备进行蛋白质组的绝对定量能力。相对于免疫等传统蛋白质检测技术,仪器昂贵,操作复杂,自动化程度低,这些因素决定了质谱目前适用于蛋白质的临床研究,但不适用于蛋白质的临床检验,这是质谱技术面临的重要挑战之一。参考文献[1]何华勤. 简明蛋白质组学[M]. 北京:中国林业出版社, 2011:1,76,85-95,119,125-138.[2]RuediA, MatthiasM. Mass spectrometry-based proteomics[J]. Nature, 2003, 422(13):198-207.[3]甄艳, 施季森. 质谱技术在蛋白质组学研究中的应用[J]. 南京林业大学学报:自然科学版, 2011, 35(1):103-108.[4]孙瑞祥, 付岩, 李德泉,等. 基于质谱技术的计算蛋白质组学研究[J]. 中国科学E辑信息科学, 2006, 36(2):222-234.[5]WhiteleggeJ,HalgandF,SoudaP, et al. Top-down mass spectrometry of integral membrane proteins [J]. Expert Review Proteomics, 2006, 3(6):585-596.[6]ChaitBT. Mass spectrometry:bottom-up or top-down? [J]. Science, 2006, 314(5796):65-66.[7]TranDT, AdhikariJ, FitzgeraldMC. StableIsotope Labeling with Amino Acids in Cell Culture (SILAC)-based strategy for proteome-wide thermodynamic analysis of protein-ligand binding interactions [J]. Mol Cell Proteomics, 2014,13(7):1800-1813.[8]DytfeldD, KandarpaM, StrahlerJR, et al. Proteomic Profiling of Multiple Myeloma (MM) Cells Using iTRAQ and Label-Free Quantitative Proteomics for the Prediction of Complete or near Complete Response (CR/nCR) In Frontline Treatment with Lenalidomide, Bortezomib, and Dexamethasone [J]. Blood, 2010, 116(21):271-272.[9]García-SantamarinaS, BoronatS, DomènechA, et al. Monitoring in vivo reversible cysteine oxidation in proteins using ICAT and mass spectrometry [J]. Nat Protoc,2014,9(5):1131-1145.[10]MirzaSP, GreeneAS, OlivierM. 18O labeling over a coffee break:a rapid strategy for quantitative proteomics [J]. J Proteome Res, 2008,7(7):3042-3048.[11]曹冬, 张养军, 钱小红. 基于生物质谱的蛋白质组学绝对定量方法研究进展[J]. 质谱学报, 2008, 29(3):185-190.[12]ZhangZ, BastRC, YuY,et al. Three biomarkers identified from serum proteomic analysis for the detection of early stage ovarian cancer[J]. Cancer Res,2004,64(16), 5882-5890.