
如何使用快剪辑,对视频进行后期加工。[align=center][img=,690,975]https://ng1.17img.cn/bbsfiles/images/2020/08/202008211045303511_9614_3299836_3.jpg!w690x975.jpg[/img][/align][align=center][img=,690,975]https://ng1.17img.cn/bbsfiles/images/2020/08/202008211045311851_7354_3299836_3.jpg!w690x975.jpg[/img][/align]
用手机拍的视频,可用爱剪辑软件进行后期制作视频。
"铁公鸡"或源自遗传 科学家发现"吝啬基因" 如果你有一位朋友从来都不请客,甚至都很少愿意AA制,那么你也不必太生气,因为这很可能与他的基因有关系。据英国《每日邮报》11月4日报道,科学家终于找到了“吝啬基因”,这或许可以从遗传学角度解释小气鬼们为什么把钱包捂得这么严实。 德国波恩大学研究人员提取了101位年轻男性和女性嘴里的细胞样本,并在样本中检测一段名为COMT的基因。该基因分成G碱基和A碱基两种类型,其能够影响脑化学,进而有可能左右人们慷慨与否。 在实验中,志愿者被要求去玩一个赌博电脑游戏,然后告诉实验人员他们愿意将赢取的一部分还是全部奖金捐赠给秘鲁的贫困儿童。为了使实验任务更加真实,实验人员还给志愿者呈现了一个名叫莉娜的秘鲁贫困女孩的照片,以及一只由她编织的手镯。 实验结果表明,拥有G碱基的志愿者有超过20%的人将他们赢的所有钱都捐给了莉娜,但是拥有A碱基(即“吝啬基因”)的志愿者仅有不到2%的人能够像G型人这样慷慨。 通常,人类每4人中间大约就有1人携带有“吝啬基因”,他们表现得特别注重自己的钱财,比如时常讨要香烟而不是自己去买,或者定期借钱付公交车车票,但不怎么还钱。而且,那些携带“吝啬基因”的人比其他人捐赠给慈善机构的钱更少。 不过,吝啬的形成也不能完全归咎于基因。之前的研究已经表明,一个人慷慨与否只能部分地用基因来解释,诸如抚养、教育和宗教等其他因素也有不同程度的影响。
本文由丁香园站友 宇宙cosmos 转载,点此查看详情据美国每日科学网站7月22日报道,美国科学家在7月21日出版的《科学》杂志上撰文指出,他们找到了DNA的第7种、第8种碱基,并在人体胚胎干细胞和实验老鼠器官染色体组的DNA中发现了这两个碱基的踪迹。科学家们指出,最新发现对干细胞和癌症研究非常重要。几十年来,科学家们一直认为DNA中只包含有4种碱基:腺嘌呤、鸟嘌呤、胸腺嘧啶和胞嘧啶,这4种碱基已成为我们对基因代码如何形成生命的认识的基础。然而不久前,科学家们将碱基的数量扩展到了6种(第5种碱基:5-胞嘧啶甲基,第6种碱基:5-胞嘧啶甲基羟基)。现在,北卡罗来纳大学医学院生物化学和生物物理学教授张毅(音译)领导的研究团队则表示,他们已经发现了DNA的第7种碱基5-胞嘧啶甲酰(5-formylcytosine)和第8种碱基5-胞嘧啶羧基(5-carboxylcytosine)。科学家指出,最新的这两种碱基实际上都是胞嘧啶经由Tet蛋白修改后得到的“变身”。Tet蛋白是一种分子实体,其在DNA脱甲基过程和干细胞重新编程方面起关键作用。此前,科学家们已对第5种碱基有所了解——当一个化学标签或甲基被固定到一个胞嘧啶上时,第5种碱基就会出现。这个甲基化作用同基因沉默有关,因为它会导致DNA的双螺旋折叠得更加紧密。去年,张毅团队报告称,在一个4步反应的第一步,Tet蛋白能将第5种碱基转变为第6种碱基,但他们没有再接再厉,继续进行该实验,导致他们与第7种、第8种碱基“失之交臂”。研究团队最终发现,问题不在于Tet没有参与第二步和第三步,而是他们的实验工具的敏感度不足以探测到两种新碱基的存在。因此,他们重新设计了实验并探测到了最新的两种DNA碱基,并在人体胚胎干细胞和实验老鼠器官染色体组的DNA中发现了它们的踪迹。张毅表示:“新碱基代表了DNA脱甲基过程中的一个中间状态。通过去甲基化或重新激活DNA甲基化所沉默的肿瘤抑制基因,它们可能为干细胞重新编程和癌症研究提供非常重要的信息。”
碱基编辑(base editing,BE)作为前沿的基因组编辑技术,能够在基因组水平上实现精确、高效的单碱基编辑。该技术广泛应用于基础研究、基因治疗和细胞工厂构建等领域。常用的DNA碱基编辑器主要是通过将可编程的DNA结合蛋白(如Cas9)与碱基脱氨酶融合实现的,包括胞嘧啶碱基编辑器(CBE)、腺嘌呤碱基编辑器(ABE)以及糖基化酶碱基编辑器(GBE)等,可以实现C-to-T、A-to-G以及C-to-G等种类的碱基编辑。然而,这些碱基编辑器是针对C和A碱基的直接编辑,且所包含的脱氨酶可能导致非Cas9依赖的DNA或RNA脱靶。中国科学院天津工业生物技术研究所研究员毕昌昊带领的合成生物技术研究团队,联合研究员张学礼带领的微生物代谢工程研究团队,[b]开发了不依赖脱氨酶(deaminase-free,DAF)的碱基编辑器DAF-CBE和DAF-TBE,分别在大肠杆菌中实现C-to-A、T-to-A的碱基颠换,在哺乳动物细胞中实现C-to-G、T-to-G的碱基颠换编辑。?[/b]该研究通过定向进化改造了人源尿嘧啶糖基化酶(UNG)的两个突变体UNG(N204D)和UNG (Y147A),获得了两种高活性的DNA糖基化酶,分别可以作用于胞嘧啶碱基的CDG4和胸腺嘧啶碱基的TDG3。进而,研究将这两种DNA糖基化酶与nCas9(Cas9、D10A)融合,构建了CDG4-nCas9和TDG3-nCas9两种碱基编辑器,用于在大肠杆菌中进行C-to-A和T-to-A的编辑。实验结果显示,CDG4-nCas9和TDG3-nCas9在大肠杆菌中的编辑效率最高分别达到58.7%和54.3%。进一步,研究针对Homo sapiens密码子优化版本的CDG4-nCas9和TDG3-nCas9,在HEK293T细胞中实现了C-to-G和T-to-G的颠换编辑,编辑效率分别达到38.8%和48.7%。这两种编辑器的脱靶效果低于常用的胞嘧啶碱基编辑器(BE4max)和糖基化酶碱基编辑器(CGBEs)。因此,研究将这两个编辑器命名为DAF-CBE和DAF-TBE。此外,通过进一步的工程改造,该团队优化了CDG和TDG的空间位置,得到了DAF-CBE2和DAF-TBE2的新版本。它们的编辑窗口从原来的间隔序列(protospacer sequence)5'端移动到中间区域,且C-to-G和T-to-G的编辑效率分别提高了3.5倍和1.2倍。DAF-CBE和DAF-TBE实现了人诱导多功能干细胞(hiPSC)高效编辑。综上所述,[b]经过定向进化改造,该团队开发的DAF-CBEs和DAF-TBEs碱基编辑器在大肠杆菌和哺乳动物细胞中实现了高效的碱基颠换编辑,无需使用脱氨酶。与现有的引导编辑器(prime editing)或糖基化酶碱基编辑器(GBEs)相比,DAF-BEs具有相当的编辑效率、更小的尺寸和更低的脱靶率,这扩展了碱基编辑器的编辑类型,为工业菌株铸造和生物医药等领域的相关研究提供了新的技术工具。?[/b]近日,相关研究成果发表在《自然-生物技术》([i]Nature Biotechnology[/i])上。研究工作得到国家重点研发计划、国家自然科学基金、天津市合成生物技术创新能力提升行动专项、中国科学院青年创新促进会和天津市自然科学基金的支持。[url=https://www.nature.com/articles/s41587-023-02050-w][color=#ff0000]论文链接[/color][/url][align=center][img]https://img1.17img.cn/17img/images/202401/uepic/ac641426-7515-499c-b780-d1c8c7b21f00.jpg[/img][/align][align=center]DAF-BEs碱基编辑器的设计及进化[/align][来源:天津工业生物技术研究所][align=right][/align]
基因芯片技术进展及应用 作者:刘炎 [关键词] 基因芯片;核酸探针序列;杂交 1 基因芯片概述 随着人类基因组计划( Human Genome Project)即全部核苷酸测序的即将完成,人类基因组研究的重心逐渐进入后基因组时代( Postgenome Era)向基因的功能及基因的多样性倾斜[1,2]。通过对个体在不同生长发育阶段或不同生理状态下大量基因表达的平行分析,研究相应基因在生物体内的功能,阐明不同层次多基因协同作用的机理,进而在人类重大疾病如癌症、心血管疾病的发病机理、诊断治疗、药物开发等方面的研究发挥巨大的作用。它将大大推动人类结构基因组及功能基因组的各项基因组研究计划。 基因芯片的工作原理与经典的核酸分子杂交方法(southern 、northern)是一致的,都是应用已知核酸序列作为探针与互补的靶核苷酸序列杂交,通过随后的信号检测进行定性与定量分析,基因芯片在一微小的基片(硅片、玻片、塑料片等)表面集成了大量的分子识别探针,能够在同一时间内平行分析大量的基因,进行大信息量的筛选与检测分析[3,4]。基因芯片主要技术流程包括:芯片的设计与制备;靶基因的标记;芯片杂交与杂交信号检测。 基因芯片的设计实际上是指芯片上核酸探针序列的选择以及排布,设计方法取决于其应用目的,目前的应用范围主要包括基因表达和转录图谱分析及靶序列中单碱基多态位点(single nucleotide polymorphism,SNP)或突变点的检测,表达型芯片的目的是在杂交实验中对多个不同状态样品(不同组织或不同发育阶段、不同药物刺激)中数千基因的表达差异进行定量检测,探针序列一般来自于已知基因的cDNA 或EST库,设计时序列的特异性应放在首要位置,以保证与待测目的基因的特异结合,对于同一目的基因可设计多个序列不相重复的探针,使最终的数据更为可靠。基因单碱基多态检测的芯片一般采用等长移位设计法[5],即按靶序列从头到尾依次取一定长度的互补的核苷酸序列形成一探针组合,这组探针是与靶序列完全匹配的野生型探针,然后对于每一野生型探针,将其中间位置的某一碱基分别用其它三种碱基替换,形成三种不同的单碱基变化的核苷酸探针,这种设计可以对某一段核酸序列所有可能的SNPs位点进行扫描。 芯片制备方法主要包括两种类型:(1)点样法:首先是探针库的制备, 根据基因芯片的分析目标从相关的基因数据库中选取特异的序列进行PCR扩增或直接人工合成寡核苷酸序列[6],然后通过计算机控制的三坐标工作平台用特殊的针头和微喷头分别把不同的探针溶液逐点分配在玻璃、尼龙以及其它固相基片表面的不同位点上,通过物理和化学的方法使之固定,该方法各技术环节均较成熟,且灵活性大,适合于研究单位根据需要自行制备点阵规模适中的基因芯片。(2)原位合成法[7~10]:该法是在玻璃等硬质表面上直接合成寡核苷酸探针阵列,目前应用的主要有光去保护并行合成法,压电打印合成法等,其关键是高空间分辨率的模板定位技术和高合成产率的DNA化学合成技术,适合制作大规模DNA探针芯片,实现高密度芯片的标准化和规模化生产。待分析样品的制备是基因芯片实验流程的一个重要环节, 靶基因在与芯片探针结合杂交之前必需进行分离、扩增及标记。标记方法根据样品来源、芯片类型和研究目的的不同而有所差异。通常是在待测样品的PCR扩增、逆转录或体外转录过程中实现对靶基因的标记。对于检测细胞内mRNA表达水平的芯片,一般需要从细胞和组织中提取RNA,进行逆转录,并加入偶联有标记物的dNTP,从而完成对靶基因的标记过程[11],对于阵列密度较小的芯片可以用同位素,所需仪器均为实验室常规使用设备,易于开展相关工作,但是在信号检测时,一些杂交信号强的点阵容易产生光晕,干扰周围信号的分析。高密度芯片的分析一般采用荧光素标记靶基因,通过适当内参的设置及对荧光信号强度的标化可对细胞内mRNA的表达进行定量检测。近年来运用的多色荧光标记技术可更直观地比较不同来源样品的基因表达差异,即把不同来源的靶基因用不同激发波长的荧光素标记,并使它们同时与基因芯片杂交,通过比较芯片上不同波长荧光的分布图获得不同样品间差异表达基因的图谱[12,13],常用的双色荧光试剂有Cy3- dNTP和Cy5- dNTP。对多态性和突变检测型基因芯片采用多色荧光技术可以大大提高芯片的准确性和检测范围,例如用不同的荧光素分别标记靶序列及单碱基失配的参考序列,使它们同时与芯片杂交,通过不同荧光强弱的比较得出靶序列中碱基失配的信息[14]。 基因芯片与靶基因的杂交过程与一般的分子杂交过程基本相同,杂交反应的条件要根据探针的长度、GC碱基含量及芯片的类型来优化,如用于基因表达检测,杂交的严格性较低,而用于突变检测的芯片的杂交温度高,杂交时间短,条件相对严格。如果是用同位素标记靶基因,其后的信号检测即是放射自显影,若用荧光标记,则需要一套荧光扫描及分析系统,对相应探针阵列上的荧光强度进行分析比较,从而得到待测样品的相应信息。由于基因芯片获取的信息量大,对于基因芯片杂交数据的分析、处理、查询、比较等需要一个标准的数据格式,目前,一个大型的基因芯片的数据库正在构建中,将各实验室获得的基因芯片的结果集中起来,以利于数据的交流及结果的评估与分析。
一、通过聚合酶链式反应(PCR)将靶基因亚克隆到质粒载体上(一)PCR引物设计的基本原则与PCR反应组分和条件1. PCR引物设计的基本原则(1)引物与靶基因间配对的碱基一般为15-20。(2)引物碱基尽可能随机分布,避免出现嘌呤、嘧啶堆积现象,引物G + C含量宜在45-55%左右。(3)引物内部不应形成二级结构,两个引物之间尤其在3’末端不应有互补链存在。(4)引物的碱基顺序不应与非扩增区域有同源性。可用计算机进行辅助检索分析。(5)引物3’末端一般以单个C或G结尾。 2. PCR反应组分和条件PCR反应体系一般选用50 μl体积,其中含有:10×Reaction buffer,5 μl2个引物,各12.5-25 pmol (终浓度各0.25-0.50 μmol/L)4种底物(dATP + dCTP + dGTP + dTTP),各200μmol/L模板DNA,100 ng左右Taq DNA聚合酶,2.5-3 U PCR反应条件一般为:(1)94℃,5分钟([font=Times New Ro
中国科技网讯 DNA序列中最轻微的变异也会影响深远,无论对研究还是医学应用,可靠识别这些序列都非常重要。据物理学家组织网近日报道,美国华盛顿大学和莱斯大学研究人员合作,开发出一种荧光DNA探测分子,能检查出一段目标DNA链中单个碱基的变化。而这些微小突变可能是造成某些疾病的根源,或耐抗生素细菌的原因。这一成果有助于诊断和治疗像癌症、肺结核这样的疾病。相关论文发表于7月28日的《自然·化学》杂志网站上。 不同的DNA序列为不同生物设定了独特的基因标记。现代基因组学研究表明,仅一个碱基对的变化都足以引发严重的生物后果,可能决定了一种疾病能否被治愈,也解释了疾病的突发或某些疾病对常规抗生素治疗无效的原因。论文领导作者、华盛顿大学电力工程和计算机科学与工程副教授乔治·塞利格说,比如造成肺结核的细菌有很强的耐药性,这种能力通常来自其基因序列中的少量突变。现在,人们已能预先查出这种突变。 “我们真正改进了以往的方法。”塞利格说,“新方法不需要任何复杂的反应或添加酶,就只用DNA。这意味着无论温度及其他环境变量怎样变化,该方法都是稳定的,所以很适合用于低资源设置中的诊断。” 这种探测分子经过专门设计,采用了新的编程机制,能与一个可疑的DNA序列结合,对其双螺旋链生成互补的DNA序列。把含有两种序列的分子在盐水试管中混合,如果两条链的碱基对都是完好的,它们自然地匹配在了一起,探测分子会发出荧光;如果不发光,则意味着上面有碱基对发生了突变。与以往技术不同的是,探测分子会检查目标DNA双螺旋的两条链是否发生了突变,而不是一条,这使检验更加全面具体。 此外,探测分子由许多寡核苷酸构成,克服了合成上的局限,可以探测更长的DNA序列中更详细的变异信息,达到200个碱基对,而现有探测突变的方法只能检查20个。 目前,研究人员与华盛顿大学商业化中心一起对该技术提出了专利申请,他们希望把这种技术和诊断试纸结合用于疾病测试。(常丽君) 《科技日报》(2013-8-7 二版)
在基因表达研究中,研究者比较注意选择合适的表达载体和宿主系统,而往往忽视基因本身是否与载体和宿主系统为最佳匹配这样一个实质性问题。基因的最佳化表达可以通过对基因的重新设计和合成来实现,如消除稀有密码子而利用最佳化密码子,二级结构最小化,调整GC含量等。以下就密码子最佳化、翻译终止效率和真核细胞中异源蛋白表达的问题加以说明。密码子最佳化(codon optimization)遗传密码有64种,但是绝大多数生物倾向于利用这些密码子中的一部分。那些被最频繁利用的称为最佳密码子(optimal codons),那些不被经常利用的称为稀有或利用率低的密码子(rare or low-usage codons)。实际上用做蛋白表达或生产的每种生物(包括大肠杆菌,酵母 ,哺乳动物细胞,Pichia,植物细胞和昆虫细胞)都表现出某种程度的密码子利用的差异或偏爱。大肠杆菌、酵母 、果蝇、灵长类等每种生物都有独特的8个密码子极少被利用。有趣的是,灵长类和酵母 有6个同样的利用率低的密码子。大肠杆菌、酵母 和果蝇中编码丰度高的蛋白质的基因明显避免低利用率的密码子。因此,重组蛋白的表达可能受密码子利用的影响(尤其在异源表达系统中)的事实并不很奇怪。你的基因利用的密码子可能不是你正在利用的蛋白生产系统进行高水平表达所偏爱的密码子,这种情况是可能的。利用偏爱密码子(preferred codons)并避免利用率低的或稀有的密码子可以合成基因,基因的这种重新设计叫密码子最佳化。在同源表达系统中,同较低水平表达的基因相比,较高表达的基因可能有很不同的密码子偏爱。通过对密码子利用的归类分析,人们可以真正预测任何基因在酵母 中的表达水平。在诸如Zea mays的其他生物中,大量高表达基因强烈偏爱以G或C结尾的密码子。而且,在Dictyostelium中,同低水平表达的基因比较,高表达基因有较大数目的偏爱密码子。在大肠杆菌中表达哺乳动物基因是不可预测和具有挑战的。例如直到最近才实现了人血红蛋白的过表达。为了达到血红蛋白的好的表达水平,Alpha-球蛋白cDNA不得不用大肠杆菌偏爱的密码子进行重新合成。在异源宿主中实现象血红蛋白这样复杂的蛋白质的过表达可能需要最佳化密码子,这些研究者为此提供了令人信服的资料。成簇的低利用率的密码子抑制了核糖体的运动,这是基因不能以合适水平表达的一个明显机制。核糖体翻译由九个密码子组成的信使(含几个低利用率密码子或全部为低利用率密码子)时的运动速度要比翻译不含低利用率密码子的同样长的信使的速度慢。即使低利用率密码子簇位于3'端,信使最后也会被核糖体”拥挤”而损害,核糖体又回到5'端。3'端低利用率密码子簇的抑制效应可以和全部信使都由低利用率密码子组成的抑制效应一样大。如果低利用率密码子簇位于5'端,其效应是起始核糖体数目的全面减少,导致蛋白合成中信使的低效率。散在分布的稀有密码子对翻译的效应还未很好地研究,但是有证据表明这种情况的确对翻译效率有负面效应。其他因素也可以影响蛋白表达,包括使mRNA去稳定的序列。重新设计合成基因可以去除或改变这些序列,导致高水平表达。消除稀有密码子、去除任何去稳定序列和利用最佳密码子的基因的重新设计都可能增加蛋白产量,使的蛋白生产更有效和经济。翻译终止效率蛋白表达水平受许多不同因素和过程影响。蛋白稳定性、mRNA稳定性和翻译效率在蛋白生产和积累中起主要作用。翻译过程分为起始、延伸和终止三个期。对于翻译的起始,原核mRNA需要5'端非翻译前导序列中有一段叫Shine-Dalgarno序列的特异核糖体结合序列。在真核细胞,有效的起始依赖于围绕在起始密码子ATG上下游的一段叫Kozak序列的序列。密码子利用或偏爱对延伸有深刻的影响。例如,如果mRNA有很多成簇的稀有密码子,这可能对核糖体的运动速度造成负面影响,大大减低了蛋白表达水平。翻译终止是蛋白生产必须的一步,但其对蛋白表达水平的影响还没有被研究清楚。但是最近的科学研究表明终止对蛋白表达水平有很大的影响。总的来说,更有效的翻译终止导致更好的蛋白表达。绝大多数生物都有偏爱的围绕终止密码子的序列框架。酵母 和哺乳动物偏爱的终止密码子分别是UAA和UGA。单子叶植物最常利用UGA,而昆虫和大肠杆菌倾向于用UAA。翻译终止效率可能受紧接着终止密码子的下游碱基和紧靠终止密码子的上游序列影响。在酵母 中通过改变围绕终止密码子的局部序列框架,翻译终止效率可能被减低几个100倍。对于UGA和UAA,紧接着终止密码子的下游碱基对有效终止的影响力大小次序为GU,AC;对于UAG是U、ACG。对于大肠杆菌,翻译终止效率可因终止密码子及临近的下游碱基的不同而显著不同,从80%(UAAU)到7%(UGAC)。对于UAAN和UAGN系列,终止密码子下游碱基对翻译的有效终止的影响力大小次序为UGA、C。UAG极少被大肠杆菌利用,相比UAAN和UGAN,UAG表现了有效的终止,但其后的碱基对有效终止的影响力为GU,AC。对于哺乳动物,偏爱的终止密码子为UGA,其后的碱基可以对in vivo翻译终止有8倍的影响(A、GC、U)。对于UAAN系列,in vivo终止效率可以有70倍的差别,UGAN系列为8倍。如果终止密码子附近序列没有最佳化,可能发生明显增加的翻译通读,因此减少了蛋白表达。例如,在兔网状细胞无细胞翻译系统里,UGAC的翻译通读可以高达10%,而第四个碱基如果为A,G或C,翻译通读为1%。总的来说,翻译起始框架、翻译终止序列框架和密码子利用应该仔细选择,以利于蛋白的最高水平表达。翻译终止序列框架能几倍地改变蛋白生产水平。真核细胞中的异源蛋白表达异源蛋白质在细菌中表达是目前使用的主要的蛋白生产系统。大肠杆菌一直是最经济的系统之一。然而为了生产需要特异修饰、胞外分泌或有特异折叠需要的蛋白质,其他表达系统也是需要的。真核细胞在表达原核来源的基因、真核基因的cDNA拷贝或其他无内含子的基因时可能表现很多特异问题。富含AT的基因在很多真核细胞中表达时会遭遇很剧烈的障碍。主要的真核信号序列如 加poly-A的位点、酵母 转录终止位点和真核mRNA去稳定序列都是富含AT的。内含子序列也趋向于富含AT,尽管他们有参与剪切过程的很特异的识别序列。虽然绝大多数原核基因没有剪切或聚腺苷过程,但这些真核过程需要的保守序列可能存在于原核基因中,因此当这些基因在真核细胞中表达时可能引起特异的问题。而且诸如哺乳动物和单子叶植物细胞的特异真核表达系统可能不能有效地表达无内含子的基因。 真核mRNA在离开细胞核进而在胞浆的核糖体上被翻译前需要特异的处理和修饰。这些过程包括去除内含子、5'端甲基化帽子形成和3'端加poly-A。内含子去除需要5'剪切位点、G75/G100U100A65AG65U保守序列、3'剪切位点、富含密啶NC66A100G100/G56保守序列和C72T98R77A100Y75保守序列。有效的加poly-A和mRNA剪切需要一个由两个部分组成的信号:加poly-A保守序列AAUAAA和在切割位点内的50个碱基的富含GT的序列。酵母 真核转录终止序列(几个不同的富含AT序列,如含TTTTTATA,TATATA,TACATA,TAGTAGTA的一个38bp区域)被研究的最清楚。这些结果来自对酵母 突变体CYCI mRNA的mRNA水平和相对长度的确定的实验。近期用in vivo质粒稳定性分析的研究结果证明:TATATA似乎和原始的38bp野生型区域一样有效地终止转录,而TAGATATATATGTAA和TACATA效率差些,TTTTTTTATA几乎没有效率。所有这些序列在反方向时没有终止转录功能。不幸的是几乎没有其他真核表达系统转录终止序列方面的信息。内含子对几个哺乳动物基因的正常表达是必需的,包括Beta-球蛋白、SV40 late mRNA和二氢叶酸还原酶基因。单子叶植物细胞充分表达乙醇脱氢酶的cDNA拷贝、报告基因氯霉素乙酰转移酶、Beta葡萄糖苷酸酶和其他缺乏内含子的基因时也依赖内含子。转录区域内引入内含子可以通过未确定的转录后机制增强表达。(免疫球蛋白基因)内含子可能也包含转录增强子,因此通过转录机制增强表达。 总的来讲,如果存在某些DNA序列,真核异源蛋白表达可能是个难题。为避免剧烈的表达减少,需要对基因进行扫描,确认是否含上述提及的富含AT的序列。而且,在几个真核系统表达无内含子基因可能需要引入内含子以实现外源蛋白的充分表达。
生物科学正迅速地演变为一门信息科学。最明显的一个例子就是目前正在进行的HGP(human genome project),最终要搞清人类全部基因组的30亿左右碱基对的序列。除了人的遗传信息以外,还有其它生物尤其是模式生物(model organism)已经或正在被大规模测序,如大肠杆菌、啤酒酵母、秀丽隐杆线虫以及中国和日本科学家攻关的水稻基因组计划。但单纯知晓生物基因组序列一级结构还远远不够,还必须了解其中基因是怎样组织起来的,每个基因的功能是什么,又是怎样随发育调控和微环境因素的影响而在特定的时空域中展开其表达谱的,即我们正由结构基因组时代迈入功能基因组时代。随着这个功能基因组学问题的提出(后基因组时代,蛋白组学),涌现出许多功能强大的研究方法和研究工具,最突出的就是细胞蛋白质二维凝胶电泳(2-D-gel)(及相应的质谱法测蛋白分子量)和生物芯片(Biochip)技术。一、什么是基因芯片生物芯片,简单地说就是在一块指甲大小(1cm3)的有多聚赖氨酸包被的硅片上或其它固相支持物(如玻璃片、硅片、聚丙烯膜、硝酸纤维素膜、尼龙膜等,但需经特殊处理。作原位合成的支持物在聚合反应前要先使其表面衍生出羟基或氨基(视所要固定的分子为核酸或寡肽而定)并与保护基建立共价连接;作点样用的支持物为使其表面带上正电荷以吸附带负电荷的探针分子,通常需包被以氨基硅烷或多聚赖氨酸等)将生物分子探针(寡核苷酸片段或基因片段)以大规模阵列的形式排布,形成可与目的分子(如基因)相互作用,交行反应的固相表面,在激光的顺序激发下标记荧光根据实际反应情况分别呈现不同的荧光发射谱征,CCD相机或激光共聚焦显微镜根据其波长及波幅特征收集信号,作出比较和检测,从而迅速得出所要的信息。生物芯片包括基因芯片、蛋白质芯片、组织芯片。而基因芯片中,最成功的是DNA芯片,即将无数预先设计好的寡核苷酸或cDNA在芯片上做成点阵,与样品中同源核酸分子杂交的芯片。基因芯片的基本原理同芯片技术中杂交测序(sequencing by hybridization,SBH)。即任何线状的单链DNA或RNA序列均可被分解为一个序列固定、错落而重叠的寡核苷酸,又称亚序列(subsequence)。例如可把寡核苷酸序列TTAGCTCATATG分解成5个8nt亚序列: (1) CTCATATG (2) GCTCATAT (3) AGCTCATA (4) TAGCTCAT (5) TTAGCTCA这5个亚序列依次错开一个碱基而重叠7个碱基。亚序列中A、T、C、G4个碱基自由组合而形成的所有可能的序列共有65536种。假如只考虑完全互补的杂交,那么48个8nt亚序列探针中,仅有上述5个能同靶DNA杂交。可以用人工合成的已知序列的所有可能的n体寡核苷酸探针与一个未知的荧光标记DNA/RNA序列杂交,通过对杂交荧光信号检测,检出所有能与靶DNA杂交的寡核苷酸,从而推出靶DNA中的所有8nt亚序列,最后由计算机对大量荧光信号的谱型(pattern)数据进行分析,重构靶DNA 的互补寡核苷酸序列。二、芯片类型一般基因芯片按其材质和功能,基本可分为以下几类:(一)元件型微阵列芯片1 .生物电子芯片2 .凝胶元件微阵列芯片3 .药物控释芯片(二) 通道型微阵列芯片1.毛细管电泳芯片2 .PCR扩增芯片3 .集成DNA分析芯片4 .毛细管电层析芯片(三)生物传感芯片1 .光学纤维阵列芯片2 .白光干涉谱传感芯片小鼠基因表达谱芯片(MGEC)附:目前国内基因芯片常见品种(上海博星公司)http://www.biomart.cn/upload/asset/2008/08/01/1217591301.gifhttp://www.biomart.cn/upload/asset/2008/08/01/1217591302.gifhttp://www.biomart.cn/upload/asset/2008/08/01/1217591303.gif
创作者研习社4-仪器操作”视频拍摄教学(杨宇晨 仪器信息网 视频剪辑师)[color=#cc0000][b]杨老师曾任CCTV1专业摄影师[/b][/color]
最近,美国洛斯阿拉莫斯国家实验室(LANL)的一个遗传学小组和一国际财团联合提出了一套旨在阐明可公开获取的基因测序数据信息的质量标准。新标准最终可使遗传研究人员开发出更有效的疫苗,或有助于公共健康部门或安全人员更迅速地应对潜在的公共卫生突发事件。 在10月9日的《科学》杂志上,LANL遗传学家帕特里克钱恩和他的同事提出了6个基因组测序数据标签,可将基因测序数据按其完整性、准确性以及由此带来的可靠性进行归类。这些标签可在公共数据库中获取,而目前使用的标签仅为两个。此项成果的重要性在于,研究人员必须每天使用这样的数据,以对未知遗传数据和已知生物体的遗传数据进行相互参照,而有了这样的新的分类标准,数据的获取与对比工作的效率将大大提高。 每个生物体的细胞内都有DNA,由4个分子构建模块(或称碱基对)组成,碱基对排成特定序列时就可构成基因。这些基因序列可包含对生物体有益或有害的遗传指令。基因组研究人员编目了数以千计的基因数据,并将其放在公众数据库中以供其他研究者使用。然而,由于基因数据的复杂性,公共数据库中的遗传信息范围从粗略到精致一概都有。过去,这些基因数据常被归类为“草图”和“成品”两大类,给基因数据的准确性留下了太多的不确定性。 钱恩表示,在过去几年里,基因测序技术已取得重大进步,公众可获得的基因数据已呈爆炸性增长,每天产生的碱基对序列数据量要比过去几年产生的数据量还要多几十亿次。不同的测序技术具有不同的精确度。一个序列中的高度不确定性可能会引导研究人员走向一条耗时长达一年甚至数年的错误道路。因此,有必要建立一个标准,为研究人员提供对遗传测序数据质量的明确评估。 钱恩联合了大大小小的数个基因组测序中心,如美国能源部联合基因组研究所、桑格研究所、人类微生物群系项目Jumpstart联盟测序中心、密歇根州立大学以及安大略省癌症研究所等,共同提议将现有的测序数据分类从两大类充实为6大类。这6个标准涵盖了从代表公众提交最低要求的“标准草图序列”到代表最高标准的“完成序列”,而“完成序列”的验收标准是每10万个碱基对中最多只能包含一个错误。 LANL基因科学小组负责人、联合基因组研究所LANL研究中心主任克里斯戴特表示,该项研究的目的是为了让所有主要的基因组中心和基因组研究小组都能用上符合其需要的分类基因组测序数据。而为了尽可能保证基()因组序列的完整性,一些较小的研究中心也可采用这个分类等级来建立和提交其研究成果,以帮助其他科学家了解既已完成的工作。(科学网)
据8月28日的《科学》杂志报道说,蚕虫驯养已经有1万多年历史了。蚕为人类提供了宝贵的丝绸和蛋白。但是,现在对蚕基因进行序列测试还为人们提供了一张有关这些随时会为我们提供如此多宝贵物质的昆虫的基因变异图。由西南大学、深圳华大基因带领的国际研究团队为29种家蚕和11种野蚕世系的基因组成功地进行了测序并找到了这些世系之间的差别。共获得了40个家蚕突变品系和中国野桑蚕的全基因组序列,共测632.5亿对碱基序列,覆盖了99.8%的基因组区域,是多细胞真核生物大规模重测序研究的首次报道;绘制完成了世界上第一张基因组水平上的蚕类单碱基遗传变异图谱,这是世界上首次报道的昆虫基因组变异图。科学家还发现了驯化对家蚕生物学影响的基因组印记,从全基因组水平上揭示了家蚕的起源进化。 研究发现,家蚕很明显地在基因上与其野生对应物不同,但即使在各家蚕世系之间,它们仍然维持着大量的变异性。这提示,家蚕只经历了一次牵涉有大量个体的单一且短暂的驯养过程,并在此后在家蚕与野蚕种群之间很少有基因流动。研究人员还能够识别出特别的能够增进丝的生产、蚕虫的繁殖和生长的基因(这些基因很可能是被人类挑选出的)。他们甚至还寻找到了在驯养过程中由蚕虫所获取的行为特征,例如极端的拥挤和容忍人的靠近和操作,以及它们在驯养过程中所丧失的如逃逸及躲避掠食者和疾病等的特征。(
科技日报2007年12月20日讯 人类基因组测序工作的最终完成,花费了全球6个国家的顶尖科学家们10年多的时间和精力以及30亿美元的财力。虽然不断有科学家报道他们关于治病基因的发现成果,但含有30亿碱基对的人类基因组数量太庞大,基因疗法距离实际运用还需要很长时间的等待。几十年来,不断有科学家认为,基因组中有很多DNA(脱氧核糖核酸)并没有特殊功能,甚至有科学家将这些DNA称为“垃圾DNA”,这些垃圾DNA从人体去除后也不会对人类基本活动带来严重后果,但是反对者却认为每个DNA都有自己特定的功能,关于垃圾DNA是否存在一直存在争论。[em0716] [em0715]
无需进行文库制备,所用DNA样本比标准方法更少2012年12月13日 来源: 中国科技网 作者: 陈丹 中国科技网讯 据物理学家组织网12月12日(北京时间)报道,英国研究人员简化了基因组测序的标准流程,首次无需进行文库制备便完成了DNA(脱氧核糖核酸)单分子测序,而且新方法只要很少量的DNA就能获得序列数据,用量可低至不到1纳克(10亿分之一克),仅为常规测序方法的500分之一到600分之一。 文库制备是指从测序前基因组样本中提取不同长度的DNA片段,这一过程不仅费力、费时,还会浪费DNA,而新技术能极大地减少DNA的损耗,并缩短测序时间。 该研究论文的第一作者、英国威康信托基金会桑格研究所的保罗·库普兰说:“我们用这种方法对病毒和细菌的基因组测序后发现,即使在相对较低的水平,我们也能够确定所检测的是何种有机物,不论样本中是否存在特定的基因或质粒(这对于确定抗生素耐药性很重要),或者其他信息,如对特定DNA碱基的修改等。”他表示,一旦技术得到优化,将在快速、高效地识别医院和其他医疗场所中的细菌和病毒方面具有很大的应用潜力。 研究小组利用第三代单分子测序系统PacBio RS演示了这种简化的直接测序方法。他们仅仅用800皮克(千分之一纳克)DNA来分析一个生物体的基因组,尽管测序仪只读取了基因组的70个序列片段,相对于常规测序方法获得的数据来说不过是很小的一部分,但这些信息足以让研究人员确定他们所检测的生物体的品种。 这项技术也使得科学家能够对此前无法识别的宏基因组(也称微生物环境基因组)样本中的生物体进行确认。“为微生物测序,首先需要能够在实验室中培养它们。”论文的主要作者、英国巴布拉汉研究所的塔米尔·钱德拉说,“这不仅耗费时间,而且有时候微生物不生长,为它们的基因组测序极其困难。”他表示,新方法可以直接对微生物测序,短时间内便可确定其“身份”。 论文的另一主要作者、威康信托基金会桑格研究所的哈罗德·斯维尔德洛说:“我们的技术可以在对所测序列没有任何先验知识、没有特定微生物试剂的条件下,在很短的时间内操作,这是一种很有前途的替代手段,可应用于控制感染等临床需要。”(记者陈丹) 总编辑圈点 长久以来,基因测序等围绕基因科学所展开的研究,都被人们贴上了从本源上解开人体生命奥秘、彻底解除遗传疾病威胁等殷切的标签。多国为提高社会健康水平,都开展了解码国民DNA的活动,有些甚至覆盖全基因组。然而,面对由30亿个碱基对构成的人类基因组,精确测序注定将是一场浩大而又漫长的工程。如何能快速、准确地将海量DNA数据转化为有帮助的实用信息,已经成为该领域科学家们面临的重大挑战之一。因而我们说,英国科学家此番取得的突破,不管是从整个学科研究的方法论层面,还是从临床应用的角度,都提高了基因研究服务于人类的速度。 《科技日报》(2012-12-13 一版)
科学家发现海洋巨型病毒拥有73万个碱基对 北京时间10月29日消息,据物理学家组织网报道,英属哥伦比亚大学(UBC)已经发现世界上最大、最复杂的海洋病毒,Cafeteria roenbergensis病毒主要感染那些吃海洋生态系统中非常重要和分布广泛的浮游生物的掠食者。 这种病毒的基因组比一些细胞生物的基因组还大,它的遗传复杂性使科学家感到疑惑,不知道该把它归为“无生命”生物,还是“有生命”生物行列。海洋微生物学和环境病毒学专家、这项研究的第一论文作者和英属哥伦比亚大学教授柯蒂斯·苏特勒说:“我们一般认为病毒都很小,是简单生物体,只有少量基因。然而我们在这种病毒里发现的大量遗传机制,只能在有生命的细胞生物体里找到,它们需要很多基因才能产生DNA、RNA、蛋白质和糖。” 该研究成果发表在本周的《美国国家科学院院刊》上。一般情况下,病毒在活宿主细胞外无法自我复制,它们需要利用宿主提供的蛋白质进行复制,自我复制形式是区分“无生命”和“有生命”生物的分界线。然而最新发现的这种巨型病毒却对上述归类标准发起了挑战,它们虽然仍需要一个细胞进行复制,但它们是在自己的基因组里进行编码的。 20世纪90年代初,有人在德克萨斯州沿海水域发现这种巨型海洋病毒。苏特勒和他的科研组确定该病毒的基因组含有大约73万个碱基对。这使Cafeteria roenbergensis病毒成为目前已知的世界最大海洋病毒和第二大病毒,排名仅次于淡水病毒——多噬棘阿米巴模仿病毒,后者拥有120万个碱基对。Cafeteria roenbergensis病毒还感染在海洋食物链中处于重要地位的浮游动物。 苏特勒说:“尽管这些海洋浮游生物的掠食行为在海洋和淡水系统的碳转移及营养循环过程中起着重要作用,但是我们对该病毒在这个系统里所扮演的角色几乎一无所知。毫无疑问,这种病毒可能还是一大组未知但是具有生态重要性的海洋巨型病毒的代表。”

[img=,351,220]http://ng1.17img.cn/bbsfiles/images/2018/02/201802061354219789_7475_1667_3.jpg!w351x220.jpg[/img]ABI 3130 / ABI 3130XL测序仪的主要性能特点:? 3130xl 采用 16 道毛细管,3130 系统采用 4道毛细管? 24 小时无人监控操作? 仪器设置更方便更简单? 新型自动灌胶系统进行灌胶? 检测池加热器改进了温度控制? 通过 96 孔和 384 孔板自动进样? 3130 POP-7?、POP-6? 和 POP-4? 分离胶? 多色荧光检测? 一种分离胶一种毛细管用于多种应用系统组成美国应用生物系统公司 3130 和 3130xl 基因分析仪由以下组件组成:? 毛细管电泳仪? 计算机工作站:用于仪器控制和数据分析? 软件:用于仪器控制、数据收集和样品文件自动分析? 分析软件包括:- 用于碱基识别的序列分析软件- 用于微卫星、SNP、AFLP 和 LOH 分析的 GeneMapper?/GeneMapper? ID 软件毛细管束提供预组装的 4 根或 16 根内壁无涂层毛细管。毛细管束有多种长度,为多种应用和分析方法提供支持。这些毛细管束在 3130xl 系统上的特定使用寿命为 100 次,在 3130 系统上为 150 次。它们与业界标准的 96 孔和 384 孔板配合使用。分离胶3130 POP-7?、POP-6? 和 POP-4? 三种分离胶(性能优化分离胶)均可以在美国应用生物系统公司的 3130 和 3130xl 基因分析仪上用作分离介质。在每次分析之前,毛细管自动使用新胶进行补充,这些新胶动态覆盖毛细管壁,以消除电渗流。试剂美国应用生物系统公司提供下列用于 3130 系列系统的试剂:? 基因序列分析试剂盒- BigDye? Terminator 荧光标记终止物试剂盒- dGTP BigDye? Terminator 荧光标记终止物试剂盒- BigDye? Primer Cycle 荧光标记引物试剂盒 Sequencing Ready Reaction 试剂盒,M13Rev/-21 M13- dRhodamine Dye Terminator 荧光标记终止物试剂盒? 片段分析试剂盒- Linkage Mapping Set Version 2.5 疾病基因定位克隆试剂盒- GeneScan?-400 HD 荧光标记分子量内标- GeneScan?-500 荧光标记分子量内标- GeneScan?-120 Liz? 荧光标记分子量内标- 其它专用试剂盒样品要求3130 系列系统可以分析由各种样品制备方法制备的多种类型的模板。可以从 96 孔和 384 孔微孔板中自动进样。? 激光 氩离子多波长单模激光器,主要激发波长为:488 和 514.5 nm。? 检测光学系统 美国应用生物系统公司 3130 系统基因分析仪使用专利的激发光和荧光检测光路 来增强荧光信号强度的均一性。这些检测光学系统实时检测来自各泳道毛细管的低本底、全波长光谱数据。毛细管的外径 (od)、内径 (id) 和间距都已进行优化,将因折射引起的信号丢失降至最低。? 电泳电压 最高 可达20 kV? 操作温度范围 18°C-65°C?计算机最低要求- 硬件:Pentium? IV 处理器,2.00 GHz 处理器- 操作系统:Windows XP? Professional Edition- 安装内存 (RAM):1 GB- 硬盘容量:两个 36 GB 硬盘驱动器- 外围设备:CD/RW光盘刻录?运行环境- 温度: 20°C-25°C- 仪器运行时,室温应在 ±2° 范围内波动。- 湿度:40%-60%(无冷凝)? 主电源电压- 220V ±5%- 50-60 Hz ±10%? 电流 最大:15 安培?最大功率 2,000 瓦特(大约)仪器大小尺寸电泳设备:?宽度(门关时):74 cm 宽度(门开时):148.6 cm(左右门同时打开}? 厚度:54.8 cm? 高度:81 cm? 重量:130 kg(大约)
作为生命遗传的基本单位,基因正变得愈来愈为大众所熟知。由32亿个碱基对组成的人类基因组,是一部蕴藏着生命奥秘的天书。始于1990年的国际人类基因组计划,由6国科学家共同完成,花费27亿美金,在2000年6月宣布完成。 时至今日,基因组测序的费用已经大大下降。中科院北京基因组研究所陈科博士在接受《中国科学报》记者采访时给出了下面的数据:“基因组测序费用从27亿美元到1万元人民币,时间成本从13年变成13天,人力成本从当时上千人的六国科学家,到今天的3到5人就可以搞定。总体投入小到对大部分人来说都是可用得上的。” 安吉丽娜·朱莉因为基因检测确信自己未来会罹患乳腺癌而进行了预防性切除手术,随着基因检测费用的降低,是否意味着每个普通人都可以享受到好莱坞女星般的待遇? 平价到仅一餐的价格 电视从业者田晓岩联系到《中国科学报》记者,说她最近发现了一款只要299元的基因检测产品。可以检测肥胖基因以及一些营养需求情况。 浏览该公司网站不难发现,该产品的互联网属性非常明显。目前仅有299元套餐一项产品,新用户注册立减50元,也就是说只要249元人民币、相当于一餐饭的价格,就可以体验一次“高大上”的生命解码。 付费之后,不久便可以收到该公司寄来的DNA采集包。打开一个蓝白色相间的大信封,里面装有一份说明书、两份一次性DNA 采集包——其中包含两套专用植绒棉棒、两个采样管、一份酒精消毒棉片、一份DNA 采样寄回袋,还有一张写着寄回地址的快递单。 用户只需按照说明书指示,用植绒棉棒在口腔内两侧皮肤上下刮拭15次以上,将拭子头部放进采集管即可。完成基因采样动作之后,直接用快递单把采样包寄回,两周后就可以取得自己的基因分析报告了。 田晓岩告诉记者实际上只用了大概一周的时间自己就收到了报告,用户体验也非常不错:“很方便,说明非常详细,完成整个操作只需要10分钟的时间。甚至快递单都填写好了、而且是到付。”最终她收到的报告显示自己存在新陈代谢过慢的风向,以及需要加强补充维生素E和叶酸。 “报告的内容有点简单,而且其中提供了一个人群比较数据,比较好奇这个比较是怎么来的。是基于自己的数据库吗?还是说有别的数据支撑。”田晓岩提出了自己的困惑。虽然只是抱着体验的心态,但是跟自己健康相关的东西,多少还是会有些介意。
如何查找特定基因序列的心得与大家共享,下面以小鼠EDNRB基因为例子说明:1. 查找一个基因的启动子之前,最好是要对这个基因做一个初步的了解,了解方法为:http://www.ncbi.nlm.nih.gov/sites/entrez/上面查找ednrb http://img.dxycdn.com/trademd/upload/userfiles/image/2014/01/A1388735412_small.jpg 注意:在research下面选择 Gene 在for后面输入ednrb2. 这个是查找结果,由于很多哺乳类动物都有该基因,查找结果显示了很多五种的ednrb基因,我们选择其中的Mus musculus 这个是我们要找的小鼠种类,请大家正确选择自己目标基因的动物种类,Rattus norvegicus是大鼠的学名,Mus musculus是小鼠的学名,由于我的目标是小数的ednrb基因,故选择Mus musculus。 http://img.dxycdn.com/trademd/upload/userfiles/image/2014/01/A1388735413_small.jpg 3: 对Official Symbol Ednrb and Name: endothelin receptor type B Other Aliases: AU022549, ET-B, ETB, ETb, Sox10m1, sOther Designations: piebaldChromosome: 14; Location: 14 51.0 cMAnnotation: Chromosome 14, NC_000080.5 (104213843..104242913, complement)GeneID: 13618解释:Official Symbol Ednrb and Name 这个是官方对这个基因的全称,也就是内皮素受体B 这个是该基因编码的蛋白质名称Other Aliases 该基因的别名Chromosome: 14; Location: 14 51.0 cM 说明该基因位于第14条染色体上面Annotation: Chromosome 14, NC_000080.5 (104213843..104242913, complement)GeneID: 13618这个说明基因的碱基分布是104213843到104242913.4. 该基因的具体信息 http://img.dxycdn.com/trademd/upload/userfiles/image/2014/01/A1388735414_small.jpg 5. 下面这个图片是上个图片的下半部分 http://img.dxycdn.com/trademd/upload/userfiles/image/2014/01/A1388735415_small.jpg 6. 以上部分是找到对该基因的基本信息,在最下面Reference assembly (C57BL/6J)上面找到基因具体序列的链接,点击其中的GenBank, ednrb基因碱基的排列就出来了,这里说明 一下,genbank上面给的序列都是与相应mRNA互补的序列,也就是模板链的序列,也就是-(minus strand) 上面的C57BL/6J是得出该序列的小鼠品系,如果大家要做相关实验的话 最好是选择该品系的小鼠或者相应的细胞。 http://img.dxycdn.com/trademd/upload/userfiles/image/2014/01/A1388735416_small.jpg 7. http://www.ncbi.nlm.nih.gov/entrez/viewer.fcgi?val=NC_000080.5&from=104213843&to=104242913&strand=2&dopt=gb 上面是该基因的信息和碱基序列链接,上面有碱基的序列,mRNA 的序列。
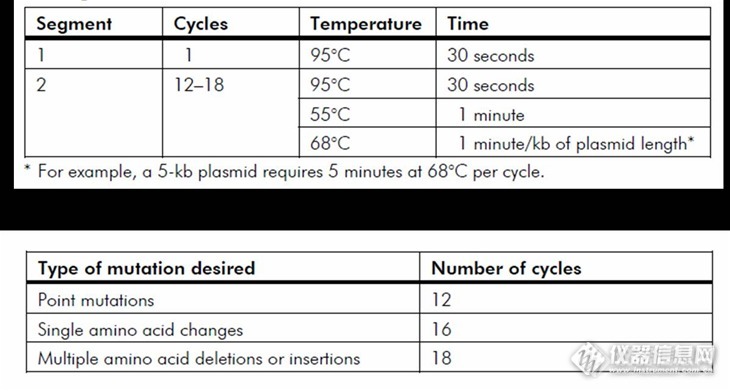
基因定点突变,顾名思义,就是把目的基因上的一个碱基有目的的替换为另一个碱基。体外定点突变时目前分子生物学领域中一种快速有效地手段。产生定点突变操作及所需仪器简单,随着技术的发展,方法也越来越快捷简便。定点突变除了可以生成点突变,多点突变外,还可以人为产生碱基删除和添加等。体外基因定点突变是实验室中优化改造基因,研究基因功能的常用方法之一。通过改造基因序列,可以对相应氨基酸序列进行改变,进而影响蛋白质的结构和功能,探讨突变氨基酸位点在蛋白结构和功能中所起的作用。在我多年的实验中,体外基因定点突变是其中一项重要的内容。在这里,我与大家分享一下我的实验方法和心得体会。多年来,我所采用的基因定点突变的方法主要有三种,这可能也包括了目前所有的定点突变的方法。基因定点突变使用的主要仪器就是PCR扩增仪(biometra),而这是每一个分子生物学实验室的必需品,也就是大家吃饭的家伙。下面从繁到简对我所采用的突变方法进行一下简单描述。http://ng1.17img.cn/bbsfiles/images/2012/12/201212032022_409068_1306303_3.jpg我所使用过的定点突变方法简单的说就是三步,两步和一步PCR法。三步PCR法是我早期在实验室使用的定点突变方法。利用三步PCR法构建一个点突变,需要4条PCR引物:L1,L2,R1和R2。L1和R1是目的基因5‘和3‘端的引物,分别包含起始和终止密码子。突变点设计在引物L2和R2的正中间位置,L2和R2是完全互补的两条PCR引物,长度一般在28-40 bp之间,推荐长度31-35 bp,这样可以保证突变的两边有效搭在一起。http://ng1.17img.cn/bbsfiles/images/2012/12/201212032026_409072_1306303_3.jpg三轮PCR均采用常规PCR反应条件,第一轮分别以L1和R2,L2和R1扩增获得目的片段L和R。第二轮PCR,不需要引物,仅加入模板L和R各100 nmol,进行约20 轮的PCR反应,获得产物m1。第三轮PCR以m1为模板,L1和R1为引物进行PCR扩增。最终得到目的产物m。得到的PCR产物进行克隆转化得到进一步扩增,便获得了突变的目的基因。在随后的实验中,我将三步PCR法简化为两步法PCR。两步法PCR和三步法PCR一样,需要引物四条,引物设计也与三步法相同,只是在第一轮PCR获得产物L和R之后不再单独进行第二轮PCR,而直接在PCR体系中加入等mol的产物L和R
一直以来都想把自己在基因克隆方面的心得写出来,让更多的刚刚进入生命科学领域的人受益,因为自己刚开始做克隆时也遇到过各种问题,经过较长时间的总结和实践,我的题组现在的基因克隆都是一次到位的,基本不需要重复做。其实我只是一个只有几十万科研经费的小青椒,不过我对科研非常热爱,我喜欢买实验用的各种酶啊,好用的耗材之类的东东超过我对自己的衣服鞋子的热爱,所以我看起来穿的及其普通,可是我的实验花费有点奢华,呵呵,可能像我这样的人不多吧,哈哈,反正无所谓开心就好。下面言归正传(1)是酶切位点的选择。我的实验室有Takara、Promega以及NEB三种公司的常用的酶,这极大的丰富了我们的选择,所以在设计PCR产物的酶切位点之前首先要看看哪两个酶之间是可以进行同时酶切的。因为这三家公司的双酶切表的组合完全不同,最佳的方案是我们能够按照需要去选择合适的酶。有人说这得花很多钱吧,其实不然,Takara几乎每年9月都有一次促销活动,在他们七折的时候我一下买了三千块钱的酶,这一年来有用之不尽的感觉。Promega的酶也非常好用,而且长期五折,我也是常用的酶买了一批放在实验室里。至于NEB的实在是有一点贵的,我一般不批量买了,在NEB买的一般都是不常用的酶,比如FseI、AscI等等。酶的选择是实验成功的关键吆。(2)PCR引物的设计这一点我不想多说,虽然有很多的攻略里面讲到了PCR引物设计的原则等等,大家设计的时候要参考各种原则,我认为不然,因为做过实验的战友都清楚,有的时候很多PCR引物的选择是没有选择的,比如我要扩增一个完整的基因的ORF框,那么它的起始密码子,终止密码子部分都要克隆出来的,不能多也不能少一个碱基,即使起始部位或者终止部位的AT含量很高,高到你难以忍受,那怎么办呢,基本我们没有选择,如果实在是没办法的条件下,只能在PCR引物的5端加入人为设计的碱基而把引物的扩增部分后移或前移来避开难以扩增的部位,我不知道说清楚没有,如果引物序列OK,可以忽略上句话。所以大多数情况下引物我们是没得选择的,那么我们只能从PCR扩增条件上下功夫。(3)PCR扩增对于PCR扩增其实不同的基因可能策略不同,我来说几点相同的。首先很多新手会忽略引物的浓度问题,我在最开始做PCR的时候因为当时的基因非常容易扩增,所以其实我的条件并不是最佳的,但当时把基因扩出来了我也没有在意,直到有一天我需要在基因的5端加入3个HA标签,这样的PCR引物长度差异很大,一支引物100多bp,一支引物只有30bp,于是当我还有以前的条件时我扩不出任何的基因。当时扩了几次都不成功,各种温度都试过了也不成。于是我静下心来,把PCR的实验条件进行了全方位优化,在PCR反应体系中,把引物调整到各种浓度的,把模板调整到各种浓度的,有的加Mg2+,有的加BSA,还使用梯度PCR的条件,试了各种扩增温度的,结果让我很开心,最后我的基因被扩增出来了,而且好亮好亮的那种。记得当时自己高兴地跳了起来。也许这就是科研的魅力吧!在这次试验中我找到了最佳的PCR条件,这是三年前的事了,这个条件让我在三年中屡试不爽,几十个基因的扩增从未失手过。其实体系很简单,50ul体系中buffer 5ul、Mg2+ 1mM、dNTP 0.2mM、引物每支1ul(配成10umol/L浓度)、PCR酶一般是0.5ul、其余部分用水补平,混匀,离心一下,进行PCR扩增。其中引物从公司拿到干粉后我一般用水溶解至100umol/L浓度保存,吸取少量稀释十倍后用于PCR反应,这个浓度是最佳的。所以PCR体系中引物并不是越多越好,同样的模板的量也很关键,一般我都在10ng-100ng之间,太少或太多都会抑制PCR反应。当然,不同的基因其退火温度差异较大,建议第一次做直接做梯度PCR,设置的温度范围宽些,总会有扩出来的。反正把反应体系加好,把温度控制好应该就万事大吉了,如果这样仍然扩不出来,那就直接调整DNA模版的量吧,其他的因素应该不是原因(当然得保证引物,以及酶的质量得前提下)。(4)PCR产物的酶切,这是最简单的一步,一般我都是酶切过夜的。因为我认为PCR产物切得尽可能的充分对克隆很重要,毕竟保护性碱基只有几个。(5)质粒的酶切。虽然质粒的酶切很简单但是却很讲究,决定着克隆的成败。质粒提取我一般都用试剂盒,天根的很便宜了,现在好像一盒已经六折,一盒有200个,可以用很久。质粒提取完毕后我会用紫外分光光度法对质粒进行定量测定,根据A260的值计算出质粒的量,然后再进行酶切,一般酶切体系60ul,60ul体系中我只切总量1ug的质粒,一次切两管,酶切过夜后切胶回收或者不切胶直接回收,这取决于两个限制性内切酶之间的距离,十几bp以内我就直接回收了,如果偏大就要切胶回收。(6)连接 连接我采用的是Promega公司的T4 DNA连接酶,它的特点是22度连接三小时以上几款,这样我就可以在上午把质粒片段以及PCR片段回收后马上做连接,连接一个白天,到下午可以做转化了,涂板,过夜培养第二天早上看结果。然后挑克隆(一般我一个基因就挑四个克隆足已)培养一白天,下午稍晚些提质粒,然后马上酶切鉴定,一般酶切鉴定体系中我都做20ul体系,酶用0.5微升就够了(呵呵,该省的就省点吧),酶切一个小时跑胶就可以知道克隆是否成功了。这样从PCR到克隆鉴定完毕,一共三天。不过从我带学生的经验来看,从一个懵懵懂懂的新手到成功掌握该技术快则半个月,多则一个月,引人而异。各位也试试看吧!以上为本人在基因克隆方面的一家之言,难免有疏漏或过于肯定之处,感谢各位战友多提宝贵意见,多多交流,以后我会陆续贴出各种技术的实验心得,欢迎大家相互交流!
美国加州的山景城是“硅谷”的重要组成部分。现在,一个与硅芯片相关的潜力大产业正在这里兴起,那就是基因组测序技术产业。这个产业的发展是随着多家大公司的激烈竞争开始的。不过,一家名为“整合基因”(Complete Genomics,CG)的公司不像别的公司一样研发和销售测序仪器,而是为科学家提供外包的测序服务,更绝的是,在这家公司里做测序的,并不是研究人员,而是一排排的机器人。近日,《新科学家》杂志探秘了这家充满科幻意味的公司。前台都是“机器人”走进CG公司,连前台都由计算机终端出任。它会主动向来客问好,询问姓名、身份和来访意图。旁边连接的一台打印机则自动打出访客挂牌。与此同时,一份电子邮件已经发送到内部接应人员的电脑上。这家公司的生产线更像科幻电影里的实验室,昏暗蓝色的房间里到处都是高级仪器,室内温度保持在28℃和相对较高的湿度,几名穿着实验服,带着发罩的工作人员在监视着电脑屏幕,查看着机器人的运作状态。这儿已经成为了世界上最大的人类基因组测序工厂。只是在这里工作的不是人类,而是机器人。在一个大约只有半个网球场大的房间里,“坐着”16台机器人,不间断地进行着人类基因组测序的工作。去年,它们完成800个人的DNA测序工作其中三分之一是后半年做出来的。到了今年,它们已经可以每个月生产出400个人的基因了。CG公司只是目前迅速形成产业的诸多基因组测序公司中的一家,但是它十分独特。公司市场总监图柯特(Jennifer Turcotte)对《新科学家》杂志解释说,通常而言,DNA测序是在一个密封的机器里进行的,但在这家公司的实验室里,机器人却是在一个开放暴露的环境下做基因组测序,这是为了便于维修。实验室特定的温度和湿度是为了符合测序中出现的生化反应,微弱的蓝光是为了避免荧光探测剂在探测基因代码符号时受到其他频率光波的破坏。这儿所进行的基因组测序,已是目前最新的第三代基因组测序技术,称为“DNA纳米球测序技术”。这种新方法是将DNA链放置在一小块硅芯片上进行调节,自我组装成所谓的“纳米球”。这样的测序所需要的试剂更少,得到的数据则更多。技术人员都穿着无尘室服装,因为任何一点灰尘都会干扰测序,除非哪儿出问题了,一般而言这些技术人员不会干预机器人的工作。机器人则会自动添加试剂,操作样本,每个DNA纳米球上携带着70个核苷酸,其排列顺序会通过光信号被拍摄记录下来。费用正在逐步降低这些机器人正在做的工作,是一个浩大庞杂的工程蓝图中的第一步,所有的人类基因组中有着30亿对碱基对,而CG计划将其全部组装出来。这需要非常大的计算量,公司为此也建了一个自动数据中心。不过,这个数据中心设在距离公司大约有20分钟车程的地方那儿的电费更便宜。目前CG公司只针对研究者和制药公司开放,个人还没法购买他们的服务。在这里,每对基因组测序要价9500美元,如果购买1000对以上,则每对价格降为5000美元。这个价格是随着基因组测序技术突飞猛进而急剧下降的,要知道,十年前,第一对人类基因组序列完成时,其价格是以十几亿美元计量的。而科学家现在已经预计几年后,基因组测序的价格可能会降到一般人都可能支付得起的程度。基因组测序的流水线完全是由机器人来做的,而职员做什么呢?公司共有185名职员,部分是科研人员,忙于改善公司的测序技术,另一部分则是做市场和联络,与各类客户打交道。基因组测序工程是一项既有非常光明的前途但又异常庞大的科学工程,而自动化则可能成为处理这项工作的最佳工具。基因学家们认为,通过一些基因扫描,是可以找到导致人类易感疾病的一些基因变异,人类基因谱上,有一些常见明显变异,但是就整个遗传问题来看,还有大量的混乱的遗传变异隐藏在DNA双螺旋体中,这些也导致了世界上千奇百怪的遗传疾病。如何去捕猎这些神秘莫测的错误基因代码呢?只剩下一个方法,那就是将整个人类基因谱测序,来捕捉一些可能和疾病有关的基因变异。这个方法虽然听上去如同“大海捞针”一样不靠谱,但目前一些迹象表明,今后或许基因组序列会成为医疗记录的一部分,或者科学家可以通过家庭的基因组测序来纠正基因错误。比如,去年西雅图系统生物学研究所的胡德(Leroy Hood)及其小组与CG公司进行了合作,在《科学》杂志上刊登了一篇论文。他们对一家四口的基因组进行了测序。这是个特殊的家庭,两个孩子都患有两种隐性遗传病米勒综合征和纤毛运动障碍,而父母则完全正常,在分别测出这家人的基因序列后,研究者将父母和子女基因组序列进行比较,验证了米勒综合征这种非常罕见遗传病的致病突变。提供测序外包的服务目前,站在基因组测序产业化起跑线上的企业包括了同样位于加州的生物科学公司Pacific Bio。这个公司创立了首次可以对单个DNA进行测序的仪器。和CG公司一样,目前,这家公司也只向研究者提供服务。有一些大型的、从事基因组测序产业的公司已经将基因组测序做到医院和个人普及的地步了,如研发制造大型测序分析仪器的Illumina公司。这个公司在2008年美国成长最快的科技公司评选中,风头甚至盖过了Google。它们提供的产品甚至可以直接给病人使用。而另一位基因创业企业家罗斯伯格(Jonathan Rothberg)甚至发明了可以放在桌子上的基因解码器,可以在2小时之内以很高的精度解读出1000万个基因代码符号。大部分的基因组测序企业都站在一个竞争线上,尽力提高DNA测序的速度,降低费用。而CG公司其实并非和它们是严格意义上的竞争对手他们计划组装出所有的人类基因序列,研发也是为此目的而进行。此外,他们并不如其他公司一样开发更高级更小巧的基因组测序仪,而是为科学家提供基因组测序的外包服务,也就是说,研究人员无需购买、安装、培训、运行和维修仪器,而只要将样品交给这家公司,等待结果到来就可以。虽然很多人不理解他们的做法,但这家公司始终坚持自己的观点,认为这样的服务最能让科学家将时间从捣腾仪器设备的工作中解放出来,专心放在生物学和假说验证上。从这几年CG公司取得的成绩来看,这种做法确实是有效的。2009年,CG公司宣布其测出了第一个人类基因序列,并移交给美国生物科技信息中心数据库。同一年,他们在《科学》上刊文,发布了三个完整人类基因组序列分析的结果,当时文章还宣布,测序的成本已经可以降到1726美元。这在生物界引起了轰动。到了那一年结束,他们已经做出了50个人的基因序列。此外,他们的名字也随着来自各地的科学家一起多次登上了权威学术杂志。除了去年帮助科学家解开了米勒综合征突变难题给科学界留下难忘的印象之外,美国的罗氏公司还曾经借助CG的基因组测序技术,完成了人类科学史上第一例肺癌患者的全基因组比较。相关研究结果刊登在《自然》杂志上。而美国癌症学会也开始和CG公司联手,希望通过其服务比较正常人和癌细胞基因组序列的差异。或许在不久的将来,解开癌症之谜的第一个贡献就属于这些蓝光照耀下的机器人。
第一步:目的基因的制取: 用限制性内切酶直接对基因组DNA进行部分酶切,产生一系列大小不等的DNA片段。那里面含有一种或几种遗传信息的全套遗传密码。获取目的基因是基因工程操作的关键。基因工程的原料就是目的基因。所谓目的基因,是指已被或欲被分离、改造、扩增和表达的特定基因或DNA片段,能编码某一产物或某一性状。目前获取目的基因的方法主要有三种:反向转录法、内切酶切割分离法和人工合成法. 第二步:基因载体的选取: 用人工方法,取得目的基因的适宜的载体,即质粒(一种环状双链DNA)或病毒。载体一般带有必要的标志基因,以便进行检测。 基因克隆载体必须具备三个条件: a.具有能使外源DNA片段组入的克隆位点。 b.能携带外源DNA进入受体细胞,或游离在细胞质中进行自我复制,或整合到染色体DNA上随染色体DNA的复制而复制。 c.必须具有选择标记,承载外源DNA的载体进入受体细胞后,以便筛选克隆子。http://learn.gxtc.edu.cn/NCourse/swjs/gene/Images/bz1.jpg基因工程的基本过程(点击放大) 第三步:DNA的体外重组: 即用人工方法,让目的基因与运载体相结合,首先要用限制性内切酶和其他一些酶类,切割或修饰载体DNA和目的基因,然后用连接酶将两者连接起来,使目的基因插入载体内,形成重组DNA分子。这些工作都在生物体外进行,所以基因工程操作又叫体外DNA重组。 第四步:DNA重组体导入受体细胞: 将外源DNA片段与载体DNA连接形成DNA重组体,即重组DNA。 这种重组体连接的方法主要有: 粘性末端连接法:应用同一种限制性内切酶切割载体和外源DNA分子,可产生相同的粘性末端(接口处的碱基互补),进一步用DNA连接酶将断口连好,即可获得重组DNA分子。http://learn.gxtc.edu.cn/NCourse/swjs/gene/Images/zhuru.jpgDNA重组体导入受体细胞 钝性末端连接法:用化学合成法或逆转录法得到的外源DNA片段,均为钝性末端,这种末端也可以用特殊的连接酶连接,但效率太低。通常需要用人工方法加上粘性末端,再进行连接。第五步:受体细胞的繁殖扩增: 含重组DNA的活受体细胞,再在适当的培养条件下,并进行繁殖和扩增,使得重组DNA分子在受体细胞内的拷贝数大量增加。 第六步:克隆子的筛选和鉴定: 受体细胞经转化(传染)或传导处理后,真正获得目的基因并能有效表达的克隆子一般来说只是一小部分,而绝大部分仍是原来的受体细胞,或者是不含目的基因的克隆子。为了从处理后的大量受体细胞中分离出真正的克隆子,需要对克隆子进行筛选和鉴定。 第七步:目的基因的表达。
1 原位光刻合成寡聚核苷酸原位光刻合成技术是由Affymetrix公司开发的,采用的技术原理是在合成碱基单体的5'羟基末端连上一个光敏保护基。合成的第一步是利用光照射使羟基端脱保护,然后一个5'端保护的核苷酸单体连接上去,这个过程反复进行直至合成完毕。使用多种掩盖物能以更少的合成步骤生产出高密度的阵列,在合成循环中探针数目呈指数增长。某一含n个核苷酸的寡聚核苷酸,通过4×n个化学步骤能合成出4n个可能结构。例如:一个完整的十核苷酸通过32个化学步骤,8个小时可能合成65,536个探针。 2 原位喷印合成 芯片原位喷印合成原理与喷墨打印类似,不过芯片喷印头和墨盒有多个,墨盒中装的是四种碱基等液体而不是碳粉。喷印头可在整个芯片上移动并根据芯片上不同位点探针的序列需要将特定的碱基喷印在芯片上特定位置。该技术采用的化学原理与传统的DNA固相合成一致,因此不需要特殊制备的化学试剂。 3 点样法 点样法是将合成好的探针、cDNA或基因组DNA通过特定的高速点样机器人直接点在芯片上。采用的机器人有一套计算机控制三维移动装置;多个打印/喷印针的打印/喷印头;一个减震底座,上面可放内盛探针的多孔板和多个芯片。根据需要还可以有温度和湿度控制装置;针洗涤装置。打印/喷印针将探针从多孔板取出直接打印或喷印于芯片上。直接打印时针头与芯片接触,而在喷印时针头与芯片保持一定距离。打印法的优点是探针密度高,通常1平方厘米可打印2,500个探针。缺点是定量准确性及重现性不好, 打印针易堵塞且使用寿命有限。喷印法的优点是定量准确,重现性好,使用寿命长。缺点是喷印的斑点大,因此探针密度低,通常只有1平方厘米400点。国外有多家实验室和公司研究开发打印/喷印设备,目前有一些已经商品化。军事医学科学院目前正在利用打印/喷印技术进行生物芯片的研究和开发,预计2年内将有用于实验室研究或临床诊断的基因芯片产品问世。 4 电子芯片电子芯片是由美国Nanogen公司开发的,目前国内清华大学和复旦大学也在开发这一技术。这种芯片为带有阳电荷的硅芯片、芯片经热氧化,制成1mm(1mm的阵列、每个阵列含多个微电极,在每个电极上通过氮化硅沉积和蚀刻制备出样品池。将链连接亲和素的琼脂糖覆盖在电极上,在电场作用下生物素标记的探针即可结合在特定电极上。电子芯片的最大特点是杂交速度快,可大大缩短分析时间。制备复杂、成本高是其不足。 5 三维芯片三维芯片是由美国的Packard、摩托罗拉、Argonne国家实验室三家机构与俄罗斯Engelhardt分子生物学研究所合作开发的一种芯片技术。三维生物芯片的实质上是一块显微镜载玻片,其上有10,000个微小聚乙烯酰胺凝胶条,每个凝胶条可用于靶DNA,RNA和蛋白质的分析。先把已知化合物加在凝胶条上,用3cm长的微型玻璃毛细管将待测样品加到凝胶条上。每个毛细管能把小到0.2nl的体积打到凝胶上。以上几家机构构合作研究的生物芯片系统具有其它生物芯片系统不具有的几个优点。一是凝胶条的三维化能加进更多的已知物质,增加了敏感性。二是可以在芯片上同时进行扩增与检测。一般情况下,必须在微量多孔板上先进行PCR扩增,再把样品加到芯片上,因此需要进行许多额外操作。本芯片所用凝胶体积很小,使PCR扩增体系的体积减小1,000倍(总体积约纳升级),从而节约了每个反应所用的PCR酶(约减少100倍)。三是以三维构象形式存在的蛋白和基因材料可以其天然状态在凝胶条上分析,可以进行免疫测定,受体-配体研究和蛋白组分析。 6 流过式芯片(flow-thru chip) Gene Logic 正在开发一种在芯片片基上制成格栅状微通道,Gene Logic设计及合成特定的寡核苷酸探针,结合于微通道内芯片的特定区域。从待测样品中分离DNA或RNA并对其进行荧光标记,然后,该样品流过芯片,固定的寡核苷酸探针捕获与之相互补的核酸,采用Gene Logic's信号检测系统分析结果。流通式芯片用于高通量分析已知基因的变化,其特点在于(1)敏感性高:由于寡核苷酸吸咐表面的增大,流过式芯片可监测稀有基因表达的变化;(2)速度快:微通道加速了杂交反应,减少了每次检测所需时间;(3)价格降低:由于采用了特殊的共价化学技术将寡核苷酸吸咐于微通道内,使每一种流过式芯片可反复使用多次,从而使其成本降低。
2007年最令研究人员惊叹的是,从一个人到另一个人的基因组差异程度之大,科学家开始懂得这些差异在疾病和个体特性中的作用。《科学》杂志及其出版者美国科学促进会(AAAS) 将“人类基因组差异”评为2007年首要进展,并在12月21日出版的杂志上列出本年度其他9项最重要的科学成就。负责评选的《科学》杂志物理类科学新闻副主编Robert Coontz 说,“多年来,我们一直谈人与人如何相像,甚至人与猿如何类似。2007年的几项前沿研究第一次将人与人的DNA存在很大的不同讲透彻了。这是一个巨大的概念性跳跃,将会对所有的事情产生影响:从医生如何治病到我们如何看待自己以及保护我们的隐私。”2007年,几位个人的基因组被测序。随着技术的提高,我们中的许多人将会了解部分或全部的个人基因组,也将了解自己有患哪些疾病的风险。自人类基因组序列测出以来,生物学家一直在绘制基因组的一个碱基上的小差异,这种差异被称为单核苷酸多态性(SNPs)。这些差异是2007年十几个研究项目的关键,研究人员在这些被称为基因组范围关联的研究中比较了几千位患病或无病个体的DNA,从而确定哪些小的基因差异带来疾病风险。这种信息能帮助研究人员发现疾病基因,比如近年发现的几个2型糖尿病基因。今年的基因组范围关联研究为许多疾病提供了线索,包括心房颤动、自身免疫疾病、双相障碍、大肠癌、1型和2型糖尿病、心脏病、高血压、多发性硬化症以及风湿性关节炎。2007年,生物学家还了解到,在DNA上亿个碱基中,成千到上百万的碱基可能丢失、增加或以某种方式被拷贝,这些变化在几代人内就能改变基因的活性。这些被称为“拷贝数差异”的影响在高淀粉饮食的人群中有表现,这些人群比有狩猎采集传统的人群有更多的消化淀粉DNA的拷贝。研究儿童自闭症的遗传学家发现了导致患自闭症风险增加的一个新的DNA修饰。名列《科学》2007年十大进展第二位的是重新编程细胞的技术。日本和美国小组分别在6月宣布他们用小鼠皮肤制造了诱导性多能干(iPS)细胞,这些iPS细胞能产生身体的所有细胞,包括卵子和精子,从而显示iPS细胞具有胚胎干细胞的能力。11月份,两个小组分别报告了用人类皮肤细胞制造iPS细胞的研究。这项研究可能改变干细胞研究的科学与政策。Coontz说:“与首要进展一样,一旦科学家能清除几个障碍,重新编程细胞可能为生物医学研究开辟新方向。”《科学》评选出的其他8项进展包括:跟踪宇宙射线来自阿根廷Pierre Auger天文台的研究人员报告说,进入地球大气的宇宙线可能来自天空中存在着许多活跃星系核的区域。这些宇宙线可能是经过黑洞附近的磁场时获得加速度的。受体结构研究人员确定了人类Beta2-肾上腺素能受体的结构,这是一个重要的G蛋白偶联受体,它通过传递体内的激素、血清素以及其他分子的信息管理人体内部系统。从抗组胺剂到beta阻滞剂的一系列药物以这些受体为靶标。结构知识可能带来新的药物。超越硅电子器件过渡金属氧化物研究的进展也许预示了下一个材料革命,2007年,几个研究小组将两种氧化物结合在一起,制造了带有各种有用的电子和磁性性能潜力的界面。量子霍尔效应理论和实验物理学家制造了预测的量子霍尔效应,这是电子从某些材料中流过时在外加电场作用下的奇怪行为。如果这一效应在室温下工作,它可能导致新的低功率的“自旋电子学”计算的设备。分而治之研究揭示,与病毒和肿瘤作战的T细胞有立刻保护和长期保护的分工,改进的疫苗也许使这项研究成果得到应用。研究人员发现,当他们捕捉到刚刚分化的T细胞时,在T细胞相反的两极有两类蛋白质被生成,一边的蛋白带有“战士”的分子标记,另一边的显示“记忆细胞”的特征,记忆T细胞能潜伏多年以防备未来的入侵。以少胜多合成化学家研制了一个高效低成本的制造药物和电子化合物的技术。返回未来用人和大鼠作的研究提出,记忆和想象扎根于大脑的海马区,该区是记忆的一个关键中心。研究人员推测,大脑的记忆也许能通过重新整理过去的经历来产生未来的情景。游戏结束一个人工智能编程的精心杰作使双陆棋成为迄今为止计算机解决了的最复杂的游戏。研究人员发现,如果竞技双方不犯任何错误,双陆棋将以平局结束。2008年应该注意的领域包括microRNA、人工制造的微生物、新的计算机芯片材料、人类细菌以及尼安德特人的基因组、人类神经回路以及来自CERN的大型强子对撞机的数据。
"20世纪70年代以来,随着生物技术的飞速发展,尤其是基因工程技术的成熟,转基因技术在农作物的改造中得到广泛运用,转基因农作物的面积越来越大。正是因为转基因品种具有他独特的优势,比如具有很强的抗病虫害能力、高产、减少劳动投入等,给种植的人带来了巨大的经济效益,也降低了成本和人的劳动。但由于转基因食品对于人体的影响,并未经受长期的检验或者有明确的论点证明,因此转基因食品遭到大多数国家及其民众的反对和质疑。将未经验证安全可靠的食品投入市场为民所食用,是极度不安全也是不负责任的做法。因此国家食品安全法指出必须对转基因食品进行标识。而且个人认为,改变传统生物进化的做法是有违进化论,以及人类生命特征发展的。基因是决定一个个体的基础,倘若基因变了,对于食用者真的就没有一丝改变么,而这发生的改变是好是坏如今依旧无法定论。因此对于转基因食品应该做出检测并贴出明确的标识。对转基因食品的检测方法,目前主要有对外源基因的检测和对外源蛋白质的检测二大类。1. 外源基因的检测现阶段对于外源基因的检测主要是对转入的外源基因进行PCR扩增,进而在做紫外检测或荧光检测。PCR技术全称“聚合酶链反应技术”,这是一种聚合反应,是在体外由引物介导的DNA聚合酶催化的,能在短时间内准确地大量复制序列。目前英格尔检测公司(ICAS)是用自己独立的实验室,通过这种方法在做转基因检测。2.蛋白质印迹法蛋白质印迹法将电泳的较高的分离能力、抗体的特异性和显色酶反应的灵敏性结合起来,是检测复杂混合物中特异蛋白质的最有力的工具之一,普遍用于分离、检测特异的目的蛋白质,灵敏度为1-5ng。它可确定一个样品中是否含有低于或超过预定限值水平的目的蛋白质,特别用于不可溶蛋白质的分析。
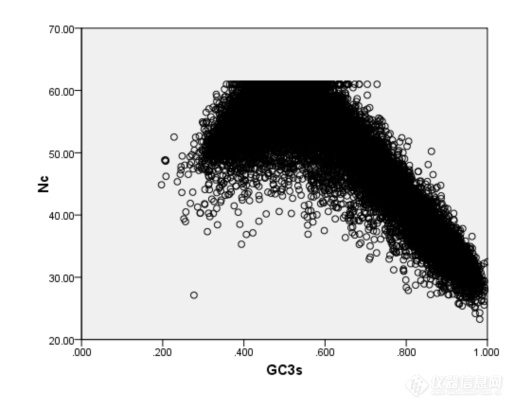
[align=center]短柄草全基因组密码子用法分析分析[/align]摘要:本研究运用CodonW程序分析了短柄草全基因组的密码子使用特性,并且通过对应分析探讨了若干重要因子对短柄草全基因组序列密码子用法的影响。结果表明短柄草基因组存在高[url=https://insevent.instrument.com.cn/t/Mp]gc[/url]含量和低[url=https://insevent.instrument.com.cn/t/Mp]gc[/url]含量的基因,它们在密码子使用上差异较大。Nc-plot曲线表明基因组的密码子组成受到碱基组成的影响;对应分析显示,在DNA水平上发生的核苷酸突变可能是造成短柄草基因组密码子使用偏好的主要因素;同时,基因长度和蛋白质疏水性对密码子的使用也存在一定偏性,但影响程度不大。确定了UUC等27个以G或C碱基结尾的密码子为“最优密码子”,研究结果可为短柄草基因的鉴定、表达、结构、功能等的深入研究提供参考。关键词:同义密码子偏好性,短柄草基因组,对应分析近年来,随着分子生物学的快速发展,许多小基因组的低等生物和高等模式生物的全基因组序列均被测定,为利用生物信息学方法挖掘海量基因组数据提供了便利。密码子是生物体内遗传信息传递的基本环节,是核酸携带信息和蛋白质携带信息间对应的基本规则。在长期进化过程中,任一物种的基因都会逐渐适应宿主的基因组环境,而形成特定的且符合宿主基因组的密码子用法,因此不同生物具有不同的密码子使用模式。以生物基因组数据为基础,研究其密码子使用模式,为深入研究基因的结构、功能和基因组进化,以及指导基因转化等具有重要意义。密码子具有简并性,生物在同义密码子的使用上并不是完全随机的,而是具有一定的偏向性,对有的密码子使用频率高,有的使用频率低,甚至避免使用,这种不均衡使用密码子的现象普遍存在于原核和真核生物中。早在20世纪70年代,人们在研究基因的异源表达时,就已经意识到密码子偏性的重要性[1],随着不同生物基因组数据的获得和各种数据库的构建,更多的研究者对密码子偏性的研究产生了浓厚的兴趣,尤其在分子进化,翻译调控等研究领域,通过对不同物种的密码子使用偏性的大量研究[2~4],发现不同物种的基因在密码子使用上存在着明显的偏性。 短柄草是一种广泛分布于温带地区的禾本科植物,与小麦,大麦和燕麦同属早熟禾亚科,原产于非洲北部,欧洲南部和亚洲中部,包含约10个亚种。该植物为一年生,自花授粉,植株高度15~20cm,生育期70~80d,柄草植株较小,适应性强,不象种植水稻那样需要严格的生长条件。生育期短,籽粒产量较高,一年可以繁殖4~5代,繁殖系数达140左右。未成熟胚和成熟胚愈伤组织诱导率高,农杆菌介导和基因枪介导的转化体系已经建立,胚性愈伤组织分化率90%以上,转化效率最高可达55%左右。基因组小,染色体少,DNA重复序列低,获得突变体容易,突变性状容易显现,具备了模式植物的所有基本特征。加之短柄草基因组序列与黑草麦,小麦,大麦等早熟禾亚科植物高度相似,很多重要农艺性状与温带禾草类植物相似,如株型,穗型,粒型,抗逆性,生长习性和病原菌等,其中麦类作物白粉病菌,条锈病菌和稻类作物瘟病菌都可侵染短柄草植株,引起相应症状[7]。其籽粒不含高分子量麦谷蛋白亚基,低分子量麦谷蛋白亚基也很少,并与小麦一样具有二倍体,四倍体和六倍体,因此短柄草是小麦等基因组庞大的重要农作物理想的模式植物,借此来获得目前小麦等早熟禾类植物中尚缺少的遗传信息和基因共线区,进而对小麦等重要植物进行基因定位,克隆,突变,测序和功能等方面的研究[8]。 目前,在短柄草的生物学、细胞学和遗传学特性方面开展了大量研究,并且其全基因组测序也基本完成[9],为深入研究其密码子用法提供了便利。因此本研究将以短柄草全基因组序列为基础,分析其基因的密码子用法特性和影响密码子使用的因素等,其研究结果将对指导转基因及对基因进行特定分子改造,提高其在短柄草中的表达效率和完善基因预测软件,提高基因预测和基因组注释准确性等均具有重要的参考价值,同时也为深入开展基因结构和功能,分子进化等研究提供理论基础。1.实验材料与方法1.1材料 短柄草全基因组DNA序列来源于短柄草官方数据库(http://www.brachypodium.org/node/8),根据基因组序列的注释信息,获得蛋白编码基因序列,为了减少长度较短的基因变异带来的样本误差,根据国际惯例,去除小于300bp的基因,去除中间不表达的密码子,终止密码子。编写程序提取剩下的蛋白编码基因的CDS(coding sequence)序列。1.2方法用codonw软件计算短柄草全基因组的密码子用法相关参数,主要包括有效密码子数(Effective Number of Codon,ENC)、基因的G+C含量([url=https://insevent.instrument.com.cn/t/Mp]gc[/url]%)、[url=https://insevent.instrument.com.cn/t/Mp]gc[/url]3s%、相对同义密码子使用度(relative synonymous codon usage,RSCU)、氨基酸组分指数(平均亲水性值(gravy))、基因长度即氨基酸数(L_aa)。其中,有效密码子数(Effective Number of Codon,ENC)描述密码子使用偏离随机选择的程度,能反映密码子家族中同义密码子的非均衡性的偏好;其取值范围在20到61之间,即如果每种氨基酸只使用一种密码子则有效密码子数为20,如果各种同义密码子的使用机会完全均等,则有效密码子数为61,数值越小偏性越强。此值是以描述密码子使用偏离随机选择的程度,能反映密码子家族中同义密码子的非均衡性的偏好。基因密码子偏爱程度越大,ENC值越小。RSCU是指对于某种特定的密码子在编码对应氨基酸的同义密码子间的相对频率;[url=https://insevent.instrument.com.cn/t/Mp]gc[/url]3s%表示同义密码子第三位碱基的G+C的含量。为进一步了解该家族基因密码子使用特征和影响密码子使用的因素,对7个基因的相对同义密码子使用度进行了对应性分析(correspondence of analysis,COA)。2 结果与分析2.1 基因的碱基组成对密码子使用的影响图一 短柄草基因NC值散点图[img=,515,409]https://ng1.17img.cn/bbsfiles/images/2019/10/201910311236371230_3093_3295053_3.png!w515x409.jpg[/img]2.2短柄草基因密码子使用特性的对应性分析[img=,690,535]https://ng1.17img.cn/bbsfiles/images/2019/10/201910311237226440_1452_3295053_3.png!w690x535.jpg[/img][img=,690,534]https://ng1.17img.cn/bbsfiles/images/2019/10/201910311237233450_935_3295053_3.png!w690x534.jpg[/img]2.3 确定最优密码子Phe UUU 0.05 (323) 1.23 (19733) Ser UCU 0.22 (990) 1.60 (23834) UUC* 1.95 (13527) 0.77 (12294) UCC* 2.55 (11715) 0.64 (9499) Leu UUA 0.02 ( 93) 0.83 (11755) UCA 0.14 (629) 1.52 (22651) UUG 0.16 (1003) 1.37 (19558) UCG* 1.53 (7023) 0.35 (5159) CUU 0.14 (847) 1.55 (21987) Pro CCU 0.22 (1306) 1.57 (17584) CUC* 3.38 (20676) 0.61 (8661) CCC* 1.35 (7940) 0.47 (5299) CUA 0.07 (452) 0.70 (9983) CCA 0.20 (1184) 1.62 (18078) CUG* 2.23 (13637) 0.94 (13401) CCG* 2.22 (13058) 0.34 (3792) Ile AUU 0.12 (398) 1.41 (21216) Thr ACU 0.10 (401) 1.46 (16515) AUC* 2.76 (9124) 0.70 (10557) ACC* 1.75 (7291) 0.66 (7397) AUA 0.12 (380) 0.89 (13461) ACA 0.12 (509) 1.56 (17636) Met AUG 1.00 (8512) 1.00 (20892) ACG* 2.03 (8478) 0.32 (3563) Val GUU 0.10 (693) 1.67 (23852) Ala [url=https://insevent.instrument.com.cn/t/Mp]gc[/url]U 0.14 (1914) 1.65 (26184) GUC* 1.71 (12491) 0.63 (9025) [url=https://insevent.instrument.com.cn/t/Mp]gc[/url]C* 1.98 (27398) 0.58 (9131) GUA 0.05 (349) 0.75 (10713) [url=https://insevent.instrument.com.cn/t/Mp]gc[/url]A 0.13 (1802) 1.48 (23459) GUG* 2.14 (15605) 0.95 (13562) [url=https://insevent.instrument.com.cn/t/Mp]gc[/url]G* 1.75 (24170) 0.29 (4678) Tyr UAU 0.05 (229) 1.28 (14480) Cys UGU 0.06 (194) 1.10 (9360) UAC* 1.95 (8126) 0.72 (8075) U[url=https://insevent.instrument.com.cn/t/Mp]gc[/url]* 1.94 (6645) 0.90 (7595) TER UAA 0.42 (172) 0.82 (335) TER UGA 1.63 (665) 1.30 (530) UAG 0.94 (384) 0.87 (356) Trp UGG 1.00 (4992) 1.00 (10053) His CAU 0.15 (598) 1.42 (16785) Arg CGU 0.16 (750) 0.85 (6945) CAC* 1.85 (7568) 0.58 (6825) C[url=https://insevent.instrument.com.cn/t/Mp]gc[/url]* 2.75 (12565) 0.49 (4043) Gln CAA 0.15 (627) 1.05 (20215) CGA 0.11 (500) 0.64 (5273) CAG* 1.85 (7975) 0.95 (18259) CGG* 1.92 (8761) 0.55 (4527) Asn AAU 0.12 (465) 1.31 (26650) Ser AGU 0.05 (235) 1.13 (16754) AAC* 1.88 (7141) 0.69 (13985) A[url=https://insevent.instrument.com.cn/t/Mp]gc[/url]* 1.52 (7002) 0.77 (11441) Lys AAA 0.11 (552) 0.98 (27077) Arg AGA 0.10 (445) 1.94 (15854) AAG* 1.89 (9406) 1.02 (28423) AGG 0.96 (4387) 1.53 (12516) Asp GAU 0.15 (1344) 1.44 (39136) Gly GGU 0.11 (882) 1.34 (18423) GAC* 1.85 (16539) 0.56 (15322) G[url=https://insevent.instrument.com.cn/t/Mp]gc[/url]* 2.53 (20795) 0.71 (9826) Glu GAA 0.17 (1437) 1.13 (36292) GGA 0.19 (1522) 1.26 (17423) GAG* 1.83 (15812) 0.87 (27746) GGG* 1.18 (9700) 0.69 (9476) 注:Number of codons in high bias dataset 372333 Number of codons in low bias dataset 915109标注*的密码子是(p 0.01)3 讨论密码子使用偏好是突变偏好、自然选择和遗传漂变等共同作用的结果,与碱基组成、翻译选择压力、基因表达水平、基因长度、蛋白质氨基酸组成、碱基突变频率和模式、mRNA二级结构稳定性等很多因素有关[17]。张晓峰[18]等研究表明,单子叶植物基因组的[url=https://insevent.instrument.com.cn/t/Mp]gc[/url]含量在同义密码子使用偏性的产生过程中起着决定性的作用,同义密码子使用偏性强烈的基因往往偏爱使用C或G结尾的密码子,且第三位密码子突变往往是密码子偏好性发生变化的决定原因。短柄草基因密码子使用模式的调查表明其中有高含量的[url=https://insevent.instrument.com.cn/t/Mp]gc[/url],并且[url=https://insevent.instrument.com.cn/t/Mp]gc[/url]3的含量高于[url=https://insevent.instrument.com.cn/t/Mp]gc[/url]1和[url=https://insevent.instrument.com.cn/t/Mp]gc[/url]2。这表明相对于以A和T结尾的密码子而言,这些基因偏好于使用以G或C结尾的密码子。从原核生物到真核生物的基因中,密码子使用偏好是一个被广泛研究的重要进化现象。研究发现,许多因素,比如碱基组成,基因表达水平,蛋白质疏水性等影响着密码子的使用。为了解释密码子使用偏好的起因,也有许多假设被提了出来。其中被广为接受理论是“选择——突变——漂移”模型。该模型认为在对偏好密码子的选择和通过突变-漂移对非偏好密码子的保留之间,同义密码子的使用偏性存在一种平衡。本文的研究结果显示,[url=https://insevent.instrument.com.cn/t/Mp]gc[/url]3s值与ENC值密切相关,并且基因也位于第一轴线,揭示了碱基组成是影响短柄草基因组中的密码子使用偏好的主要因素。碱基组成是影响短柄草基因密码子使用的主要因素,基因长度和蛋白质的疏水性在短柄草基因密码子使用中也起到了一定的作用,相似的结果在水稻、小麦中被发现[15,19]。本研究发现,在基因长度和[url=https://insevent.instrument.com.cn/t/Mp]gc[/url]之间存在很强的负相关性。这表明,高[url=https://insevent.instrument.com.cn/t/Mp]gc[/url]含量的基因越短,密码子偏好就越大。可能的原因是富含AT基因的翻译效率比富含[url=https://insevent.instrument.com.cn/t/Mp]gc[/url]基因的翻译效率更高,这种效率的差异对长的基因更为重要。通常,全基因组的基因表达值在许多多细胞真核生物中并不能得到,特别是基因表达水平在不同的组织和不同发育阶段不一样时。因此,要定量相当困难。在短柄草基因组中,目前还缺少相当数量的基因表达的准确数据。另外,我们发现[url=https://insevent.instrument.com.cn/t/Mp]gc[/url]含量特别是在第三个碱基位置的[url=https://insevent.instrument.com.cn/t/Mp]gc[/url]含量较大的影响着密码子的偏好时,暗示着碱基突变可能是重要因素,同时,碱基突变又受控于翻译选择。所以,尽管基因表达水平影响着密码子的使用,但这影响还是远远小于核苷酸组成对密码子使用的影响。因此,我们没有进一步分析基因表达的影响。通过优化密码子,提高外源基因在微生物、植物、动物中的表达已有不少成功报道,而确定最优密码子可为合理有效进行密码子改造提供可靠信息。本文确定了UUC等27个密码子为短柄草全基因组的最优密码子。分析结果可为指导转基因及对基因进行特定分子改造,提高其在短柄草中的表达效率和完善基因预测软件,提高基因预测和基因组注释准确性等提供重要的参考价值。参考文献[1] Stanley D,Farnden K J F, MacRae E A. Plant a-amylases:Func-tions and roles in carbohydrate metabolism[J]. Biologia,Bratislava,2005.60(suppl l6):65-71[2] Smith AM. Zeeman SC, Smith S M. Starch degradation[J]. Annu Rev Plant Biol,2005,56(25):73-98[3] Asatsuma S, Sawada C, Itoh K et al. Involvement of α-amylase I-1 in starch degradation in rice chloroplasts[J]. Plant Cell Physiol,2005,4:858-869[4] Kaplan F, Guy C L. β-amylase induction and the protective role of maltose during temperature shock[J]. Plant Physiol, 2004, 1:1674-1684 [5] Kaplan F,Guy C L. RNA interference of Arabidopsis beta-amylase 8 prevents maitose accumulation upon cold shock and increases sensitivity of PSII photochem-ical efficiency to freezing stress[J]. Plant J.2005,44(13):730-743[6] Joho Mundy, Anders Brandt. Messenger RNAs from the Scutellum and Aleurone of Germinating Barley Encode (lm3,14)--D-Glucanase, a-Amylase and Carboxypeptidase[J]. Plant Physiol, 1985,79(5):867-871 [7] 言普,李桂双.高压对水稻种子细胞膜透性和淀粉酶活性的影响[J]. 浙江大学学报(农业与生命科学版),2007,33(5):174-179[8] Monica M, Sanwo and Darleen A. DeMason. Characteristics of a-Amylase during Germination of Two High-Sugar Sweet Corn Cultivars of Zea mays L[J]. Plant Physiol, 1992,99(8):1184-1192[9] Goldman N , Yang Z. A codon based model of nucleotide substitution for protein coding DNA sequences[J]. Molecular Biology and Evolution,1994,11(9):725-736[10] Schmidt W. Phylogeny reconstruction for protein sequences based on amino acid properties[J]. Mol Evol,1995,41(8) :522-530[11] 时成波, 吕安国.改造稀有密码子提高SEA蛋白表达量[J]. 生物工程学报,2002,18(4):477-480[12] Ghosh T C , Gupta S K, Majumdar S. Studies on codon usage in Entamoeba histolytica[J]. Int J Parasitol,2000,30(6): 715-722[13] Musto H, Cruveiller S. Translational selection on codon usage in Xenopus laevis[J].Molecular Biology and Evolution,2001,18(9):1703-1707[14] 廖登群,张洪亮等. 水稻(Oryza sativa L.)a-淀粉酶基因的进化及组织表达模式[J]. 中国农业大学学报,2009,14(5):1-11[15]刘汉梅,何瑞. 玉米密码子用法分析[J]. 核农学报,2008,22(2):141-147[16] Jia M, Luo L. The relation between Mrna folding and protein structure[J]. Biophys Res Commum, 2006,343(4):177-182[17] 赵耀,刘汉梅. 玉米waxy基因密码子偏好性分析[J]. 玉米科学,2008,16(2):16-21 [18] Wang H C,Hickey D A. Rapid divergence of codon usage patterns within the rice genome[J].BMC Evol Biol,2007,15(8):347-356
记者昨日从香港消委会获悉,在近日一项针对香港市面销售的豆浆的测试发现,声称"有机"或"非基因改造大豆制造"的样本竟然检出微量基因改造大豆成分,包括了标称为"大和豆浆(原味)"和"永和非基因改造豆浆(原味)"两款产品。 据介绍,该会测试的50款样本都是预先包装豆浆,包括即饮和冲剂两类产品,购自香港当地的超市、便利店、专门店或零售店。结果显示,25款样本没有检出基因改造大豆成分。其余25款样本检出微量的基因改造大豆成分,大都低于定量限值。 全部检出含微量基因改造成分的样本都没有标示"含基因改造成分"或相关字眼。其中4款的基因改造成分足以被定量,含量由0.2%至1.1%.在上述4款被定量的样本中,两款豆浆附有"非基因改造"的标示。 昨日,家乐福、华润万家、百佳、吉之岛等广州大型超市负责人均表示,没有出售这两款豆浆。
基因芯片技术对于疾病耐药性检测可从两个方面加以实现:1.在肿瘤中,通过检测肿瘤耐药基因的表达变化来分析对药物的抗性;2.在感染性疾病中,病原体的耐药性检测可通两种方式:表达谱芯片检测药物诱导的表达改变来分析其耐药性;寡核苷酸芯片检测基因组序列的亚型或突变位点从而分析其耐药性。一、多药耐药基因的表达检测肿瘤治疗中对细胞毒素药物的抗性是引起治疗失败的重要原因,是限制化疗的重要因素。机制是复杂的,由肿瘤的综合特征决定,如存活细胞的比例、血液的供给是否充分、特殊的细胞机制及多药耐药表型,多药耐药是指当肿瘤细胞暴露在某一化学治疗药物后会产生对此药及其他结构上没有联系的药物的交叉抗性,可由不同的机制引起,如MDR1、MRP、LRP等基因的过度表达,拓扑异构酶II和谷胱甘肽代谢的改变等,另外,其他促进DNA修复和抑制细胞凋亡的基因表达改变也可能导致多药耐药。检测多药耐药基因表达的变化不但可以研究恶性肿瘤的不同耐药机制,还可以用于临床诊断,以指导制定治疗方案。目前已建立了几种多药耐药检测方法,在RNA水平上有:Northern blot、Slot blot、RT-PCR、Rnase protection assay和原位杂交,从蛋白水平上的检测方法有免疫组化、Western blot及流式细胞仪等。这些方法一次只能对一个基因进行研究,效率低,难以定量检测耐药基因表达增加的幅度。基因表达谱芯片可同时对成千上万的基因表达进行检测,可以大大加速这方面的研究,在设计芯片时,可以将已知肿瘤相关基因及标记基因都点到芯片上,同时,芯片上还包含目前所有报导过的耐药基因。这样可以同时得到肿瘤的各个方面的信息。另外基因芯片还可以帮助发现新的耐药基因。二、病原体耐药性检测细菌对三种以上不同类抗菌药物耐药者即可称为多重耐药菌(multi-drug resistant bacteria, MDR)。MDR感染在全球的状况十分严重,对婴幼儿、免疫缺陷者和老年人的威胁巨大,1992年美国疾病控制中心(CDC)的资料表明,有13300例住院患者,是因为对所使用的抗菌药物耐药,细菌感染得不到控制而死亡。MDR感染已成为治疗上的难点和研究上的热点。MDR大多为条件致病菌,革兰阴性杆菌(GNR)占较大比例,如肠杆菌科中的肺炎杆菌、大肠杆菌、阴沟杆菌、粘质沙雷菌、枸橼酸菌属、志贺菌属、沙门菌属等,以及绿脓杆菌、不动杆菌属、流感杆菌等。革兰阳性菌中有甲氧西林耐药葡萄球菌(MRS),尤以MRSA和MRSE为多;万古霉素耐药肠球菌(VRE),近年来在重症监护室(ICU)中的发病率有明显增高;青霉素耐药肺炎链球菌(PRSP),常引起肺炎、脑膜炎、菌血症和中耳炎,人结核分支菌等。此外尚有淋球菌、脑膜炎球菌、霍乱弧菌等。耐药性又称抗药性,一般是指病原体的药物反应性降低的一种状态。这是由于长期应用抗菌药,病原体通过产生使药物失活的酶、改变原有代谢过程,而产生的一种使药物效果降低的反应,因而作用的剂量要不断增加。细菌对抗菌药物的耐药机制可有多种,最重要者为灭活酶的产生,如β-内酰胺酶、氨基糖苷钝化酶等;其次为靶位改变如青霉素结合蛋白(PBPs)的改变等;其他尚有胞膜通透性改变,影响药物的进入;细菌泵出系统增多、增强,以排出已进入细菌内的药物;以及胞膜主动转运减少、建立新代谢途径、增加拮抗药物等,两种以上的机制常可同时启动。耐药菌及MDR的发生和发展是抗菌药物广泛应用,特别是无指征滥用的后果。找到耐药菌的耐药基因,从而根据这些耐药基因设计新型抗生素,或将耐药菌分成不同的亚型,针对不同的亚型在临床上使用相应的抗生素,达到改善治疗效果的目的。国外采用基因芯片技术,检测耐药菌基因的改变,即检测耐药基因。如Michael Wilson就曾使用此方法检测到肺结核杆菌中脂肪酸合成酶II、fbpC、efpA、fadE23、fadE24和ahpC基因发生改变与耐药性有关。提供了新药物作用的靶目标,并指导抑制这些靶目标试剂和药物的合成。在感染性疾病中,病原体的耐药性检测可通过两种方式:1.表达谱芯片检测药物诱导的基因表达改变来分析其耐药性;2.寡核苷酸芯片检测基因组序列的亚型或突变位点从而分析其耐药性。用基因芯片不仅可以同时检测耐药菌的多个耐药基因,还可以同时对多个耐药菌的多个耐药基因进行检测。对临床上用药和新药物的合成均具有指导作用。
随着生命科学迅猛的发展,与之相适应的实验技术手段越来越复杂和专业化,对于科研人员实验技能要求也越来越高。由于生物学实验高度的复杂性、使得一个科研人员往往只能精通某一方向的实验。因此许多生物技术公司应孕而生,他们提供了许多专业的服务,其中最常规的技术服务包括核酸、蛋白测序、引物合成,SNP分型,动物培养及转基因动物实验。在国外,生命科学的研究已经初步形成了一个大型技术平台共享的研究形式,科研人员的主要工作是进行实验方案的设计、若干实验和实验数据的分析工作,而大量的基础性的实验工作则由相应的技术公司来完成,据不完全统计,2004年全美60%的SNP分型工作就是外包的生物技术公司完成。因为专业的技术公司在各自领域有着雄厚的技术背景和丰富的实验经验,试验成功率更高,实验数据也更可靠,这样科研人员也能够将主要精力放在真正的科研-实验设计和数据分析中去。出具有针对性的SNP分型技术,“巢式PCR-RFLP”基因分型技术,不同于传统的只能对于有酶切位点的传统的SNP分型的技术,本公司开发的“巢式PCR-RFLP是针对任意的基因分型技术,使任何位点最终都能进行常规限制性内切酶鉴定;同时该SNP分型技术利用巢式PCR技术,使得多个位点同时进行分析称为可能。即使低浓度的DNA也能够进行快速准确的分型工作。 与现有常规的分型技术相比,其最大的优势包括:(一)所需DNA浓度低。1-2ng/uL也能完成检测工作,而Taqman技术所需的DNA含量至少5ng/uL。(二)价格低。由于不需要合成荧光探针,使得该技术比Taqman技术价格低,该优势在对多个位点同时进行分型时更明显。与Taqman技术和普通技术的详细对比分型技术 PCR-RFLP技术 Taqman技术 普通PCR-RFLP技术 最佳运用时机 200-600样本,任何DNA多态位点 样本量大于500人份 仅适合含有现成酶切位点的多态位点的分型 试剂投入 低 荧光探针,需3000-4000元。有可能实验失败需重新花钱设计荧光探针。 低 技术的适用性 可适合任何多态位点的分型 基本适合任何多态位点的分型,但有时若干位点不能成功进行试验;当分型位点过于接近也不能用该方法。 仅适合含有现成酶切位点的多态位点的分型,有时出现的是一些稀有酶的酶切位点,导致效率低下或费用昂贵; 结果判断 直观,明确,稳定 间接,每管反应必须加荧光参照ROX,否则结果判断不确定 一般直观,明确;有时酶切位点出现在非主频等位基因上,导致结果判读困难 结果稳定性 稳定 一般 稳定 结果准确性 准确度高 一般 准确度高 样本消耗量 1-2ng 5-10ng 50-100ng 实验耗时 2-3周 由于要定制MGB荧光探针,和预实验,也需要2-3周 2-3周操作流程 1.基本信息收集具体内容包括客户信息、样本数(case,control)数、位点信息等。 2 位点信息的查询 主要包括:2.1 位点上下游各300bp的确切序列,基因频度;2.2 有无文献报道证明该位点多态存在于中国人群;2.3 如无上述相关文献,在J-SNP数据库中查询该位点多态是否存在于东亚人群中。为了保证结果的可靠,实验方案设计原则上应以野生型进行酶切鉴定,因此确认snp的频度是非常重要的。3.样品质控从全部样品中取8%样品,即96个样品中取8个样品,用我们的一对特异的PCR引物进行样品质控,以检测送来样品是否良好。因为客户样品质量是实验成功的关键因素之一,只有客户提供的样品质量可靠稳定,那么结果自然就好。4.引物设计同时设计AB两套方案,分别以正链和负链为模板设计引物。5.单管测试进行实验方案可行性尝试,首先要在多种温度,多种Mg离子、多种添加剂加入的情况下进行方案的可行性测试,对于所有单管测试的结果都送去测序,以保证序列的正确性,在实验结果得到确认的基础上,挑选稳定的方案进入中试过程。6.中试抽取8个样品做小规模批量实验,以模拟和检查批量PCR情况和酶切情况。7.大规模实验准备将DNA模板按方案进行一次性分板(有冗余),并引入阳性和阴性对照,同时将所有相关的PCR体系进行一次性大规模分装,防止污染。 8.第一板数据先用一板模板进行一轮PCR和酶切,以检验体系和分板的情况。 9.正式实验相关技术要点 1.实验设计必需保证野生型的被限制性内切酶切开因为对于只有少量突变型的SNP位点,如果突变型被切开,那么所得到的实验结果就是大量没有被酶切开的PCR产物,而这个结果与酶切实验失败的结果非常类似,不易区分,对结果的判读造成很大的麻烦,因此保证了主频碱基被解开,从根本上保证了实验的可靠性。2.偏向性扩增的解决。所谓偏向性扩增是因为SNP位点上会往往存在嘌吟和嘧啶的替换,在PCR过程中,聚合反应对嘧啶(C或T)比较敏感,使得嘌吟(A或T)的扩增非常少,而出现偏向性扩增,这在SNP位点附近嘌吟含量较高时特别明显。为此,本公司专门设计一个方案,用以克服偏向性扩增。3.方案设计中的内切酶的选择虽然从理论上,我们的方案可以进行任意位点改造,但事实上在改造过程中会出现一系列的问题,包括扩增效率,碱基划移等一系列问题,使得改造方案失败。对此,我们公司专业开发了一个引物设计软件,通过运算获得最佳的改造方案,同时通过在正向和反向序列上同时设计方案,以保证试验的成功。4.PCR产物的污染问题在大规模的PCR过程中,最严重的问题是PCR产物污染,据我们经验总结,80%以上的污染是由于接触式污染,剩余的污染则包括气溶胶污染等环境造成的污染。对于PCR污染这个问题,本公司制订了严格的实验措施,具体包括使用滤芯抢头、所有PCR体系全部分装、PCR体系与各类模板严格分离、配置体系人员与接触PCR产物人员严格分离等一系列措施,同时在处理PCR管,eppendorf管时也规定了详细的操作过程。5.关于质量控制问题在每板PCR过程中,我们会放入两个阴性对照和一个重复对照,以确认PCR的过程中不会产生污染和PCR的结果确实可信。技术基本原理和实例采用的巢式PCR-RFLP分型技术,其基本原来是利用PCR引物的3’端,对SNP位点附近的碱基进行人为改造,产生一个常规的限制性内切酶识别序列,如SNP rs1321425(rs1321425-来自NCBI的refSNP数据库)的C/G,其任何一个碱基和上下游碱基无法形成一个可直接被限制性内切酶识别的序列,于是我们对SNP位点前的上游碱基进行改造,通过对序列的分析和PCR效果的计算,我们将原本四个碱基AGAT改成CTGC,结合后一个碱基A以及SNP位点G,形成CTGCAG(PstI)酶切识别位点,经测序和序列对比,改造成功。又比如rs9309462和rs4646642,成功的将2个碱基,3个碱基进行成功的改造。rs1321425TCACAAAAAATAAAAAATTTTAATTTTAAAGGAGATA C/G ACAAGAAATGAGCATGTGTGAAAGCAC TTCTGTAAACTACATGCACTAAT