美国与中国共产党战略竞争众议院特别委员会主席迈克加拉格尔(R-WI)和高级成员拉贾克里希纳莫蒂(D-IL)今天提出了一项法案,以确保外国生物技术公司无法获得美国纳税人的资金。一旦颁布,该立法将限制联邦资助的医疗服务提供者使用外国对手生物技术公司,包括华大基因集团及其子公司华大智造和Complete Genomics,以及另一家中国-公司药明康德。罗姆尼参议员表示: “中国的生物技术公司正在通过医疗诊断测试收集世界各地数百万人的基因和敏感健康数据,以使中国占据上风。” “我们的两党立法禁止与中国有联系的公司签订联邦合同和融资机制,这有助于确保美国纳税人的钱不能被用来补贴威胁我们国家安全的生物技术公司。”“每天,美国人都会抽血或接受其他医学检查以保护他们的健康。但是,很少有人确切知道谁有权访问这些样本中包含的 DNA 信息,或者他们会如何使用这些信息。从 DNA 检测试剂盒到医疗诊断,随着生物技术领域在日常生活中变得越来越重要,外国对手控制的生物技术公司构成的威胁持续增长。”?“这项法案将保护美国人的个人健康和遗传信息免受外国对手的侵害,这些对手有能力和动机利用这些信息来破坏我们的国家安全。美国Complete Genomics (CG) 公司成立于2005年,是全球首家提供人类基因组测序服务的生命科学公司。CG公司独有DNA纳米球(DNA nanoball,DNB) 芯片及组合探针错定连接 (combinatorialprobe anchor ligetion,cPAL) 这两种测序相关技术,2013年被华大基因收购,并成立华大智造公司单独发展基因测序仪,依托于相关技术,华大智造目前已经是国际主流基因测序仪供应商之一。[来源:仪器信息网] 未经授权不得转载[align=right][/align]
4月11日,在第89届中国国际医疗器械博览会(CMEF)上,华大智造重磅推出MGISEQ-2000RS FluoXpert多组学分析仪。这是中国首款具有自主知识产权、集成了病理染拍功能和测序功能的基因测序仪,一台能够做病理组织切片空间蛋白组学的测序仪,该设备将帮助用户解锁测序仪全新功能,更高效、更低成本地拥抱多组学浪潮。MGISEQ-2000RS FluoXpert将为用户提供在“测序模式”及“免疫荧光染色模式”两种模式中轻松切换的愉悦体验。针对不同批次的样本,用户可以在同一套设备平台上分别获得不同维度的研究所需的工具支撑;针对同一批次的样本,用户既可以根据基因测序获得初筛后的大量靶标数据,进行蛋白层面的检测和空间位置分析;又可以通过蛋白水平的分析结果,自行判断是否需要展开进一步的基因水平的研究。[align=center][img=640.jpg,500,333]https://img1.17img.cn/17img/images/202404/uepic/09a8fcb7-58f8-4c68-b34a-cc4237be4e8b.jpg[/img][/align][align=center]MGISEQ-2000RS FluoXpert[/align]DNBSEQ测序原理:当前,MGISEQ-2000系列测序仪主要应用领域多达20个以上,包括了WGS、空间组学、单细胞测序、肿瘤检测、病原快检、分子育种、肠道微生物等不同应用场景。它获得了90多个国家和地区的准入资质,在业界享有广泛赞誉,用户众多。截至目前,MGISEQ-2000已支持用户发表文章数量累计超过2500篇,10分以上文章超过200篇,总影响因子10,000+。[align=center][img=640 (1).jpg,500,333]https://img1.17img.cn/17img/images/202404/uepic/988aabb1-659b-46ed-a9d2-9fc7ab567db0.jpg[/img][/align][align=center]MGISEQ-2000RS FluoXpert的双载片平台[/align][b]MGISEQ-2000RS FluoXpert具备哪些优势特点[/b]从技术角度来说,全自动染拍一体的设计,极大地提高了操作效率;高效染色的生化试剂,进一步降低检测抗体用量;温和的洗脱过程使组织在经历多轮染色后仍可保留完整;先进的图像拼接算法,实现全片的无痕拼接。对于用户而言,MGISEQ-2000RS FluoXpert具备极高的易用性和灵活性。它采用了智能化的操作系统和友好的用户界面,研究人员能够轻松上手并快速完成实验操作。基于染拍一体化、系统化和模块化的设计理念,MGISEQ-2000RS FluoXpert通过操作软件实现对荧光成像技术和高分辨率扫描系统的模块化控制整合,从而实现对组织或细胞中蛋白质的高通量、高灵敏度检测。同时,它还具有极高的空间分辨率和定位精度,能够准确描绘出蛋白质在组织或细胞中的分布和变化。这样,研究人员能够更加深入地了解蛋白质在疾病发生和发展过程中的作用机制,为疾病研究提供更加精准的依据。FluoXpert可支持多种样本类型,能够满足不同研究项目的需求,兼容现有病理切片预处理流程,无需特殊样本预处理,手动操作步骤大幅减少,真正实现“样本进、图片出”的便捷体验。此外,该多组学分析的多重免疫荧光试剂盒为半开放式,兼容市售抗体,用户可根据项目需求自由组合抗体Panel即可开展蛋白组学分析;同时我们也支持定制化项目,充分满足不同研究领域的特殊需求。在数据结果产出上,MGISEQ-2000RS FluoXpert稳定可靠,其检测结果可对标金标准IHC染色结果,为科研工作者提供值得信赖的数据支持。多领域应用:加速推进精准医学发展MGISEQ-2000RS FluoXpert在基因组学领域以其卓越的性能和广泛的应用场景被冠以“全能王”称号。与常规的蛋白染拍一体机相比,MGISEQ-2000RS FluoXpert测序数据可以为染拍结果提供一步到位的分子检测数据作为补充或参考;与常规的基因测序仪相比,其测序性能优越,支持多种测序读长,通量灵活(55Gb-1440Gb),支持不同规格的测序载片独立运行,能全面满足广泛的测序需求,是国内外测序实验室的首选机型之一。通过对蛋白组学的拓展,MGISEQ-2000RS FluoXpert实现了基于同一套设备即可同时获得蛋白组学分析结果和基因测序下机数据,无需额外购买或操作多套不同设备,无需外送/等待时间,也无需复杂的样本前处理,支持全流程自动化。在肿瘤研究领域,多重免疫荧光能够与基因测序配合为疾病进行更精准地进行肿瘤分型、预后评估,为转化医学和肿瘤微环境研究提供有力支持。同时,MGISEQ-2000RS FluoXpert还能在病理研究中对切片进行高效、准确的分析,为病理学家提供更为详尽的细胞结构和组织信息。在药物发现和病人分组方面,FluoXpert同样能够发挥重要的工具支撑作用。它能够高效赋能研究人员揭示疾病的发病机制,为药物研发提供关键线索,并为病人实验分组提供科学依据,助力精准药物研发。此外,与传统免疫荧光组化相比,FluoXpert在临床研究方面也将具有一定优势。通过自动化、标准化的检测流程,它能够快速、准确地提供疾病相关的辅助信息,节约珍稀样本,为精准医学提供参考。MGISEQ-2000RS FluoXpert的推出,是华大智造在测序仪领域纵深布局的体现,在引领测序与免疫荧光技术的双重革新的同时,展现了模块化研发和多元化产品组合的无限可能。[来源:仪器信息网] 未经授权不得转载[align=right][/align]
[align=center][b][font=宋体]利用[/font][font='Times New Roman']MGI[/font][font=宋体]平台对大豆进行全基因组重测序分析[/font][/b][/align][b][font=宋体]摘要[/font][/b][font=宋体][font=宋体]:本研究建立了[/font][font=Times New Roman]MGI[/font][font=宋体]平台全基因重测序的方法。[/font][font=Times New Roman]MGI[/font][font=宋体]平台对大豆的全基因进行重测序结果显示,测序数据质量良好,且与参考基因组比对率较高,符合后续分析要求,对其进行[/font][font=Times New Roman]SNP[/font][font=宋体]和[/font][font=Times New Roman]Indel[/font][font=宋体]的变异检测和注释,此结果说明今后可利用[/font][font=Times New Roman]MGI[/font][font=宋体]平台对其它样品进行全基因重测序分析。[/font][/font][b][font=宋体]关键词[/font][/b][font=宋体][font=宋体]:[/font][font=Times New Roman]MGI[/font][font=宋体]平台;全基因重测序[/font][/font][align=center][font='Times New Roman']Whole genome resequencing analysis of soybeans using the MGI platform[/font][/align][b][font='Times New Roman']Abstract:[/font][font=宋体] [/font][/b][font=宋体][font=Times New Roman]In this study, a method for whole gene resequencing on the MGI platform was established. The results of resequencing the whole genes of soybean by MGI platform showed that the sequencing data was of good quality and had a high comparison rate with the reference genome, which met the requirements of subsequent analysis, and the variation detection and annotation of SNP and Indel were carried out, which indicated that the MGI platform could be used to perform whole gene resequencing analysis on other samples in the future.[/font][/font][b][font='Times New Roman']Keywords:[/font][font=宋体] [/font][/b][font=宋体][font=Times New Roman]MGI platform Whole gene resequencing[/font][/font][font='Times New Roman'] [/font][b][font='Times New Roman']1 [font=宋体]研究背景[/font][/font][/b][font='Times New Roman'][font=宋体]大豆是重要的粮食作物和油料作物,也是人类最主要的植物蛋白来源[/font][/font][font=宋体][font=Times New Roman][1][/font][/font][font=宋体][font=宋体]。我国是野生大豆的发源地,有着极其丰富的大豆种质资源基础,但是育种和产量较其他大豆主产国显得略有不足,究其原因是我国对大豆的研究和发掘力度存在不足,因此,对大豆育成品种的改良势在必行。自[/font][font=Times New Roman]2010[/font][font=宋体]年起,大豆群体水平的重测序也全面开展,在大豆的全基因组变异图谱上也得到了一定的研究进展[/font][/font][font=宋体][font=Times New Roman][2][/font][/font][font=宋体][font=宋体]。本研究利用[/font][font=Times New Roman]MGI[/font][font=宋体]平台对大豆全基因组进行重测序分析,挖掘全基因组水平上的突变。[/font][/font][b][font=宋体][font=Times New Roman]2 [/font][font=宋体]实验仪器[/font][/font][/b][font=宋体]主要实验仪器:[/font][font=宋体][font=Times New Roman]MGISP-960[/font][font=宋体]、[/font][font=Times New Roman]MGIDL-T7[/font][font=宋体]、[/font][font=Times New Roman]DNBSEQ-T7[/font][/font][b][font=宋体][font=Times New Roman]3 [/font][font=宋体]实验结果[/font][/font][font=宋体][font=Times New Roman]3.1 [/font][font=宋体]测序数据质量[/font][/font][/b][font=宋体][font=宋体]根据[/font][font=Times New Roman]MGI[/font][font=宋体]平台的测序特点,使用双端测序的数据,要求[/font][font=Times New Roman]Q30[/font][font=宋体]平均比例在[/font][font=Times New Roman]85%[/font][font=宋体]以上,可以看出大豆重测序数据[/font][font=Times New Roman]Q30[/font][font=宋体]平均比例在[/font][font=Times New Roman]94.72%[/font][font=宋体]以上,说明大豆测序数据质量良好,满足分析要求。[/font][/font][font='Times New Roman'] [/font][font='Times New Roman'] [/font][b][font=黑体][font=黑体]表[/font][font=Times New Roman]1 [/font][font=黑体]测序数据统计表[/font][/font][/b][table][tr][td][align=center][font='Times New Roman']Samples[/font][/align][/td][td][align=center][font='Times New Roman']ID[/font][/align][/td][td][align=center][font='Times New Roman']Clean reads[/font][/align][/td][td][align=center][font='Times New Roman']Clean bases[/font][/align][/td][td][align=center][font='Times New Roman']GC Content[/font][/align][/td][td][align=center][font='Times New Roman']%[/font][font=等线]≥[/font][font='Times New Roman']Q20[/font][/align][/td][td][align=center][font='Times New Roman']%[/font][font=等线]≥[/font][font='Times New Roman']Q30[/font][/align][/td][/tr][tr][td][align=center][font='Times New Roman']P117[/font][/align][/td][td][align=center][font='Times New Roman']P117[/font][/align][/td][td][align=center][font='Times New Roman']169494922[/font][/align][/td][td][align=center][font='Times New Roman']25424238300[/font][/align][/td][td][align=center][font='Times New Roman']36.18%[/font][/align][/td][td][align=center][font='Times New Roman']98.49%[/font][/align][/td][td][align=center][font='Times New Roman']95.27%[/font][/align][/td][/tr][tr][td][align=center][font='Times New Roman']P118[/font][/align][/td][td][align=center][font='Times New Roman']P118[/font][/align][/td][td][align=center][font='Times New Roman']166483906[/font][/align][/td][td][align=center][font='Times New Roman']24972585900[/font][/align][/td][td][align=center][font='Times New Roman']36.47%[/font][/align][/td][td][align=center][font='Times New Roman']98.61%[/font][/align][/td][td][align=center][font='Times New Roman']95.70%[/font][/align][/td][/tr][tr][td][align=center][font='Times New Roman']P119[/font][/align][/td][td][align=center][font='Times New Roman']P119[/font][/align][/td][td][align=center][font='Times New Roman']186127112[/font][/align][/td][td][align=center][font='Times New Roman']27919066800[/font][/align][/td][td][align=center][font='Times New Roman']35.89%[/font][/align][/td][td][align=center][font='Times New Roman']98.57%[/font][/align][/td][td][align=center][font='Times New Roman']95.61%[/font][/align][/td][/tr][tr][td][align=center][font='Times New Roman']P120[/font][/align][/td][td][align=center][font='Times New Roman']P120[/font][/align][/td][td][align=center][font='Times New Roman']192397276[/font][/align][/td][td][align=center][font='Times New Roman']28859591400[/font][/align][/td][td][align=center][font='Times New Roman']36.46%[/font][/align][/td][td][align=center][font='Times New Roman']98.22%[/font][/align][/td][td][align=center][font='Times New Roman']94.72%[/font][/align][/td][/tr][tr][td][align=center][font='Times New Roman']P198[/font][/align][/td][td][align=center][font='Times New Roman']P198[/font][/align][/td][td][align=center][font='Times New Roman']141636468[/font][/align][/td][td][align=center][font='Times New Roman']21245470200[/font][/align][/td][td][align=center][font='Times New Roman']37.11%[/font][/align][/td][td][align=center][font='Times New Roman']98.67%[/font][/align][/td][td][align=center][font='Times New Roman']95.84%[/font][/align][/td][/tr][tr][td][align=center][font='Times New Roman']P199[/font][/align][/td][td][align=center][font='Times New Roman']P199[/font][/align][/td][td][align=center][font='Times New Roman']169468714[/font][/align][/td][td][align=center][font='Times New Roman']25420307100[/font][/align][/td][td][align=center][font='Times New Roman']36.55%[/font][/align][/td][td][align=center][font='Times New Roman']98.60%[/font][/align][/td][td][align=center][font='Times New Roman']95.66%[/font][/align][/td][/tr][tr][td][align=center][font='Times New Roman']P200[/font][/align][/td][td][align=center][font='Times New Roman']P200[/font][/align][/td][td][align=center][font='Times New Roman']155078286[/font][/align][/td][td][align=center][font='Times New Roman']23261742900[/font][/align][/td][td][align=center][font='Times New Roman']37.90%[/font][/align][/td][td][align=center][font='Times New Roman']98.77%[/font][/align][/td][td][align=center][font='Times New Roman']96.14%[/font][/align][/td][/tr][/table][font=Calibri] [/font][font=宋体][font=宋体]样品原始数据碱基质量值可由图[/font][font=Times New Roman]1[/font][font=宋体]看出不存在异常碱基,[/font][font=Times New Roman]6[/font][font=宋体]个大豆碱基测序错误率分布均如图[/font][font=Times New Roman]1[/font][font=宋体]。[/font][/font][align=center][img=,321,]file:///C:/Users/xuxu/AppData/Local/Temp/ksohtml9716/wps1.jpg[/img][font=Calibri] [/font][/align][align=center][b][font=黑体][font=黑体]图[/font] [font=Times New Roman]1 [/font][font=黑体]碱基测序错误率分布图[/font][/font][/b][/align][font=宋体][font=宋体]碱基类型分布检查可用于检测有无[/font][font=Times New Roman]AT[/font][font=宋体]、[/font][font=Times New Roman]GC[/font][font=宋体]分离现象,若有碱基分离现象可能是测序或建库所带来的,并会影响后续分析。高通量所测序为基因组随即打断后的[/font][font=Times New Roman]DNA[/font][font=宋体]片段,由于位点在基因组上的分布是近似均匀的,同时,[/font][font=Times New Roman]G/C[/font][font=宋体]、[/font][font=Times New Roman]A/T[/font][font=宋体]含量也是近似均匀的。因此,根据大数定理,在每个测序循环上,[/font][font=Times New Roman]GC[/font][font=宋体]、[/font][font=Times New Roman]AT[/font][font=宋体]含量应当分别相等,且等于基因组的[/font][font=Times New Roman]GC[/font][font=宋体]、[/font][font=Times New Roman]AT[/font][font=宋体]含量。同样因为重叠等的关系会导致样品前几个碱基[/font][font=Times New Roman]AT[/font][font=宋体]、[/font][font=Times New Roman]GC[/font][font=宋体]不等波动较大,高于其他测序区段,而其它区段的[/font][font=Times New Roman]GC[/font][font=宋体]、[/font][font=Times New Roman]AT[/font][font=宋体]含量相等,且分布均匀无分离现象,如图[/font][font=Times New Roman]2[/font][font=宋体]所示。[/font][/font][align=center][img=,321,]file:///C:/Users/xuxu/AppData/Local/Temp/ksohtml9716/wps2.jpg[/img][font=Calibri] [/font][/align][b][font=黑体][font=黑体]图[/font][font=Times New Roman]2 ATGC[/font][font=黑体]含量分布图[/font][/font][font=宋体][font=Times New Roman]3.2 [/font][font=宋体]与参考基因组的序列比对[/font][/font][font='Times New Roman']3.2.1 [font=宋体]比对结果[/font][/font][/b][font=宋体][font=宋体]将测序得到的大豆样品与参考基因进行序列比对,[/font][font=Times New Roman]bwa[/font][font=宋体]软件主要用于二代高通量测序得到的短序列与参考基因组进行比对,比对结果见表[/font][font=Times New Roman]2[/font][font=宋体],根据比对结果可评估测序数据是否满足后续分析。[/font][/font][align=center][b][font=黑体][font=黑体]表[/font][font=Times New Roman]2 [/font][font=黑体]比对效率统计表[/font][/font][/b][/align][table][tr][td][align=center][font='Times New Roman']Sample_ID[/font][/align][/td][td][align=center][font='Times New Roman']Mapped(%)[/font][/align][/td][td][align=center][font='Times New Roman']Properly_mapped(%)[/font][/align][/td][td][align=center][font='Times New Roman']Averge_depth[/font][/align][/td][/tr][tr][td][align=center][font='Times New Roman']P117[/font][/align][/td][td][align=center][font='Times New Roman']99.99%[/font][/align][/td][td][align=center][font='Times New Roman']98.53%[/font][/align][/td][td][align=center][font='Times New Roman']25.44[/font][/align][/td][/tr][tr][td][align=center][font='Times New Roman']P118[/font][/align][/td][td][align=center][font='Times New Roman']99.99%[/font][/align][/td][td][align=center][font='Times New Roman']98.55%[/font][/align][/td][td][align=center][font='Times New Roman']24.9[/font][/align][/td][/tr][tr][td][align=center][font='Times New Roman']P119[/font][/align][/td][td][align=center][font='Times New Roman']99.99%[/font][/align][/td][td][align=center][font='Times New Roman']98.63%[/font][/align][/td][td][align=center][font='Times New Roman']27.75[/font][/align][/td][/tr][tr][td][align=center][font='Times New Roman']P120[/font][/align][/td][td][align=center][font='Times New Roman']99.98%[/font][/align][/td][td][align=center][font='Times New Roman']98.28%[/font][/align][/td][td][align=center][font='Times New Roman']28.58[/font][/align][/td][/tr][tr][td][align=center][font='Times New Roman']P198[/font][/align][/td][td][align=center][font='Times New Roman']99.99%[/font][/align][/td][td][align=center][font='Times New Roman']98.58%[/font][/align][/td][td][align=center][font='Times New Roman']21.26[/font][/align][/td][/tr][tr][td][align=center][font='Times New Roman']P199[/font][/align][/td][td][align=center][font='Times New Roman']99.98%[/font][/align][/td][td][align=center][font='Times New Roman']98.50%[/font][/align][/td][td][align=center][font='Times New Roman']25[/font][/align][/td][/tr][tr][td][align=center][font='Times New Roman']P200[/font][/align][/td][td][align=center][font='Times New Roman']99.99%[/font][/align][/td][td][align=center][font='Times New Roman']98.13%[/font][/align][/td][td][align=center][font='Times New Roman']23.13[/font][/align][/td][/tr][/table][font=宋体][font=宋体]将比对到不同染色体的[/font][font=Times New Roman]Reads[/font][font=宋体]进行位置分布统计,绘制[/font][font=Times New Roman]Mapped Reads[/font][font=宋体]在参考基因组上的覆盖深度分布图,见图[/font][font=Times New Roman]3[/font][font=宋体]。[/font][/font][align=center][img=,321,]file:///C:/Users/xuxu/AppData/Local/Temp/ksohtml9716/wps3.jpg[/img][font=Calibri] [/font][/align][align=center][b][font=黑体][font=黑体]图[/font][font=Times New Roman]3 Mapped Reads[/font][font=黑体]在参考基因组上的位置及覆盖深度分布图[/font][/font][/b][/align][font=宋体][font=宋体]统计[/font][font=Times New Roman]Mapped Reads[/font][font=宋体]在指定的参考基因组不同区域的数目,绘制基因组不同区域样品[/font][font=Times New Roman]Mapped Reads[/font][font=宋体]的分布图,见图[/font][font=Times New Roman]4[/font][/font][align=center][img=,321,]file:///C:/Users/xuxu/AppData/Local/Temp/ksohtml9716/wps4.jpg[/img][font=Calibri] [/font][/align][b][font=黑体][font=黑体]图[/font][font=Times New Roman]4 [/font][font=黑体]基因组不同区域[/font][font=Times New Roman]Reads[/font][font=黑体]分布图[/font][/font][font=宋体][font=Times New Roman]3.2.2 [/font][font=宋体]插入片段长度检验[/font][/font][/b][font=宋体][font=宋体]通过检测双端序列在参考基因组上的起止位置,可以得到样品[/font][font=Times New Roman]DNA[/font][font=宋体]打断后得到的测序片段的实际大小,即插入片段大小([/font][font=Times New Roman]Insert Size[/font][font=宋体]),它是信息分析时的一个重要参数。插入片段大小的分布一般符合正态分布,且只有一个单峰,[/font][font=Times New Roman]Insert Size[/font][font=宋体]分布图可以展示各个样品的插入片段的长度分布情况。各样品的插入片段长度模拟分布图见图[/font][font=Times New Roman]5[/font][font=宋体]。[/font][/font][align=center][img=,321,]file:///C:/Users/xuxu/AppData/Local/Temp/ksohtml9716/wps5.jpg[/img][font=Calibri] [/font][/align][align=center][b][font=黑体][font=黑体]图[/font][font=Times New Roman]5 [/font][font=黑体]插入片段长度模拟图[/font][/font][/b][/align][b][font=宋体][font=Times New Roman]3.2.3[/font][/font][font='Times New Roman'][font=宋体]深度分布统计图[/font][/font][/b][font='Times New Roman']Reads[font=宋体]定位到参考基因组后,可以统计参考基因组上碱基的覆盖情况。参考基因组上被[/font][font=Times New Roman]reads[/font][font=宋体]覆盖到的碱基数占基因组的百分比称为基因组覆盖度;碱基上覆盖的[/font][font=Times New Roman]reads[/font][font=宋体]数为覆盖深度。基因组覆盖度可以反映参考基因组上变异检测的完整性,覆盖到的区域越多,可以检测到的变异位点也越多。[/font][/font][font='Times New Roman'][font=宋体]覆盖度主要受测序深度以及样品与参考基因组亲缘关系远近的影响。基因组的覆盖深度会影响变异检测的准确性,在覆盖深度较高的区域(非重复序列区),变异检测的准确性也越高。[/font][/font][font='Times New Roman'][font=宋体]另外,若基因组上碱基的覆盖深度分布较均匀,也说明测序随机性较好。样品的碱基覆盖深度分布曲线和覆盖度分布曲线见图[/font][/font][font=宋体][font=Times New Roman]6[/font][font=宋体]。[/font][/font][align=center][img=,321,]file:///C:/Users/xuxu/AppData/Local/Temp/ksohtml9716/wps6.jpg[/img][font=Calibri] [/font][/align][align=center][b][font=黑体][font=黑体]图[/font] [font=Times New Roman]6 [/font][font=黑体]深度分布统计图[/font][/font][/b][/align][b][font=宋体][font=Times New Roman]3.3 [/font][font=宋体]变异检测[/font][/font][font=宋体][font=Times New Roman]3.3.1 SNP[/font][font=宋体]检测与注释[/font][/font][/b][font='Times New Roman'][font=宋体]根据变异位点在参考基因组上的位置以及参考基因组上的基因位置信息,可以得到变异位点在基因组发生的区域(基因间区、基因区或[/font]CDS[font=宋体]区等),以及变异产生的影响(同义非同义突变等)。软件可以使用[/font][font=Times New Roman]vcf[/font][font=宋体]格式文件作为输入和输[/font][/font][font=宋体][font=宋体]出,见图[/font][font=Times New Roman]7[/font][font=宋体]和图[/font][font=Times New Roman]8[/font][font=宋体]。[/font][/font][align=center][img=,321,]file:///C:/Users/xuxu/AppData/Local/Temp/ksohtml9716/wps7.jpg[/img][font=Calibri] [/font][/align][align=center][b][font=黑体][font=黑体]图[/font][font=Times New Roman]7 SNP[/font][font=黑体]突变类型分布图[/font][/font][/b][/align][align=center][img=,344,]file:///C:/Users/xuxu/AppData/Local/Temp/ksohtml9716/wps8.jpg[/img][font=Calibri] [/font][/align][b][font=黑体][font=黑体]图[/font][font=Times New Roman]8 SNP[/font][font=黑体]注释分类图[/font][/font][font=宋体][font=Times New Roman]3.3.2 Indel[/font][font=宋体]检测与注释[/font][/font][/b][font=宋体][font=宋体]根据所有样品在[/font][font=Times New Roman]CDS[/font][font=宋体]区和全基因范围的[/font][font=Times New Roman]Indel[/font][font=宋体]长度进行统计,其长度分布如图[/font][font=Times New Roman]9[/font][font=宋体]。[/font][/font][align=center][img=,355,]file:///C:/Users/xuxu/AppData/Local/Temp/ksohtml9716/wps9.jpg[/img][font=Calibri] [/font][/align][align=center][b][font=黑体][font=黑体]图[/font][font=Times New Roman]9 [/font][font=黑体]全基因和编码区[/font][font=Times New Roman]Indel[/font][font=黑体]长度分布图[/font][/font][/b][/align][font='Times New Roman'][font=宋体]根据样品检测得到的[/font]Ind[/font][font=宋体][font=Times New Roman]el[/font][/font][font='Times New Roman'][font=宋体]位点在参考基因组上的位置信息,对比参考基因组的基因、[/font]CDS[font=宋体]位置等信息,可以注释[/font][font=Times New Roman]Indel[/font][font=宋体]位点是否发生在基因间区、基因区或[/font][font=Times New Roman]CDS[/font][font=宋体]区、是否为移码突变等。发生移码突变的[/font][font=Times New Roman]Indel[/font][font=宋体]可能会导致基因功能的改变,具体注释结果见[/font][/font][font=宋体][font=宋体]图[/font][font=Times New Roman]10[/font][font=宋体]。[/font][/font][align=center][img=,344,]file:///C:/Users/xuxu/AppData/Local/Temp/ksohtml9716/wps10.jpg[/img][font=Calibri] [/font][/align][align=center][b][font=黑体][font=黑体]图[/font] [font=Times New Roman]10 Indel [/font][font=黑体]注释分类图[/font][/font][/b][/align][b][font=宋体][font=Times New Roman]4 [/font][font=宋体]结论[/font][/font][/b][font=宋体][font=宋体]本文基于[/font][font=Times New Roman]MGI[/font][font=宋体]对大豆进行重基因测序,实验结果可看出,大豆样品测序产出数据良好,与参考基因组序列比对率较高,符合后续分析,对其进行变异检测可得到[/font][font=Times New Roman]SNP[/font][font=宋体]和[/font][font=Times New Roman]Indel[/font][font=宋体]的结果。其它研究表明[/font][/font][font=宋体][font=Times New Roman]MGISEQ-2000[/font][font=宋体]全基因组重测序表现性能稳定、质量可靠,在实际应用上有明显的优势和应用价值[/font][font=Times New Roman][3][/font][font=宋体]。对[/font][/font][font=宋体][font=宋体]本次实验说明[/font][font=Times New Roman]MGI[/font][font=宋体]平台对样品进行重测序效果良好,后续可对其它植物进行重测序。[/font][/font][font=宋体] [/font][font=宋体] [/font][font=宋体]参考文献:[/font][font=宋体][font=Calibri][1] [/font][/font][font='Times New Roman'][font=宋体]张永芳[/font],[font=宋体]钱肖娜[/font][font=Times New Roman],[/font][font=宋体]王润梅[/font][/font][font=宋体][font=Times New Roman],[/font][font=宋体]等[/font][font=Times New Roman]. [/font][font=宋体]不同大豆材料的抗旱性鉴定及耐旱品种筛选[/font][font=Times New Roman][J].[/font][font=宋体]作物杂志[/font][font=Times New Roman],2019(5): 41-45.[/font][/font][font=宋体][font=Calibri][2] [/font][font=宋体]邬启帆[/font][font=Calibri]. [/font][font=宋体]基于基因组重测序黄淮海大豆育成品种遗传结构及重要家族遗传基础研究[/font][font=Calibri][D]. [/font][font=宋体]南昌[/font][/font][font=宋体][font=宋体]大学[/font][font=Times New Roman], 2023.[/font][/font][font=宋体][font=Calibri][3] [/font][/font][font=宋体][font=宋体]李伟宁[/font][font=Times New Roman],[/font][font=宋体]刘刚[/font][font=Times New Roman],[/font][font=宋体]周荣等[/font][font=Times New Roman]. MGISEQ-2000[/font][font=宋体]、[/font][font=Times New Roman]HiSeq 2000[/font][font=宋体]与[/font][font=Times New Roman]NovaSeq 6000[/font][font=宋体]平台全基因组重测序数据的比较分析[/font][font=Times New Roman][J]. [/font][font=宋体]中国畜牧杂志[/font][font=Times New Roman],2021,57(11):156-162.[/font][/font]
近日,深圳华大基因研究院和美国科学家共同发起“共生体基因组计划”。该计划将对海蛤蝓(又称绿叶海蜗牛)及藻类饵料进行基因组测序。有科学家认为,海蛤蝓可能是“生命之树”中动植物界的交叉点。海蛤蝓的细胞能够从藻类获取叶绿素,进行光合作用,从而为其所有生命活动提供足够的能量,包括繁殖。迄今为止,科学家在海蛤蝓基因组里发现了大约十多种藻类基因,这些基因使这种生物在叶绿素合成通道和碳固定循环中具有集光蛋白质和酶类的功能。随着研究的深入,不断有新的藻类基因在海蛤蝓基因组中被发现。海蛤蝓通过自身内被转移的藻类基因合成叶绿素,进行光合作用。这种神奇的共生现象第一次证明了一套完整的生物合成途径可以从一种多细胞生物传递到另一种多细胞生物。华大基因有关专家表示,通过对藻类和海蛤蝓的基因组进行比较,不仅将在宿主细胞中发现一组能够进行持续光合作用的基因,而且能够找到转移的特性,包括转移基因片段的大小、数量;更重要的是了解这种转移的运行机制。这些发现将对基因组的人工调控和基因治疗新技术的开发产生重大现实意义。此外,这两类生物的基因组测序将有利于比较基因组研究、进化规则、发展生物学及分类学的发展。据悉,这次联合研究是华大基因“千种动植物参考基因组计划”的一部分。该计划将在未来两年内建立1000种动植物的参考基因组序列。在“共生体基因组计划”中,华大基因主要负责测序和生物信息分析工作。《科学时报》 (2010-3-23 A1 要闻)
各位仁兄:有谁知道心电电极片的大致工艺?求助大家了!或者有谁知道锡/锡合金电极的制造方法吗?
浙大智达色谱工作站怎样修改以前的试验结果
实验室中与上司相处有六大智慧1、虚心求教。不懂的要多请教,让上司知道你是虚心学习的人。2、交流简洁。时间是管理者最宝贵的财富。3、真心仰慕。适度的表示赞美与认同。4、进退得宜。以免引起公司的蜚短流长。5、适度恭维。6、热切交流但不忘分寸。
刚买了个盖碗泡茶,结果到货以后试用,发现水往外倒的时候都顺着外壁流下去了,很难倒到小杯子里~后来咨询了卖家,说是要分杯的话要用公杯,这个盖碗就是一个人用的~好奇心就被勾起来了,研究了盖碗和公杯的区别,ms公杯的碗口处倾斜角度更大,更便于液体流出~还记得以前有个客户,需要一大批烧杯,指明要进口的,原因就是进口的烧杯倾倒口做的比国产的好,液体倒出来的时候不容易流到外壁~其实就是一个很小的区别,但对于使用者来说就是不同的使用体验;同样的,也是这么个小区别,对于生产者的要求也很高,要考虑到方方面面的因素~所以即使是小东西,要做好也是很难的,需要大智慧啊,大家有没有类似的经历呢~
[align=center][img=initpintu_副本.jpg]https://img1.17img.cn/17img/images/202401/uepic/f5adec08-16f3-46b6-b76f-d78cbdc0c884.jpg[/img][/align][align=center][size=14px][color=#7f7f7f][i]△ 刘忠范院士出席HORIBA前沿应用开发中心(ASP)的开幕典礼并致辞,同时为战略合作伙伴揭牌[/i][/color][/size][/align]2023年4月,北京石墨烯研究院(Beijing Graphene Institute, 简称BGI)与HORIBA前沿应用开发中心(Analytical Solution Plaza, 简称ASP)正式达成战略合作,双方依托BGI的尖端产研技术与HORIBA在分析检测领域的专业优势,合作建立“前沿技术创新中心”(简称创新中心),旨在共同推进石墨烯产业的基础研究与产业化核心技术开发,实现双方在产品解决方案共建、技术革新和市场机会探索方面互利共赢。 [align=center][img=图片4.png,600,444]https://img1.17img.cn/17img/images/202401/uepic/eda2b34d-f583-4fb1-bcc6-6887374c840f.jpg[/img][/align][align=center][size=14px][color=#7f7f7f][i]△ ASP门廊处悬挂的前沿技术创新中心铭牌[/i][/color][/size][font=等线][size=14px] [/size][/font][/align]BGI是由北京市政府批准、北京大学牵头建设的新型研发机构,刘忠范院士担任创始院长。截至2023年12月,BGI人员规模超过400人,是一家集新材料研发、生产,及装备制造于一身的石墨烯高新技术企业。正因其独特的“全链路”模式,BGI的多个业务环节都离不开先进的光谱分析检测技术。[align=center][img=图片6.png,600,450]https://img1.17img.cn/17img/images/202401/uepic/23cb1a81-9564-4672-9607-321dd3a040b5.jpg[/img][/align][align=center][size=14px][i][color=#7f7f7f]△ 位于BGI实验室的HORIBA LabRAM HR Evolution拉曼光谱仪[/color][/i][/size][/align][align=center][size=14px][i][color=#7f7f7f](现已升级为 HORIBA LabRAM Odyssey高速高分辨显微共焦拉曼光谱仪)[/color][/i][/size][/align]ASP内设有多台先进的光学光谱分析测试仪器,这些仪器为石墨烯研究提供了极大的便利与诸多可能性,如:CN-300离心式纳米粒度分析仪,其对大小颗粒的超高分辨率可以帮助研究人员了解各种碳材料的分散状态;LabRAM Odyssey高速高分辨显微共焦拉曼光谱仪具备超低波数和12寸晶圆检测能力,能满足石墨烯层间堆垛方式及大晶圆的检测需求。而XploRA PLUS高性能全自动拉曼光谱仪联用AFM,则不仅可以获取同区域的化学和物理信息,还可以实现纳米级空间分辨率的Raman/PL检测。在创新中心,双方专家可以深度交流沟通,使BGI强大的研发实力与HORIBA前沿光谱分析技术有机结合,加速创新成果产出。目前,利用显微拉曼光谱技术,BGI与HORIBA已建立了对石墨烯薄膜和单晶石墨烯晶圆系列产品的快速检测方法,为石墨烯的产品质控保驾护航。未来,双方将继续努力,力图打破传统手段对片层样品尺寸分析的局限性,将新一代颗粒分析技术与粉体石墨烯尺寸及层数研究相结合,建立全新的分析解决方案,进一步实现优势技术互补,为我国石墨烯产业发展带来突破性的创新成果。[align=center][img=图片1.png]https://img1.17img.cn/17img/images/202401/uepic/d8f94426-67d1-49f3-ab1a-981fa7d22b2b.jpg[/img][/align][align=center][size=14px][color=#7f7f7f][i]△ ASP中设有多台先进的分析测试仪器,可充分匹配石墨烯相关研究测试需求[/i][/color][/size][/align]唯有内外兼修方能制胜有道。在双方内部实现技术联合后,如何面向外部实现市场价值是创新中心的另一探索重任。作为新兴“石墨烯基材料”产业孵化器,BGI提供了面向广大中小企业的“研发代工”平台,针对企业的需求,开展“定制化”研发工作,以解决企业研发力量不足的现实问题。针对在此过程中产生的新需求,HORIBA将予以光学光谱检测技术的支持。同时,通过BGI“研发代工”平台赋予的触点,HORIBA ASP还可以与各中小企业建立联系,探讨潜在的机会点,这样可以为企业在行业内的提前布局、开拓市场及扩大影响力方面打下基础,让未来发展更加清晰、美好,真正实现互利共赢。合作发展并济,价值创新共融。BGI与HORIBA ASP共建前沿技术创新中心,是科技领域内企业强强联合的又一标志。双方将充分发挥各自的优势,不断探索创新合作模式,主动作为,彼此融入,同频共振,为中国成为引领世界的石墨烯新材料研发高地和创新创业基地作出卓越贡献。[来源:HORIBA][align=right][/align]
用浙大智达出的N2000色谱数据工作站在线分析完成后停止采集要保存时页面自动就关了,数据保存不上怎么回事?
有谁知道浙大智达N3000工作站(就是采集卡)的价格?谢谢各位大虾了。
大家好,有谁知道浙大智达N2000 v3.3色谱工作站的安装系列号?我们的那个弄丢了,需要重装工作站,急需!
最近想做一个设备,类似于压汞仪,但是无论是麦克的压汞仪还是康塔的压汞仪都满足不了要求,主要是样品大小限制。另外设备自动化程度过高,有一些参数设置比如实验时间设置不能满足要求。在此求可以生产国产压汞仪的产家,或有汞设备制造经验的个人或产家,要求按我们的要求非标制造一台类似的设备。在网上搜过相关的设备,发现有国产的,手动压汞仪(国产) --CN61M/YGY-J -北京中西远大科技有限公司但发现中西远大只是代理,在国内即没有产品可以看,也不告诉我产家。在此鄙视一下国内的某些代理公司。希望知道相关信息的人联系我,非常感谢!有相关信息可以给我发邮件或站内另注:实际上我要实现的功能非常简单,软件硬件的要求都不高,原理大家都知道,但我需要有汞设备制造经验的人帮我实现。
浙大智达N2010软件是不是可以兼容旧版N2000的工作站?N2000的积分处理手段太少且不好用,N2010在这一方面有没有改善?
近日,深圳华大基因研究院宣布,我国科学家将参与全球最大微生物基因组研究项目,对来自全球的20万个样本进行环境DNA测序或宏基因组测序,从而建立一个全球性的基因图谱,并承担核心工作。该项目旨在全方位、系统性研究全球范围内微生物群落功能及进化多样性,以便更好地造福社会及人类。与以往的微生物研究有所不同,该项目的研究对象不仅集中于海洋和人体环境中微生物群落,还包括土壤、空气、淡水生态系统等整个地球表面的绝大多数的微生物群落。华大基因将负责亚洲地区所有样本的收集和鉴定,并对整个项目提供DNA提取、扩增、建库、宏基因组测序以及研发生物信息学分析流程所需的计算资源。这些信息学分析流程将为项目研究产生的海量数据提供一个分析框架。项目负责人、芝加哥大学和阿贡国家实验室的教授杰克·吉尔伯特博士表示:“华大基因在测序能力、测序技术和信息分析等方面已展现出卓越的能力。此项目是一个前所未有的最大的基因组测序项目,作为全球最大基因组学研究中心,华大基因的参与至关重要。”华大基因理事长杨焕明院士表示,微生物对地球上所有的生命具有至关重要的作用,而我们对微生物的复杂性和多样性认识不足,征服这个未知的领域非常有必要。华大基因拥有国际先进水平的测序平台和强大的生物信息学分析能力,可以为促进人类对微生物群落重要性的了解贡献力量。(来源:科技日报)
2009年12月22日,加拿大竞争局发布了“加拿大产品”(Products of Canada)和“加拿大制造”(Made in Canada)的非食品产品的新标签准则。根据该准则,从2010年7月1日开始,加拿大竞争局将对标注“加拿大产品”和“加拿大制造”的非食品产品实施新的更严格的标签要求。由于这种标签向消费者传达了该产品是在加拿大制造的信息,新指南通过规定以下要求澄清了如何适当地使用该标签:产品必须在加拿大进行最后实质性转变; 声称“加拿大产品”的产品必须有98%在加拿大直接制造或生产; 声称“加拿大制造”的产品将仍然为51%在加拿大直接制造或生产,并随附一个表明该产品含有进口成分的合格声明。 加拿大竞争局已于2009年秋季对本准则草案举行了公众咨询会,并在起草最终准则时考虑了咨询会上收集的意见。该文件将取代先前的“加拿大制造”声明产品准则。新准则将于2010年7月1日生效,在这之间的过渡期,加拿大竞争局仍按照原先的准则进行。 加拿大竞争局是一个独立的执法机构,其目的在于通过保护和促进竞争市场、让消费者获得知情选择来促进加拿大的繁荣。该局主要负责《消费品包装与标签法案》(the Consumer Packaging and Labelling Act)、《竞争法案》(the Competition Act)和《纺织品标签法案》(the Textile Labelling Act)和《贵金属标记法案》(the Precious Metals Marking Act)的实施。虽然这些法律并没有明确要求在产品上识别其原产国,它们包含有禁止虚假或误导性表述的条款。加拿大竞争局将这些准则适用于原产国声明以确定该声明是否符合,并根据相关法律的虚假或误导性表述条款实施相应行动。 详情参见以下 “加拿大产品”和“加拿大制造”非食品产品的新标签准则的链接:[URL=http://www.ic.gc.ca/eic/site/cb-bc.nsf/vwapj/Dec09_MadeinCanada-e.pdf/$FILE/Dec09_MadeinCanada-e.pdf]Enforcement Guidelines – “Product of Canada” and “Made in Canada” Claims [/URL]
基本原理蛋白质与染料考马斯亮蓝G—250结合,使得染料最大吸收峰从465nm变为595nm,在一定的线性范围内,反应液595nm处吸光度的变化量与反应蛋白量成正比,测定595nm处吸光度的增加即可进行蛋白定量。试剂与器材一、 试剂1. 试剂A:考马斯亮蓝G—250原液称取100mg考马斯亮蓝G—250溶于50mL 95%的乙醇,加入100mL 85%(W/V)磷酸。2. 试剂B:0.01%考马斯亮蓝G—250测定工作液将考马斯亮蓝G—250原液与双蒸水按15:85比例混匀,过滤。4℃,棕色瓶保存。3.[size=9.
世界生物公司介绍Abcam | Abnova | American Qualex | Ambion | Amresco | ATCC | Axygen | Geijing Genomics Institute | Bender MedSystems | Biodesign | Biosource | Biotrend | Diagnostic BioSystems | Biovision | Endogen | Eppendorf | FALCON | Genesis | GeneTex | HyCult Biotechnology | ImmunoStep | Invitrogen | MASE | MI | MP | NBS | New England Biolabs(NEB) | Novagen | Pharmengen | PAA | QIAGEN | Rapidbio(RB) | RECI | R&D | Roche | Santa Cruz | Sigma | SPINREACT | Strategene | Thermo | TPI | TPP | TOYOBO | U-gene | Upstate | Viagene | Wathman | Wheaton | ZEUSAbcam Abcam公司位于英国的剑桥科学园,成立于1998年. 是著名的抗体生产厂家,提供近4000种一级抗体,500种二级抗体,400余种蛋白质及多肽,以及各类ELISA检测试剂盒。Abnova Abnova是我国台湾最大的生物公司之一,可以自主生产多达12000余种自产抗原(包括3千余种全长蛋白),和大量的多克隆和单克隆抗体。American Qualex American Qualex总部位于美国,专业提供第二抗体、标记试剂盒和小鼠单克隆抗体分型试剂盒。还可以提供多肽合成、制备抗体、纯化及标记。 Ambion 专门致力于 RNA 相关产品的研究,主要提供 RNAi 研究、体外表达、芯片分析等 RNA 实验所用试剂。其 RNAi 产品在业界很受欢迎。Amresco 其生产的生化试剂质量高,价格低廉。在分子生物学领域被广泛使用。 ATCC 美国ATCC菌种保藏中心,又称美国模式菌种收集中心,座落于马里兰洲洛克菲勒的一家私营的,非赢利性组织。目前它可以提供以下物品:细胞系(3000种);细菌和噬菌体(15000种);动植物病毒(2500种);原生动物 1200种以及重组物品等。 Axygen Axygen 产品经美国 FDA 认证无 RNA 酶、 DNA 酶、无热源,提供各种规格的枪头,离心管, PCR 管等。 北京华大基因 北京华大基因研究中心,成立于一九九九年九月,为高新技术企业。 几年来,北京华大基因研究中心与北京基因组所一起成功地完成了1%国际人类基因组计划、中国超级杂交水稻全基因组框架图及精细图、家猪基因组研究、国际鸡基因组计划、家蚕基因组计划、抗SARS研究、海啸遇难者DNA检测等多项大型科研项目,数十篇学术论文发表在Science、Nature、PLoS Biology、Nucleic Acids Research等世界顶级科学杂志上。党和国家领导人胡哥、路甬祥、陈至立、贾庆林、刘淇等曾亲临视察,江泽民、温家宝、朱榕基曾给予高度评价。 Bender MedSystems 产品涉及免疫与细胞生物学、肿瘤生物学等领域的各类研究产品。 Biodesign Biodesign是一家行业领先的单克隆抗体制造商,同时提供纯化及重组蛋白、配套检测试剂、合成多肽等服务,在世界范围内有多达50个国家的地区代理商。 Biosource 专著于细胞因子和相关活性蛋白的检测及表达。 [/si
[size=3] 中国和沙特阿拉伯研究人员9日在此间宣布,他们已成功绘制出阿拉伯骆驼的基因图谱,这是世界上首次完成阿拉伯骆驼基因破译。 沙特农业大臣法赫德在此间举行的新闻发布会上说,这项具有世界水平和重大意义的研究成果是来自沙特和中国深圳华大基因研究院的20多位研究人员耗时半年完成的。它将有助于对这种抗高温和耐干旱的骆驼的基因改良、防病等。 深圳华大基因研究院院长汪建表示,今后中国研究人员将与沙特同行继续对骆驼开展深入研究,同时将研究范围扩展到在极端条件下生存的其他生物。 [/size]
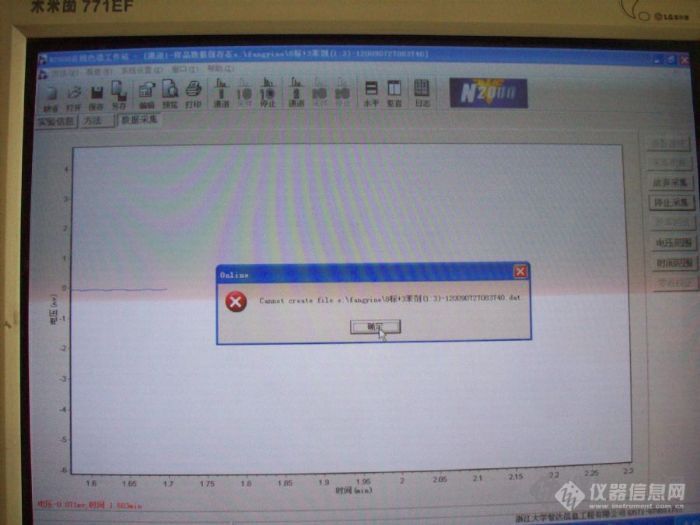
[color=#DC143C]我用得是浙大智达N2000的工作站,FID检测的,以前保存数据都很顺利,不知道为什么这两天我的数据及色谱图不能在工作站上保存调试了一下也不管用实验正在进行中,急候答复[em09508][/color][img]http://ng1.17img.cn/bbsfiles/images/2009/07/200907270848_161824_1609012_3.jpg[/img][img]http://ng1.17img.cn/bbsfiles/images/2009/07/200907270849_161825_1609012_3.jpg[/img]
[font=system-ui, -apple-system, BlinkMacSystemFont, &][size=18px]日前,在2022年机器人世界杯救援机器人比赛中,国防科大智能科学院机器人研究中心的NuBot代表队夺得两项世界冠军。[/size][/font]
基因组测序和序列的组装,为快速研究该致病菌株的致病机理创造了条件。与此同时华大基因与德国汉堡-Eppendorf医疗中心合作,也宣布完成了对致病菌株的测序工作。Guenther说:"在有限的时间里完成了对微生物的全基因组测序,极大的方便了研究者从一个整体的水平上去研究微生物,进而揭示在这些目标微生物的基因组究竟发生了哪些改变。"事实上也的确如此,科学家根据从基因组测序的数据所获得的证据,将本次的致病型大肠杆菌鉴定为致病型大肠杆菌的一个新杂交品种,并且携带了一些抗性基因。"从宏观的基因组水平上来研究这类细菌,将在很大程度上革新我们对传染病暴发的认识,3-4天内完成对某种微生物的全基因组测序及基因标注,将会开启一个新的研究领域。"在新奥尔良召开的美国微生物学会年度会议上,一些研究者指出,分子鉴定的方法正被用来打造基因组传染病学这一领域,基因组传染病学致力于重构传染病暴发的过程,以求在将来能够对传染病能进行实时有效的监控和快速反应。
因为是在一欠发达地区的小地市所工作,还没有通过CNAS认可,我们所正在努力完善一些硬件条件,继而想申请CNAS认可,所以想请有关专家介绍一下先在CNAS认可的大致程序。 我只听说校准项目,之前是不应通过JJF1033计量标准考核的,而现在是要通过JJF1033计量标准考核了。在[b]广东省计量院顺利通过CNAS能力验证提供者初次评审[/b]的帖子里,有幸得到[url=https://bbs.instrument.com.cn/user.asp?username=mm_msn]玉树临风[/url]老师指导才让我知道,CNAS认可还有能力验证的环节,所以很想知道CNAS认可的大致程序。特别是对于校准项目的CNAS认可,能力验证与通过JJF1033计量标准考核关系又怎样?恳请赐教哦!
有人清楚antek9000大致价格吗
那位大侠可以告诉我一下TA公司的高温DSC仪的技术指标和大致价格?我们实验室要买,但是需要知道大致价格!谢谢!
请问进行自动调谐的大致时间是多少分钟?假如一次调谐后在进行二次调谐,时间会不会更短些呢?
目前直径小的焊丝可以直接用剪刀剪成小粒,可是大直径的焊条、焊丝如何制样(化学法分析),用什么设备。
现在的仪器型号是:GCMS-2010 PLUS,单EI,想配个CI,大致需多少钱?都有哪些品牌可供选择?
向大家请教一下"帖烯"类物质的概念,和该类物质的大致分析方法,有经验的朋友请指点一下.
总做液相的人也许会遇到燥声大的情况,那么当燥声大时应该从以下几点考虑: 1 氘灯损坏, 2 检测池污染, 3 检测池有气泡, 4 是否接地线, 5 压力大致使镜片压坏,6 参比池污染,此时应该清洗残留液 7 检测池漏液原因很多啊,根据自己的实际情况排查一下吧http://simg.instrument.com.cn/bbs/images/brow/emyc1010.gif