基因定点突变,顾名思义,就是把目的基因上的一个碱基有目的的替换为另一个碱基。体外定点突变时目前分子生物学领域中一种快速有效地手段。产生定点突变操作及所需仪器简单,随着技术的发展,方法也越来越快捷简便。定点突变除了可以生成点突变,多点突变外,还可以人为产生碱基删除和添加等。体外基因定点突变是实验室中优化改造基因,研究基因功能的常用方法之一。通过改造基因序列,可以对相应氨基酸序列进行改变,进而影响蛋白质的结构和功能,探讨突变氨基酸位点在蛋白结构和功能中所起的作用。在我多年的实验中,体外基因定点突变是其中一项重要的内容。在这里,我与大家分享一下我的实验方法和心得体会。多年来,我所采用的基因定点突变的方法主要有三种,这可能也包括了目前所有的定点突变的方法。基因定点突变使用的主要仪器就是PCR扩增仪(biometra),而这是每一个分子生物学实验室的必需品,也就是大家吃饭的家伙。下面从繁到简对我所采用的突变方法进行一下简单描述。http://ng1.17img.cn/bbsfiles/images/2012/12/201212032022_409068_1306303_3.jpg我所使用过的定点突变方法简单的说就是三步,两步和一步PCR法。三步PCR法是我早期在实验室使用的定点突变方法。利用三步PCR法构建一个点突变,需要4条PCR引物:L1,L2,R1和R2。L1和R1是目的基因5‘和3‘端的引物,分别包含起始和终止密码子。突变点设计在引物L2和R2的正中间位置,L2和R2是完全互补的两条PCR引物,长度一般在28-40 bp之间,推荐长度31-35 bp,这样可以保证突变的两边有效搭在一起。http://ng1.17img.cn/bbsfiles/images/2012/12/201212032026_409072_1306303_3.jpg三轮PCR均采用常规PCR反应条件,第一轮分别以L1和R2,L2和R1扩增获得目的片段L和R。第二轮PCR,不需要引物,仅加入模板L和R各100 nmol,进行约20 轮的PCR反应,获得产物m1。第三轮PCR以m1为模板,L1和R1为引物进行PCR扩增。最终得到目的产物m。得到的PCR产物进行克隆转化得到进一步扩增,便获得了突变的目的基因。在随后的实验中,我将三步PCR法简化为两步法PCR。两步法PCR和三步法PCR一样,需要引物四条,引物设计也与三步法相同,只是在第一轮PCR获得产物L和R之后不再单独进行第二轮PCR,而直接在PCR体系中加入等mol的产物L和R
简介:污染物对人体的潜在危害,引起人们的普遍关注。世界上已发展了百余种短期快速测试法,检测污染物的遗传毒性效应。B.N.Ames等经十余年努力,于1975年建立并不断发展完善的沙门氏菌回复突变试验(亦称Ames试验)已被世界各国广为采用。该法比较快速、简便、敏感、经济,且适用于测试混合物,反映多种污染物的综合效应。众多学者有的用Ames试验检测食品添加剂、化妆品等的致突变性,由此推测其致癌性;有的用Ames试验检测水源水和饮用水的致突变性,探索较现行方法更加卫生安全的消毒措施;或检测城市污水和工业废水的致突变性,结合化学分析,追踪污染源,为研究防治对策提供依据;有的检测土壤、污泥、工业废渣堆肥、废物灰烬的致突变性,以防止维系生命的土壤受致突变物污染后,通过农作物危害人类;检测气态污染物的致突变性,防止污染物经由大气,通过呼吸对人体发生潜在危害;用Ames试验研究化合物结构与致变性的关系,为合成对环境无潜在危害的新化合物提供理论依据;检测农药在微生物降解前后的致突变性,了解农药在施用后代谢过程中对人类有无隐患;还有用Ames试验筛选抗突变物,研究开发新的抗癌药等等。 关键字:突变性检测,Ames试验一、目的和原理 鼠伤寒沙门氏菌(Salmonella typhimurium)的组氨酸营养缺陷型(his-)菌株,在含微量组氨酸的培养基中,除极少数自发回复突变的细胞外,一般只能分裂几次,形成在显微镜下才能见到的微菌落。受诱变剂作用后,大量细胞发生回复突变,自行合成组氨酸,发育成肉眼可见的菌落。某些化学物质需经代谢活化才有致变作用,在测试系统中加入哺乳动物微粒体酶①,可弥补体外试验缺乏代谢活化系统之不足。鉴于化学物质的致突变作用与致癌作用之间密切相关,故此法现广泛应用于致癌物的筛选。 二、步骤和方法 Ames试验的常规方法有斑点试验和平板掺入试验。 1.菌株鉴定 用于测试的菌株,需经基因型和生物学性状鉴定,符合要求才能投入使用。 目前推荐使用的一套菌株是TA97、TA98、TA100和TA102。鉴定前先进行增菌培养。为鉴定结果可靠,需同时培养野生型TV菌株,作为测试菌基因型之对照。增菌培养用牛肉膏蛋白胨液体培养基,接种后于37℃,100r/min振荡培养12h左右,细菌生长相为对数期末,含菌数应为1×109个/ml~2×109个/ml。 鉴定项目: (1)脂多糖屏障丢失(rfa)用接种环或一端挠起的接种针以无菌操作术取各菌株的增菌培养液,在营养琼脂平板上分别划平行线,然后用灭菌尖头镊夹取灭菌滤纸条,浸湿结晶紫溶液,贴放在平板上与各接种平行线垂直相交。盖好皿盖后倒置于37t温箱,培养24h后观察结果。 (2)R因子 划线接种,贴放滤纸条及培养等均同上,唯滤纸条浸湿的药液不同,为氨苄青霉素钠溶液。TA102除pKM101外,还有pAQ1,载有抗四环素的基因,故另用滤纸条浸湿四环素溶液后贴放于划线接种的平板上。 (3)紫外线损伤修复缺陷(△uvrB)在营养琼脂平板上按上述方法划线接种后,一半接种线用黑玻璃遮盖,另一半暴露于紫外光下8sec,然后盖好皿盖并用黑纸包裹平皿,防止可见光修复作用。培养同上。 (4)自发回变 预先制备底平板;向灭菌并在水浴内保温45℃的上层软琼脂中注入0.1ml菌液;混匀后倾于底平板上并铺平。平皿倒置于37℃温箱培养48h。 (5)回变特性——诊断性试验 上层软琼脂中除菌液外,还注入已知阳性物之溶液,需活化系统者并加入S9mix,余同上。 组氨酸营养缺陷型由自发回变即可知。 2.斑点试验 吸取测试菌增菌培养后的菌液0.1ml,注入融化并保温45℃左右的上层软琼脂中,需S9活化的再加0.3ml-0.4mlS9mix,立即混匀,倾于底平板上,铺平冷凝。用灭菌尖头镊夹灭菌圆滤纸片边缘,纸片浸湿受试物溶液,或直接取固态受试物,贴放于上层培养基的表面。同时做溶剂对照和阳性对照,分别贴放于平板上相应位置。平皿倒置于37℃温箱培养48h。在纸片外围长出密集菌落圈,为阳性;菌落散布,密度与自发回变相似,为阴性。 3.平板掺入试验 将一定量样液和0.1ml测试菌液均加入上层软琼脂中,需代谢活化的再加0.3ml-0.4ml S9mix,混匀后迅速倾于底平板上铺平冷凝。同时做阴性和阳性对照,每种处理做3个平行。试样通常设4个~5个剂量。选择剂量范围开始应大些,有阳性或可疑阳性结果时,再在较窄的剂量范围内确定剂量反应关系。培养同上。同一剂量各皿回变菌落均数与各阴性对照皿自发回变菌落均数之比,为致变比(MR)。MR值≥2,且有剂量-反应关系,背景正常,则判为致突变阳性。 三、应用 1.斑点试验只局限于能在琼脂上扩散的化学物质,大多数多环芳烃和难溶于水的化学物质均不适宜用此法。此法敏感性较差,主要是一种定性试验,适用于快速筛选大量受试化合物。 2.平板掺入试验可定量测试样品致突变性的强弱。此法较斑点试验敏感,获阳性结果所需的剂量较低。点试获阳性结果的浓度用于掺入试验(每皿0.1ml),往往出现抑(杀)菌作用。 3.致变作用迟缓或有抑菌作用的试样,培养时间延长至72h。 4.挥发性的液体和气体试样,可用干燥器内试验法进行测试。 5.目前,Ames试验作为检测环境诱变剂的一组试验中的首选试验,广泛应用于致突变化学物的初筛。但是,与今天快信息、高效率的社会要求相比,该试验程序还嫌繁琐,方法不够简便,有待于创建新的更为快速灵敏、简单易行的环境诱变剂短期生物测试法。
8 kb)的原理我上面已经说了,只是补充了一些我认为的注意事项。如果你更有钱的话,那么你可以叫其它公司帮你做定点突变服务,大约是改一个点1000元左右。
英国变异毒株杀入广东!粤港澳大湾区是一体,11个城市600万平方公里,七千万人口,是中国对开放的前沿阵地,与英美来往密切,大湾区的朋友们一定要警惕!这个变异毒株,重点不在毒,在“变”!感染性极强,遑论大人,过去不易感的儿童都会超大概率中招!?根据广东疾控中心通报,英国留学生12月4日入境广州,检测是阴性,隔离14后检测为阴阳性!这个变异病毒潜伏期这么长,感染性这么强,相当狡猾,这就是我说的,新冠病毒是特洛伊木马攻城,载人载物由欧美向中国大陆进击!?1月2日,广东省疾控中心在一名英国输入新冠肺炎确认病例的咽拭子样本中发现了B.1.1.7突变株,与近期英国报道的变异病毒基因序列高度相似。英国变异病毒最大的特点即其S蛋白与人类ACE2受体结合亲和力提高了100倍,传播速度比之前的毒株高达70%,已成为伦敦地区的主要毒株,英国回国的留学生,一定不要只隔离14天就出去逛,还得要居家再多隔离七天,再检查无问题后再玩,切切。保护好自己,也是保护家人,保护所在社区!
香港发现的德尔塔毒株或有全新基因突变,不排除动物传人可能性.
近日测样品,发现Rh内标回收率有突变现象,从100%突变到50%,一会儿又是160%,其他元素也都跟着变,通过内标校正计算后数据倒也可以。进样系统正常,换了雾化器,咨询工程师说是矩管前的银屏蔽圈下面的触点接触不好,换了个新的屏蔽圈,下面的也用砂纸擦了,均无大的改善!求解?大家遇到如此问题吗?
以前做锌分析时终点是突变为亮黄色,最近做样看不到突变,慢慢变亮黄无法判断终点分析方法是这样的,依次加3酸溶样冒白烟,加水至50ML,加氯化铵5g左 右,滴加氨水至铁沉淀完全并过量,加热过滤后调节PH,加抗坏血酸、硫脲、氟化钾,有时也加碘化钾,加二甲酚橙,乙酸乙酸钠缓冲溶液20ML,滴定
我是做水质化验的,现在领导安排要做一份关于水质突变的应急预案,请问各位有没有什么资料可以参考?或者教教我应该怎么写?谢谢
所有的微生物育种工作都离不开菌种筛选。尤其是在诱变育种工作中,筛选是最为艰难的也是最为重要的步骤。经诱变处理后,突变细胞只占存活细胞的百分之几,而能使生产状况提高的细胞又只是突变细胞中的少数。要在大量的细胞中寻找真正需要的细胞,就象是大海捞针,工作量很大。简洁而有效的筛选方法无疑是育种工作成功的关键。为了花费最少的工作量,在最短的时间内取得最大的筛选成效,就要求采用效率较高的科学筛选方案和手段。因为诱变育种中的筛选工作最复杂,所以,本节主要讨论诱变育种的筛选方法,这些方法也为其它育种方法的筛选提供了借鉴。 1. 菌种筛选方案在实际工作中,为了提高筛选效率,往往将筛选工作分为初筛和复筛两步进行。初筛的目的是删去明确不符合要求的大部分菌株,把生产性状类似的菌株尽量保留下来,使优良菌种不致于漏网。因此,初筛工作以量为主,测定的精确性还在其次。初筛的手段应尽可能快速、简单。复筛的目的是确认符合生产要求的菌株,所以,复筛步骤以质为主,应精确测定每个菌株的生产指标。 2. 菌种筛选的手段筛选的手段必需配合不同筛选阶段的要求,对于初筛,要力求快速、简便,对于复筛,应该做到精确,测得的数据要能够反映将来的生产水平。2.1 从菌体形态变异分析有时,有些菌体的形态变异与产量的变异存在着一定的相关性,这就能很容易地将变异菌株筛选出来。尽管相当多的突变菌株并不存在这种相关性,但是在筛选工作中应尽可能捕捉、利用这些直接的形态特征性变化。当然,这种鉴别方法只能用于初筛。有人曾统计过3,484个产维生素B2的阿舒假囊酵母(Eremothecium ashbyii)的变异菌落,发现高产菌株的菌落形态有以下特点:菌落直径呈中等大小(8-10毫米),凡过大或过小者均为低产菌株;色泽深黄色,凡浅黄或白色者皆属低产菌株。又如,在灰黄霉素产生菌荨麻青霉(Penicillium urticae)的育种中,曾发现菌落的棕红色变深者往往产量有所提高,而在赤霉素生产菌藤仓赤霉(Gibberella fujikuroi)中,却发现菌落的紫色加深者产量反而下降。 2.2 平皿快速检测法平皿快速检测法是利用菌体在特定固体培养基平板上的生理生化反应,将肉眼观察不到的产量性状转化成可见的"形态"变化。具体的有纸片培养显色法、变色圈法、透明圈法、生长圈法和抑制圈法等,见图5.6.1。这些方法较粗放,一般只能定性或半定量用,常只用于初筛,但它们可以大大提高筛选的效率。它的缺点是由于培养平皿上种种条件与摇瓶培养,尤其是发酵罐深层液体培养时的条件有很大的差别,有时会造成两者的结果不一致。图 5.6.1 平皿快速检测法示意图平皿快速检测法操作时应将培养的菌体充分分散,形成单菌落,以避免多菌落混杂一起,引起"形态"大小测定的偏差。1) 纸片培养显色法将饱浸含某种指示剂的固体培养基的滤纸片搁于培养皿中,用牛津杯架空,下放小团浸有3%甘油的脱脂棉以保湿,将待筛选的菌悬液稀释后接种到滤纸上,保温培养形成分散的单菌落,菌落周围将会产生对应的颜色变化。从指示剂变色圈与菌落直径之比可以了解菌株的相对产量性状。指示剂可以是酸碱指示剂也可以是能与特定产物反应产生颜色的化合物。2) 变色圈法将指示剂直接掺入固体培养基中,进行待筛选菌悬液的单菌落培养,或喷洒在已培养成分散单菌落的固体培养基表面,在菌落周围形成变色圈。如在含淀粉的平皿中涂布一定浓度的产淀粉酶菌株的菌悬液,使其呈单菌落,然后喷上稀碘液,发生显色反应。变色圈越大,说明菌落产酶的能力越强。而从变色圈的颜色又可粗略判断水解产物的情况。 . S:3) 透明圈法在固体培养基中渗入溶解性差、可被特定菌利用的营养成分,造成浑浊、不透明的培养基背景。将待筛选在菌落周围就会形成透明圈,透明圈的大小反映了菌落利用此物质的能力。在培养基中掺入可溶性淀粉、酪素或CaCO3可以分别用于检测菌株产淀粉酶、产蛋白酶或产酸能力的大小。4) 生长圈法 利用一些有特别营养要求的微生物作为工具菌,若待分离的菌在缺乏上述营养物的条件下,能合成该营养物,或能分泌酶将该营养物的前体转化成营养物,那么,在这些菌的周围就会有工具菌生长,形成环绕菌落生长的生长圈。该法常用来选育氨基酸、核苷酸和维生素的生产菌。工具菌往往都是对应的营养缺陷型菌株。5) 抑制圈法待筛选的菌株能分泌产生某些能抑制工具菌生长的物质,或能分泌某种酶并将无毒的物质水解成对工具菌有毒的物质,从而在该菌落周围形成工具菌不能生长的抑菌圈。例如:将培养后的单菌落连同周围的小块琼脂用穿孔器取出,以避免其它因素干扰,移入无培养基平皿,继续培养4-5天,使抑制物积累,此时的抑制物难以渗透到其它地方,再将其移入涂布有工具菌的平板,每个琼脂块中心间隔距离为2厘米,培养过夜后,即会出现抑菌圈。抑菌圈的大小反映了琼脂块中积累的抑制物的浓度高低。该法常用于抗生素产生菌的筛选,工具菌常是抗生素敏感菌。
对细菌突变,杂交重组、转化、转导、溶源性转换的研究属()范畴研究。 A、形式遗传学 B、分子遗传学 C、生理遗传学 D、生化遗传学

[align=center][b][font=黑体][font=黑体]基于[/font][font=黑体]TILLING技术的基因突变分析[/font][/font][/b][/align][align=center][/align][font=黑体]小麦是我国主要的粮食作物,其栽培面积和总产量仅次于水稻,随着生活水平的日益提高,人们对小麦品质的要求也越来越高,因此,品质改良已成为小麦育种工作的重要目标之一[/font][sup][font='Times New Roman'][[/font][/sup][sup][font=黑体][font=Times New Roman]1[/font][/font][/sup][sup][font='Times New Roman']][/font][/sup][font=黑体]。[/font][font=黑体][font=黑体]小麦是异源[/font][font=Times New Roman]6[/font][font=黑体]倍体植物,基因组庞大,突变机制较为复杂[/font][/font][font=黑体][font=黑体],反向遗传学技术,例如[/font][font=Times New Roman]T-DNA[/font][font=黑体]插入、转座子标签,已经越来越多的应用于研究中。然而,这些方法大多数,应用于基因组较小的模式植物,小麦的多倍性及转化体获得的难度,制约了这些方法的更好使用,而[/font][font=Times New Roman]TILLING[/font][font=黑体]技术不受物种倍性影响,[/font][/font][font=黑体]推动了基因功能研究和品质改良的发展[/font][font=黑体]。[/font][b][font=黑体][font=宋体]1 [/font][font=黑体]材料与方法[/font][/font][/b][font=黑体][font=Times New Roman]1.1 [/font][/font][font=黑体]植物[/font][font=黑体]材料[/font][font=黑体]春小麦([/font][i][font=黑体][font=Times New Roman]Triticum aestivum[/font][/font][/i][font=黑体] [font=Times New Roman]L.[/font][font=黑体])品种龙辐麦[/font][/font][font='Times New Roman']17[font=黑体]经航天诱变[/font][/font][font=黑体]选育而成。[/font][font=黑体][font=黑体]共获得[/font][font=Times New Roman]1122[/font][font=黑体]个[/font][/font][font=黑体][font=Times New Roman]EMS[/font][font=黑体]诱变的龙辐麦[/font][/font][font='Times New Roman']17[/font][font=黑体] [font=Times New Roman]M[/font][/font][sub][font=黑体][font=Times New Roman]2[/font][/font][/sub][font=黑体]代单粒传[/font][font=黑体]植株,苗期单株编号,采集[/font][font=黑体][font=黑体]叶片,迅速冷冻干燥,用于[/font][font=Times New Roman]DNA[/font][font=黑体]提取。[/font][/font][font=黑体][font=Times New Roman]1.2 [/font][font=黑体]基因组[/font][font=Times New Roman]DNA[/font][font=黑体]提取及[/font][font=Times New Roman]DNA[/font][font=黑体]池构建[/font][/font][font=黑体][font=黑体]将干燥后的叶片按编号置于装有珠子的[/font][font=Times New Roman]1.5ml[/font][font=黑体]离心管中,利用组织研磨器[/font][font=Times New Roman]Vibration Mill Type MM301[/font][font=黑体]([/font][font=Times New Roman]Retsch GmbH Co, Germany[/font][font=黑体])将叶片磨成粉末状;加[/font][font=Times New Roman]600[/font][/font][font='Times New Roman']μl[/font][font=黑体] [font=Times New Roman]DNA[/font][/font][font='Times New Roman'] Extraction buffer[/font][font='Times New Roman'] [/font][font='Times New Roman'][[/font][font=黑体][font=Times New Roman]0.1[/font][/font][font='Times New Roman'] mol/ L[/font][font=黑体] [/font][font=黑体][font=Times New Roman]Tris[/font][font=黑体];[/font][font=Times New Roman]0.[/font][/font][font=黑体][font=Times New Roman]1[/font][/font][font='Times New Roman'] mol/ L[/font][font=黑体] [font=Times New Roman]KCl[/font][font=黑体];[/font][font=Times New Roman]0.5M EDTA pH 8.0[/font][font=黑体];[/font][font=Times New Roman]1[/font][/font][font='Times New Roman'] mol/ L[/font][font=黑体] [font=Times New Roman]PVP 40[/font][font=黑体];[/font][font=Times New Roman]1.5[/font][/font][font='Times New Roman'] mol/ L[/font][font='Times New Roman'] Na[/font][sub][font='Times New Roman']2[/font][/sub][font='Times New Roman']S[/font][sub][font='Times New Roman']2[/font][/sub][font='Times New Roman']O[/font][sub][font='Times New Roman']6[/font][/sub][font='Times New Roman']][/font][font=黑体][font=黑体],充分混匀;保温振动[/font][font=Times New Roman]1 h[/font][font=黑体];加[/font][font=Times New Roman]200μl 5 mol/[/font][/font][font='Times New Roman']L KAc[/font][font=黑体][font=黑体],混匀并离心;取约[/font][font=Times New Roman]300 μl[/font][font=黑体]上清液加至[/font][font=Times New Roman]165 μl[/font][font=黑体]预冷的异丙醇中,沉淀[/font][font=Times New Roman]DNA[/font][font=黑体];用[/font][font=Times New Roman]70 %[/font][font=黑体]乙醇洗[/font][font=Times New Roman]DNA[/font][font=黑体],干燥后,溶于[/font][font=Times New Roman]TER[/font][/font][font='Times New Roman'][[/font][font=黑体][font=Times New Roman]Tris 10mmol/L[/font][font=黑体],[/font][font=Times New Roman]EDTA 1 mmol/L[/font][font=黑体],[/font][font=Times New Roman]RNA[/font][font=黑体]酶[/font][font=Times New Roman]0.05mg/ml[/font][/font][font='Times New Roman']][/font][font=黑体][font=黑体],[/font][font=Times New Roman]-20 [/font][font=黑体]℃保存备用。[/font][/font][font=黑体][font=黑体]利用[/font][font=Times New Roman]1.0%[/font][font=黑体]的琼脂糖凝胶电泳检测[/font][font=Times New Roman]DNA[/font][font=黑体]样品,并将其稀释至[/font][font=Times New Roman]40 ng/[/font][/font][font='Times New Roman']μ[/font][font=黑体][font=Times New Roman]l[/font][font=黑体],随机两两等量混匀,构建两倍的[/font][font=Times New Roman]DNA[/font][font=黑体]池,用于后续[/font][font=Times New Roman]TILLING[/font][font=黑体]筛选。[/font][/font][font=黑体][font=Times New Roman]1.3 [/font][font=黑体]特异性引物设计及筛选[/font][/font][font=黑体][font=黑体]根据[/font][font=Times New Roman]NCBI[/font][font=黑体]提供的目标基因序列信息,在线设计引物,并用[/font][font=Times New Roman]1%[/font][font=黑体]琼脂糖凝胶电泳检测[/font][font=Times New Roman][url=https://insevent.instrument.com.cn/t/jp][color=#3333ff]PCR[/color][/url][/font][font=黑体]产物,选择[/font][font=Times New Roman][url=https://insevent.instrument.com.cn/t/jp][color=#3333ff]PCR[/color][/url][/font][font=黑体]产物条带单一,大小在[/font][font=Times New Roman]700-1500[/font][font=黑体]左右的[/font][font=Times New Roman]4[/font][font=黑体]对引物(表[/font][font=Times New Roman]1[/font][font=黑体]),其中[/font][/font][font=黑体]除[/font][i][font=黑体][font=Times New Roman]Pinb[/font][/font][/i][font=黑体][font=黑体]引自[/font][font=Times New Roman]Wang[/font][font=黑体]等[/font][/font][sup][font='Times New Roman'][[/font][/sup][sup][font=黑体][font=Times New Roman]2[/font][/font][/sup][sup][font='Times New Roman']][/font][/sup][font=黑体][font=Times New Roman](2008)[/font][font=黑体]。[/font][/font][align=center][font=黑体][font=黑体]表[/font][font=Times New Roman]1 [/font][font=黑体]用于[/font][font=Times New Roman]TILLING[/font][font=黑体]检测的基因及其荧光标记引物序列[/font][/font][/align][align=center][font='Times New Roman']Table1 [/font][font=黑体] [font=Times New Roman]Genes and their [/font][/font][font='Times New Roman']IRDye labled primer sequence[/font][font=黑体][font=Times New Roman]s [/font][/font][font='Times New Roman']used[/font][font=黑体] [font=Times New Roman]in TILLING[/font][/font][font='Times New Roman'] [/font][/align][table][tr][td=1,2][align=center][b][font=黑体]基因[/font][/b][/align][align=center][b][font=黑体][font=Times New Roman]Gene[/font][/font][/b][/align][/td][td=2,1][align=center][b][font=黑体][font=黑体]引物序列[/font] [font=Times New Roman]Primer sequence (5[/font][font=黑体]′–[/font][font=Times New Roman]3[/font][font=黑体]′[/font][font=Times New Roman])[/font][/font][/b][/align][/td][td=1,2][align=center][b][font=黑体]长度[/font][/b][/align][align=center][b][font='Times New Roman']Length[/font][font=黑体][font='Times New Roman'] [/font][font=Times New Roman](bp)[/font][/font][/b][/align][/td][/tr][tr][td][align=center][b][font=黑体][font=黑体]正向[/font] [font=Times New Roman]Forward[/font][/font][/b][/align][/td][td][align=center][b][font=黑体][font=黑体]反向[/font] [font=Times New Roman]Reverse [/font][/font][/b][/align][/td][/tr][tr][td][align=center][i][font='Times New Roman']P[/font][font=黑体][font=Times New Roman]in[/font][/font][font='Times New Roman']b[/font][/i][/align][/td][td][align=center][font='Times New Roman']CCAACGAAACTAATGAGAAATAAAAAGGTG[/font][/align][/td][td][align=center][font='Times New Roman']AAGTTGTTGGATGGACGAATAAGGTT[/font][/align][/td][td][align=center][font='Times New Roman']1334[/font][font=黑体][font='Times New Roman'] [/font][/font][/align][/td][/tr][tr][td][align=center][i][font='Times New Roman']W[/font][font=黑体][font=Times New Roman]a[/font][/font][font='Times New Roman']x[/font][font=黑体][font=Times New Roman]y[/font][/font][/i][/align][/td][td][align=center][font=黑体][font=Times New Roman]ACCCGCATGGTGTTTGATAATTTCAGTG[/font][/font][/align][/td][td][align=center][font=黑体][font=Times New Roman]AGAATGCCACCTAGCCATGAAATGAGT[/font][/font][/align][/td][td][align=center][font='Times New Roman']790[/font][font=黑体][font='Times New Roman'] [/font][/font][/align][/td][/tr][/table][font=黑体][font=Times New Roman]1.4 TILLING[/font][font=黑体]检测[/font][/font][font=黑体]参照潘娜[/font][sup][font='Times New Roman'][[/font][/sup][sup][font=黑体][font=Times New Roman]3[/font][/font][/sup][sup][font='Times New Roman']][/font][/sup][font=黑体][font=黑体]([/font][font=Times New Roman]2011[/font][font=黑体])建立的小麦[/font][font=Times New Roman]TILLING[/font][font=黑体]技术检测平台,对目的基因进行检测。采用[/font][/font][font=黑体][font=Times New Roman]Touchdown [/font][/font][font=黑体][font=Times New Roman][url=https://insevent.instrument.com.cn/t/jp][color=#3333ff]PCR[/color][/url][/font][font=黑体]程序,[/font][/font][font=黑体][font=Times New Roman]10μl [/font][/font][font=黑体][font=Times New Roman][url=https://insevent.instrument.com.cn/t/jp][color=#3333ff]PCR[/color][/url][/font][/font][font=黑体]体系含[/font][font='Times New Roman']10 ×Buffer[/font][font=黑体] [font=Times New Roman]0.5μl[/font][font=黑体];[/font][/font][font='Times New Roman']Mg[/font][sup][font='Times New Roman']2+[/font][/sup][sup][font=黑体] [/font][/sup][font=黑体][font=Times New Roman]0.6[/font][/font][font=黑体][font=Times New Roman]μl[/font][/font][font=黑体];[/font][font=黑体][font=Times New Roman]dNTP [/font][/font][font=黑体][font=Times New Roman]0.8[/font][/font][font=黑体][font=Times New Roman]μl[/font][/font][font=黑体];[/font][font=黑体]引物各[/font][font=黑体][font=Times New Roman]0.4[/font][/font][font=黑体][font=Times New Roman]μl[/font][font=黑体];[/font][/font][font=黑体][font=Times New Roman]Ex Taq HS DNA[/font][font=黑体]聚合酶[/font][font=Times New Roman]0.05[/font][/font][font=黑体][font=Times New Roman]μl[/font][font=黑体],[/font][/font][font=黑体][font=黑体]在[/font][font=Times New Roman]Bio-Rad c1000[/font][font=黑体]仪上扩增。扩增后加[/font][font=Times New Roman]20[/font][/font][font=黑体][font=Times New Roman]μl[/font][/font][font=黑体] [font=Times New Roman]0.1M CEL[/font][font=黑体]Ⅰ酶,[/font][font=Times New Roman]45[/font][font=黑体]℃酶切[/font][font=Times New Roman]15 min[/font][font=黑体],利用[/font][font=Times New Roman]Sephadex G50[/font][font=黑体]([/font][font=Times New Roman]GE Healthcare [/font][font=黑体]公司)纯化板纯化酶切产物,并[/font][font=Times New Roman]90[/font][font=黑体]℃浓缩[/font][font=Times New Roman]35-45 min[/font][font=黑体]。利用[/font][font=Times New Roman]6[/font][font=黑体]%变性聚丙烯酰胺凝胶,在[/font][font=Times New Roman]LI-COR 4300[/font][font=黑体]仪器中电泳检测酶切产物[/font][/font][font=黑体]并[/font][font=黑体][font=黑体]采用[/font][font=Times New Roman]Gelbuddy[/font][font=黑体]软件对电泳图像分析处理,标记突变位点。[/font][/font][font=黑体][font=Times New Roman]1.5 [/font][font=黑体]基因点突变分析[/font][/font][font=黑体][font=黑体]检测到突变后对[/font][font=Times New Roman][url=https://insevent.instrument.com.cn/t/jp][color=#3333ff]PCR[/color][/url][/font][font=黑体]产物进行测序验证,利用[/font][font=Times New Roman]Invitrogen[/font][font=黑体]软件、[/font][font=Times New Roman]NCBI[/font][font=黑体]等对序列进行比对、翻译等分析,确定突变位置与类型。[/font][/font][font=黑体][font=黑体]点突变密度[/font][font=Times New Roman](%)=[/font][font=黑体]突变碱基数[/font][font=Times New Roman]/[/font][font=黑体]检测总碱基数×[/font][font=Times New Roman]100%[/font][font=黑体]。[/font][/font][b][font=黑体][font=宋体]2 [/font][font=黑体]结果与分析[/font][/font][/b][font=黑体]本实验对[/font][font=黑体]小麦籽粒硬度基因[/font][i][font=黑体][font=Times New Roman]pinb[/font][/font][/i][font=黑体][font=黑体]进行了[/font][font=Times New Roman]1122[/font][font=黑体]个单株的[/font][font=Times New Roman]TILLING[/font][font=黑体]检测。[/font][/font][font=黑体]基因的突变密度分别为[/font][font=黑体][font=Times New Roman]1/374.18 kb[/font][/font][font=黑体]。[/font][i][font=黑体][font=Times New Roman]Pinb[/font][/font][/i][font=黑体][font=黑体]基因共获得[/font][font=Times New Roman]7[/font][font=黑体]个突变株系的[/font][font=Times New Roman]4[/font][font=黑体]个不同的突变位点[/font][font=Times New Roman]([/font][font=黑体]图[/font][font=Times New Roman]1)[/font][font=黑体],有[/font][font=Times New Roman]6[/font][font=黑体]个突变单株的碱基突变位点均位于外显子区域,突变株编号及突变位点分别是[/font][font=Times New Roman]LF996[/font][font=黑体]第[/font][/font][font='Times New Roman']51[/font][font=黑体][font=Times New Roman]2bp[/font][font=黑体]位的[/font][font=Times New Roman]C-T[/font][font=黑体]碱基转换,[/font][font=Times New Roman]LF[/font][/font][font=黑体][font=Times New Roman]892[/font][font=黑体]、[/font][font=Times New Roman]LF997[/font][font=黑体]、[/font][font=Times New Roman]LF919[/font][font=黑体]第[/font][/font][font='Times New Roman']7[/font][font=黑体][font=Times New Roman]26bp[/font][font=黑体]位的[/font][/font][font='Times New Roman']G[/font][font=黑体][font=Times New Roman]-[/font][/font][font='Times New Roman']A[/font][font=黑体][font=黑体]碱基转换,及[/font][font=Times New Roman]LF791[/font][font=黑体]、[/font][font=Times New Roman]LF1114[/font][font=黑体]第[/font][/font][font='Times New Roman']750[/font][font=黑体][font=Times New Roman]bp[/font][font=黑体]位的[/font][/font][font='Times New Roman']G[/font][font=黑体][font=Times New Roman]-[/font][/font][font='Times New Roman']A[/font][font=黑体][font=黑体]碱基转换,[/font][font=Times New Roman]LF996 [/font][/font][i][font=黑体][font=Times New Roman]pinb[/font][/font][/i][font=黑体][font=黑体]基因第[/font][font=Times New Roman]37[/font][font=黑体]为氨基酸[/font][font=Times New Roman]S[/font][font=黑体]变为氨基酸[/font][font=Times New Roman]F[/font][font=黑体],[/font][font=Times New Roman]LF[/font][/font][font=黑体][font=Times New Roman]892[/font][font=黑体]、[/font][font=Times New Roman]LF997[/font][font=黑体]、[/font][font=Times New Roman]LF919[/font][/font][i][font=黑体] [font=Times New Roman]pinb[/font][/font][/i][font=黑体][font=黑体]基因第[/font][font=Times New Roman]110[/font][font=黑体]为氨基酸[/font][font=Times New Roman]G[/font][font=黑体]变为氨基酸[/font][font=Times New Roman]S[/font][font=黑体],[/font][font=Times New Roman]LF791[/font][font=黑体]、[/font][font=Times New Roman]LF1114[/font][font=黑体]第[/font][font=Times New Roman]116[/font][font=黑体]位氨基酸[/font][font=Times New Roman]W[/font][font=黑体]被终止,内含子区域突变是[/font][font=Times New Roman]LF890[/font][font=黑体]突变株第[/font][font=Times New Roman]350bp[/font][font=黑体]碱基[/font][font=Times New Roman]G[/font][font=黑体]转换为碱基[/font][font=Times New Roman]A[/font][font=黑体]。见表[/font][font=Times New Roman]1[/font][/font][align=center][img=,442,414]https://ng1.17img.cn/bbsfiles/images/2022/12/202212160839201707_5062_3237657_3.png!w442x414.jpg[/img][/align][align=center][font=黑体] [/font][/align][align=center][font=黑体][font=黑体]图[/font][font=Times New Roman]1 [/font][/font][i][font='Times New Roman']W[/font][font=黑体][font=Times New Roman]a[/font][/font][font='Times New Roman']x[/font][font=黑体][font=Times New Roman]y[/font][/font][/i][font=黑体]基因电泳图[/font][/align][align=center][font='Times New Roman']Fig[/font][font=黑体][font=Times New Roman]1 [/font][/font][font='Times New Roman']E[/font][font=黑体][font=Times New Roman]lectrophorogram of [/font][/font][i][font='Times New Roman']W[/font][font=黑体][font=Times New Roman]a[/font][/font][font='Times New Roman']x[/font][font=黑体][font=Times New Roman]y gene[/font][/font][/i][/align][align=center][i][font=黑体] [/font][/i][/align][font='Times New Roman'][/font][align=center][font=黑体][font=黑体]表[/font][font=Times New Roman]1[/font][/font][font=黑体] [font=黑体]基因点突变信息[/font][/font][/align][align=center][font=黑体][font=Times New Roman]Table1 Point[/font][/font][font='Times New Roman'] mutations[/font][font=黑体] [font=Times New Roman]of[/font][/font][font='Times New Roman'] quality[/font][i][font=黑体] [/font][/i][font=黑体][font=Times New Roman]gene[/font][/font][/align][table][tr][td][align=center][b][font='Times New Roman'][font=黑体]基因名称[/font][/font][/b][/align][align=center][b][font='Times New Roman']Gene[/font][font=黑体][font='Times New Roman'] [/font][/font][font='Times New Roman']name[/font][/b][/align][/td][td][align=center][b][font='Times New Roman'][font=黑体]登录号[/font][/font][/b][/align][align=center][b][font='Times New Roman']Accession[/font][font=黑体][font='Times New Roman'] [/font][/font][font='Times New Roman']No.[/font][/b][/align][/td][td][align=center][b][font='Times New Roman'][font=黑体]基因大小[/font][/font][/b][/align][align=center][b][font='Times New Roman']Gene size[/font][font=黑体][font='Times New Roman'] [/font][/font][font='Times New Roman'](bp)[/font][/b][/align][/td][td][align=center][b][font='Times New Roman'][url=https://insevent.instrument.com.cn/t/jp][color=#3333ff]PCR[/color][/url][font=黑体]扩增长度[/font][/font][/b][/align][align=center][b][font='Times New Roman'][url=https://insevent.instrument.com.cn/t/jp][color=#3333ff]PCR[/color][/url] size (bp)[/font][/b][/align][/td][td][align=center][b][font='Times New Roman'][font=黑体]等位变异[/font][/font][/b][/align][align=center][b][font='Times New Roman']Mutant[/font][font=黑体][font='Times New Roman'] [/font][/font][font='Times New Roman']allele[/font][/b][/align][/td][td][align=center][b][font='Times New Roman'][font=黑体]氨基酸突变[/font][/font][/b][/align][align=center][b][font='Times New Roman']amino acid[/font][font=黑体][font='Times New Roman'] [/font][/font][font='Times New Roman']mutation[/font][/b][/align][/td][td][align=center][b][font='Times New Roman'][font=黑体]突变[/font][/font][font=黑体]株编号[/font][/b][/align][align=center][b][font=Calibri]Number[/font][font=黑体][font='Times New Roman'] [/font][/font][font='Times New Roman']of mutation[/font][/b][/align][/td][td][align=center][b][font='Times New Roman'][font=黑体]总突变频率[/font][/font][/b][/align][align=center][b][font='Times New Roman']Total[/font][/b][font=Calibri][color=#434343] [/color][/font][b][font='Times New Roman']mutation density[/font][font=黑体][font='Times New Roman'] [/font][/font][font='Times New Roman'](kb)[/font][/b][/align][/td][/tr][tr][td][align=center][i][font='Times New Roman']pinb[/font][/i][/align][/td][td][align=center][font='Times New Roman']AJ302100.1[/font][/align][/td][td][align=center][font='Times New Roman']870[/font][/align][/td][td][align=center][font='Times New Roman']1334[/font][/align][/td][td][align=center][font=黑体][font=Times New Roman]C[/font][/font][font='Times New Roman']51[/font][font=黑体][font=Times New Roman]2T[/font][/font][/align][/td][td][align=center][font=黑体][font=Times New Roman]S37F[/font][/font][/align][/td][td][align=center][font=黑体][font=Times New Roman]996[/font][/font][/align][/td][td=1,4][align=center][font=黑体][font=Times New Roman]1/374.18 [/font][/font][/align][/td][/tr][tr][td][align=center][font='Times New Roman'] [/font][/align][/td][td][align=center][font='Times New Roman'] [/font][/align][/td][td][align=center][font='Times New Roman'] [/font][/align][/td][td][align=center][font='Times New Roman'] [/font][/align][/td][td][align=center][font='Times New Roman']G7[/font][font=黑体][font=Times New Roman]26[/font][/font][font='Times New Roman']A [/font][/align][/td][td][align=center][font=黑体][font=Times New Roman]G110S [/font][/font][/align][/td][td][align=center][font=黑体][font=Times New Roman]892[/font][font=黑体]、[/font][font=Times New Roman]919[/font][font=黑体]、[/font][font=Times New Roman]997[/font][/font][/align][/td][/tr][tr][td][align=center][font='Times New Roman'] [/font][/align][/td][td][align=center][font='Times New Roman'] [/font][/align][/td][td][align=center][font='Times New Roman'] [/font][/align][/td][td][align=center][font='Times New Roman'] [/font][/align][/td][td][align=center][font='Times New Roman']G750A[/font][/align][/td][td][align=center][font=黑体][font=Times New Roman]W116-[/font][/font][/align][/td][td][align=center][font=黑体][font=Times New Roman]791[/font][font=黑体]、[/font][font=Times New Roman]1114[/font][/font][/align][/td][/tr][tr][td][align=center][font='Times New Roman'] [/font][/align][/td][td][align=center][font='Times New Roman'] [/font][/align][/td][td][align=center][font='Times New Roman'] [/font][/align][/td][td][align=center][font='Times New Roman'] [/font][/align][/td][td][align=center][font='Times New Roman']G350A[/font][/align][/td][td][align=center][font='Times New Roman'][font=黑体]未改变[/font][/font][/align][/td][td][align=center][font=黑体][font=Times New Roman]890[/font][/font][/align][/td][/tr][/table][font='Times New Roman'][/font][b][b][font=黑体][font=Times New Roman]3[/font][/font][font=黑体] [font=黑体]讨论[/font][/font][/b][/b][font=黑体]籽粒硬度是小麦重要的品质性状,对食品加工及磨粉质量都有重要影响。[/font][i][font='Times New Roman']P[/font][font=黑体][font=Times New Roman]inb[/font][/font][/i][font=黑体]是调控籽粒硬度的主效基因之一,与[/font][i][font=黑体][font=Times New Roman]pina[/font][/font][/i][font=黑体]基因共同作用决定小麦胚乳质地[/font][sup][font='Times New Roman'][[/font][/sup][sup][font=黑体][font=Times New Roman]4-5[/font][/font][/sup][sup][font='Times New Roman']][/font][/sup][font=黑体]。[/font][font=黑体]本实验[/font][i][font=黑体][font=Times New Roman]Pinb[/font][/font][/i][font=黑体]基因的突变密度是[/font][font=黑体][font=Times New Roman]1/374.18 [/font][/font][font=黑体][font=Times New Roman]kb[/font][font=黑体],低于[/font][/font][font=黑体][font=Times New Roman]Feiz[/font][font=黑体]等[/font][/font][sup][font='Times New Roman'][[/font][/sup][sup][font=黑体][font=Times New Roman]6[/font][/font][/sup][sup][font='Times New Roman']][/font][/sup][font=黑体]在普通软质小麦突变体库得到的[/font][i][font=黑体][font=Times New Roman]pinb[/font][/font][/i][font=黑体]基因[/font][font=黑体][font=Times New Roman]1/12 kb[/font][font=黑体]的[/font][/font][font=黑体]突变密度,但高于[/font][font=黑体]潘娜[/font][sup][font='Times New Roman'][[/font][/sup][sup][font=黑体][font=Times New Roman]3[/font][/font][/sup][sup][font='Times New Roman']][/font][/sup][font=黑体][font=黑体]利用空间诱变新麦[/font][font=Times New Roman]18[/font][font=黑体]突变体库群体得到的该基因[/font][/font][font=黑体][font=Times New Roman]1/3073 kb[/font][font=黑体]的[/font][/font][font=黑体]突变密度。[/font][font=黑体][font=Times New Roman]Morris[/font][font=黑体]等[/font][/font][sup][font='Times New Roman'][[/font][/sup][sup][font=黑体][font=Times New Roman]5, 7[/font][/font][/sup][sup][font='Times New Roman']][/font][/sup][font=黑体]获得的硬红春小麦[/font][i][font=黑体][font=Times New Roman]pinb[/font][/font][/i][font=黑体]基因突变体[/font][font=黑体],分别发生在[/font][i][font=黑体][font=Times New Roman]pinb[/font][/font][/i][font=黑体][font=黑体]基因第[/font][font=Times New Roman]39[/font][font=黑体]位和第[/font][font=Times New Roman]44[/font][font=黑体]位的色氨基酸[/font][font=Times New Roman]T[/font][font=黑体]及第[/font][font=Times New Roman]56[/font][font=黑体]位半胱氨酸[/font][font=Times New Roman]C[/font][font=黑体],均突变为终止密码子。[/font][/font][font=黑体][font=Times New Roman]Wang[/font][font=黑体]等[/font][/font][sup][font='Times New Roman'][[/font][/sup][sup][font=黑体][font=Times New Roman]13[/font][/font][/sup][sup][font='Times New Roman']][/font][/sup][font=黑体]获得[/font][i][font=黑体][font=Times New Roman]Pinb[/font][/font][/i][font=黑体][font=黑体]基因,[/font][font=Times New Roman]382bp[/font][font=黑体]处[/font][font=Times New Roman]C [/font][/font][font='Times New Roman']–[/font][font=黑体] [font=Times New Roman]T[/font][font=黑体]碱基转换和[/font][font=Times New Roman]257bp[/font][font=黑体]处[/font][font=Times New Roman]G [/font][/font][font='Times New Roman']–[/font][font=黑体] [font=Times New Roman]A[/font][font=黑体]碱基转换。[/font][/font][font=黑体]在潘娜[/font][sup][font='Times New Roman'][[/font][/sup][sup][font=黑体][font=Times New Roman]3[/font][/font][/sup][sup][font='Times New Roman']][/font][/sup][font=黑体]创制[/font][font=黑体][font=黑体]的[/font][font=Times New Roman]TILLING[/font][font=黑体]群体中,[/font][/font][i][font=黑体][font=Times New Roman]pinb[/font][/font][/i][font=黑体][font=黑体]基因组[/font][font=Times New Roman]736 bp[/font][font=黑体]的[/font][font=Times New Roman]A[/font][font=黑体]碱基缺失,引起移码突变,而本实验突变株[/font][font=Times New Roman]LF[/font][/font][font=黑体][font=Times New Roman]791[/font][font=黑体]、[/font][font=Times New Roman]LF1114[/font][font=黑体],[/font][font=Times New Roman]LF996[/font][font=黑体],[/font][font=Times New Roman]LF[/font][/font][font=黑体][font=Times New Roman]892[/font][font=黑体]、[/font][font=Times New Roman]LF997[/font][font=黑体]、[/font][font=Times New Roman]LF919[/font][font=黑体]的基因突变依次为[/font][/font][font='Times New Roman']750[/font][font=黑体][font=Times New Roman]bp[/font][font=黑体]位的[/font][/font][font='Times New Roman']G[/font][font=黑体][font=Times New Roman]-[/font][/font][font='Times New Roman']A[/font][font=黑体]碱基转换,[/font][font='Times New Roman']51[/font][font=黑体][font=Times New Roman]2bp[/font][font=黑体]位的[/font][font=Times New Roman]C-T[/font][font=黑体]碱基转换,[/font][/font][font='Times New Roman']7[/font][font=黑体][font=Times New Roman]26bp[/font][font=黑体]位的[/font][/font][font='Times New Roman']G[/font][font=黑体][font=Times New Roman]-[/font][/font][font='Times New Roman']A[/font][font=黑体]碱基转换,[/font][font=黑体]均导致错义突变。[/font][font=黑体]王亮等[/font][sup][font='Times New Roman'][[/font][/sup][sup][font=黑体][font=Times New Roman]8[/font][/font][/sup][sup][font='Times New Roman']][/font][/sup][font=黑体][font=黑体]和[/font][font=Times New Roman]Chen[/font][font=黑体]等[/font][/font][sup][font='Times New Roman'][[/font][/sup][sup][font=黑体][font=Times New Roman]9[/font][/font][/sup][sup][font='Times New Roman']][/font][/sup][font=黑体][font=黑体]的研究中几乎所有[/font][font=Times New Roman]Puroindoline[/font][font=黑体]变异类型的籽粒硬度值都显著高于野生型,而[/font][/font][font=黑体][font=黑体]本实验,对突变株进行表型分析,籽粒硬度测定显示[/font][font=Times New Roman]LF996[/font][font=黑体]、 [/font][font=Times New Roman]LF[/font][/font][font=黑体][font=Times New Roman]892[/font][font=黑体]、[/font][/font][font=黑体][font=Times New Roman]LF1114[/font][font=黑体]突变株的[/font][/font][font=黑体]籽粒硬度均[/font][font=黑体]极显著低于野生型。此外,[/font][font=黑体]突变体自交系研究发现[/font][font=黑体],突变[/font][font=黑体]对小麦出粉率、面包体积、面粉灰分等品质性状均有影响,[/font][font=黑体][font=黑体]不同[/font][font=Times New Roman]Puroindoline[/font][font=黑体]变异类型之间在磨粉、面包等加工品质上也存在较差别[/font][/font][sup][font='Times New Roman'][[/font][/sup][sup][font=黑体][font=Times New Roman]9-10[/font][/font][/sup][sup][font='Times New Roman']][/font][/sup][font=黑体]。基于以上的研究发现,可利用本实验获得的突变株,进行后代品质研究,为品质改良奠定基础。[/font][b][b][font=黑体][font=Times New Roman]4 [/font][/font][font=黑体][font=黑体]结[/font] [font=黑体]论[/font][/font][/b][/b][font=黑体]本研究所获得的基因点突变及表型突变株为植物基因功能研究及小麦品质改良提供了新材料。[/font][font=黑体] [/font][font=黑体] [/font][font=黑体][font=Times New Roman]References[/font][/font][font='Times New Roman'][[/font][font=黑体][font=Times New Roman]1[/font][/font][font='Times New Roman']][/font][font=黑体] [/font][font=黑体][font=Times New Roman]Liu L([/font][font=黑体]刘丽[/font][font=Times New Roman]), Yang J-H([/font][font=黑体]杨金华[/font][font=Times New Roman]), Hu Y-X([/font][font=黑体]胡银星[/font][font=Times New Roman]), Cheng G([/font][font=黑体]程耿[/font][font=Times New Roman]).[/font][/font][font='Times New Roman'] Research Progress in Effects of Glutenin Subunits on[/font][font=黑体] [/font][font='Times New Roman']Wheat Processing Quality[/font][font=黑体][font=Times New Roman].[/font][/font][font='Times New Roman'] [/font][font=黑体] [/font][i][font='Times New Roman']Journal of Agricultural Science and Technology[/font][/i][font=黑体] [font=Times New Roman]([/font][font=黑体]中国农业科技导报[/font][font=Times New Roman]), 2012, 14(1): 33-42 [/font][/font][font='Times New Roman'](in Chinese with English abstract)[/font][font='Times New Roman'][[/font][font=黑体][font=Times New Roman]2[/font][/font][font='Times New Roman']][/font][font=黑体] [/font][font='Times New Roman']Wang J, Sun J Z, Liu D C, Yang W L, Wang D W, Tong Y P, Zhang A M. Analysis of [/font][i][font='Times New Roman']Pina[/font][/i][font='Times New Roman'] and [/font][i][font='Times New Roman']Pinb[/font][/i][font=黑体] [/font][font='Times New Roman']alleles in the microcore collections of chinese wheat germplasm by Ecotilling and identification of[/font][font=黑体] [/font][font='Times New Roman']a novel [/font][i][font='Times New Roman']Pinb[/font][/i][font='Times New Roman'] allele[/font][font=黑体][font=Times New Roman]. [/font][/font][font='Times New Roman'] [/font][i][font='Times New Roman']Journal of Cereal Science[/font][/i][font=黑体] [/font][font='Times New Roman']2008, 48(3): 836-[/font][font=黑体][font=Times New Roman]842[/font][/font][font='Times New Roman'][[/font][font=黑体][font=Times New Roman]3[/font][/font][font='Times New Roman']][/font][font=黑体][font=黑体]潘娜[/font][font=Times New Roman]. [/font][font=黑体]空间环境诱发小麦突变体的[/font][font=Times New Roman]TILLING[/font][font=黑体]分析[/font][font=Times New Roman], [/font][font=黑体]中国农业科学院[/font][font=Times New Roman], 2011.[/font][/font][font='Times New Roman'][[/font][font=黑体][font=Times New Roman]4[/font][/font][font='Times New Roman']][/font][font=黑体][font=Times New Roman]CaPPaerlliR[/font][font=黑体],[/font][font=Times New Roman]Bo[/font][font=黑体]币[/font][font=Times New Roman]elloqGirouxMJ[/font][font=黑体],[/font][font=Times New Roman]AmoorsoMGPuorindolineA[/font][font=黑体]一[/font][font=Times New Roman]geneexPerssion15involved[/font][/font][font=黑体][font=Times New Roman]inassociationofPuroindolinetostacrh.TheoerctialandAPPliedGenetics[/font][font=黑体],[/font][font=Times New Roman]2003[/font][font=黑体],[/font][font=Times New Roman]107:1463[/font][font=黑体]一[/font][font=Times New Roman]1468[/font][/font][font='Times New Roman'][[/font][font=黑体][font=Times New Roman]5[/font][/font][font='Times New Roman']][/font][font=黑体][font=Times New Roman]Chen F. Molecular Characterization of Puroindoline Alleles in Chinese and CIMMYT Common Wheats and Their [/font][/font][font='Times New Roman']Eeffct[/font][font=黑体] [/font][font='Times New Roman']on[/font][font=黑体] [/font][font='Times New Roman']Porcessing[/font][font=黑体] [/font][font='Times New Roman']Quaiity[/font][font='Times New Roman'][[/font][font=黑体][font=Times New Roman]6[/font][/font][font='Times New Roman']]Feiz L, Martin J M, Giroux M J. Creation and functional analysis of new Puroindoline alleles[/font][font=黑体] [/font][font='Times New Roman']in Triticum aestivum.[/font][i][font='Times New Roman']Theoretical and Applied Genetics[/font][/i][font='Times New Roman'],2009, 118:247-257[/font][font='Times New Roman'][[/font][font=黑体][font=Times New Roman]7[/font][/font][font='Times New Roman']][/font][font=黑体] [font=Times New Roman]Morris C F[/font][font=黑体],[/font][font=Times New Roman]Lillemo M[/font][font=黑体],[/font][font=Times New Roman]Simeone M C[/font][font=黑体],[/font][font=Times New Roman]Giorux M J[/font][font=黑体],[/font][font=Times New Roman]Bbab S L[/font][font=黑体],[/font][font=Times New Roman]Kimberiee K K.Pervalence of [/font][/font][font='Times New Roman']Puorindoline[/font][font=黑体] [/font][font='Times New Roman']garin[/font][font=黑体] [/font][font='Times New Roman']hdarness[/font][font=黑体] [/font][font='Times New Roman']genotypes[/font][font=黑体] [/font][font='Times New Roman']among[/font][font=黑体] [/font][font='Times New Roman']historieally[/font][font=黑体] [/font][font='Times New Roman']significant[/font][font=黑体] [/font][font='Times New Roman']North[/font][font=黑体] [/font][font='Times New Roman']American[/font][font=黑体] [/font][font='Times New Roman']s[/font][font=黑体][font=Times New Roman]p[/font][/font][font='Times New Roman']ring[/font][font=黑体] [/font][font='Times New Roman']and[/font][font=黑体] [/font][font='Times New Roman']winter[/font][font=黑体] [font=Times New Roman]wheats. [/font][/font][i][font=黑体][font=Times New Roman]Crop Scienee[/font][/font][/i][font=黑体][font=黑体],[/font][font=Times New Roman]2001,41:218-228[/font][/font][font='Times New Roman'][[/font][font=黑体][font=Times New Roman]8[/font][/font][font='Times New Roman']][/font][font=黑体] [font=Times New Roman]C[/font][/font][font=黑体][font=Times New Roman]hen F[/font][font=黑体],[/font][font=Times New Roman]He z H[/font][font=黑体],[/font][font=Times New Roman]Xia x C[/font][font=黑体],[/font][font=Times New Roman]el a1[/font][font=黑体].[/font][font=Times New Roman]Molecular and biochemical charac[/font][font=黑体]—[/font][/font][font='Times New Roman']terization of P“roindoline a and b alleles in Chinese landraces and[/font][font=黑体][font=Times New Roman]historical eultivars[J][/font][font=黑体].[/font][font=Times New Roman]Theoretical and Applied Genetics. 2006[/font][font=黑体],[/font][font=Times New Roman]112[/font][font=黑体]:[/font][font=Times New Roman]400[/font][font=黑体]—[/font][font=Times New Roman]409[/font][font=黑体].[/font][/font][font='Times New Roman'][[/font][font=黑体][font=Times New Roman]9[/font][/font][font='Times New Roman']][/font][font=黑体] [font=黑体]王亮,穆培源,桑伟,等.新疆小麦品种籽粒硬度及[/font][font=Times New Roman]Puroindoline[/font][font=黑体]基因等位变异的分子检测[/font][font=Times New Roman][J][/font][font=黑体].麦类作物学报。[/font][font=Times New Roman]2010[/font][font=黑体],[/font][font=Times New Roman]30(1)[/font][font=黑体]:[/font][font=Times New Roman]17-22[/font][font=黑体].[/font][/font][font='Times New Roman'][[/font][font=黑体][font=Times New Roman]10[/font][/font][font='Times New Roman']][/font][font=黑体] [font=黑体]赵新,王步军.不同硬度小麦品质差异的分析[/font][font=Times New Roman][J][/font][font=黑体].麦类作物学报,[/font][font=Times New Roman]2009[/font][font=黑体],[/font][font=Times New Roman]29(2)[/font][font=黑体]:[/font][font=Times New Roman]246[/font][font=黑体]—[/font][font=Times New Roman]251[/font][font=黑体]。[/font][/font]
硫醇硫测定仪怎么去的电位突变范围?
我想请问一下有没有做“致突变”相关的仪器设备
广东:首次发现新冠病毒巴西突变株,28名密切接触者已隔离
MSDS中物质如何判断是致癌,致基因突变物质?如果有SVHC中物质,如何判断符合性?
中国科技网讯 DNA序列中最轻微的变异也会影响深远,无论对研究还是医学应用,可靠识别这些序列都非常重要。据物理学家组织网近日报道,美国华盛顿大学和莱斯大学研究人员合作,开发出一种荧光DNA探测分子,能检查出一段目标DNA链中单个碱基的变化。而这些微小突变可能是造成某些疾病的根源,或耐抗生素细菌的原因。这一成果有助于诊断和治疗像癌症、肺结核这样的疾病。相关论文发表于7月28日的《自然·化学》杂志网站上。 不同的DNA序列为不同生物设定了独特的基因标记。现代基因组学研究表明,仅一个碱基对的变化都足以引发严重的生物后果,可能决定了一种疾病能否被治愈,也解释了疾病的突发或某些疾病对常规抗生素治疗无效的原因。论文领导作者、华盛顿大学电力工程和计算机科学与工程副教授乔治·塞利格说,比如造成肺结核的细菌有很强的耐药性,这种能力通常来自其基因序列中的少量突变。现在,人们已能预先查出这种突变。 “我们真正改进了以往的方法。”塞利格说,“新方法不需要任何复杂的反应或添加酶,就只用DNA。这意味着无论温度及其他环境变量怎样变化,该方法都是稳定的,所以很适合用于低资源设置中的诊断。” 这种探测分子经过专门设计,采用了新的编程机制,能与一个可疑的DNA序列结合,对其双螺旋链生成互补的DNA序列。把含有两种序列的分子在盐水试管中混合,如果两条链的碱基对都是完好的,它们自然地匹配在了一起,探测分子会发出荧光;如果不发光,则意味着上面有碱基对发生了突变。与以往技术不同的是,探测分子会检查目标DNA双螺旋的两条链是否发生了突变,而不是一条,这使检验更加全面具体。 此外,探测分子由许多寡核苷酸构成,克服了合成上的局限,可以探测更长的DNA序列中更详细的变异信息,达到200个碱基对,而现有探测突变的方法只能检查20个。 目前,研究人员与华盛顿大学商业化中心一起对该技术提出了专利申请,他们希望把这种技术和诊断试纸结合用于疾病测试。(常丽君) 《科技日报》(2013-8-7 二版)
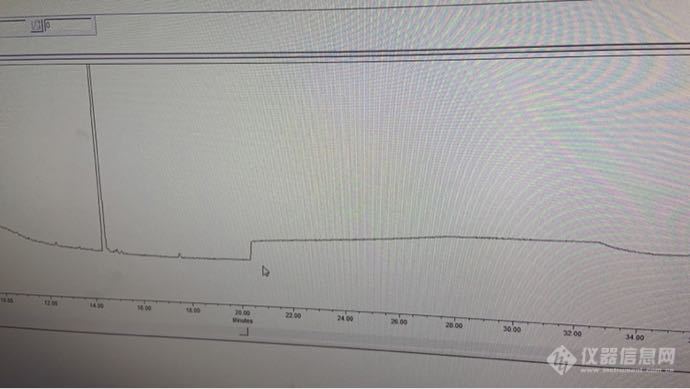
各位大佬,请问这种垂直上升的信号突变是什么原因造成的条件:安捷伦1260,dad检测器,同一个方法,一个序列中只可能有一针出现也可能一针都没有,我在不同的序列中寻找发现每次出现这种垂直的保留时间都不同,并且不止一台液相有这种情况,,并且压力也正常[img=,690,388]https://ng1.17img.cn/bbsfiles/images/2020/06/202006101637044347_7090_4134806_3.png[/img]
[b][b]CRISPR基因编辑技术遭遇迄今最大安全性质疑,这事您怎么看?科学技术造福人类,是否存在一个发展的界限?是否存在一些不可逾越的边界?[/b][/b]据《新科学家》杂志网站5月30日报道,美国科学家通过全基因组测序发现,CRISPR基因编辑技术能引起基因组内大量非靶标区内的基因发生突变,包括1500多种单核苷酸突变和100多种大片段序列的敲入和敲除。发表在《自然方法学》杂志上的这一论文表明,CRISPR的脱靶效应可能远超人们此前的估计。CRISPR基因编辑技术因其快速和高精准等特点,成为研究基因与疾病关系的热门之选,并因其能敲入新基因、敲除或修复受损基因,为基因疗法带来了更大希望。但最新论文共同作者、哥伦比亚大学医学中心病理学和细胞生物学副教授斯蒂芬曾认为,随着临床试验的相继展开,科学界是时候慎重考虑CRISPR技术脱靶效应的潜在风险了。资讯链接:CRISPR基因编辑技术遭遇迄今最大安全性质疑 [url]http://www.instrument.com.cn/news/20170601/220937.shtml[/url]
大家好最近在做DNA点突变的研究,由于基因很大,有七十几个外显子,如果一一扩增测序的话成本和时间上都不划算.所以在考虑能否用质谱的方法,或者其他什么分析的方法先进行下初筛。和标样比较,如果看到了某个峰有异常的话,再专门扩增这个外显子去测序外显子一般200-300bp。想请教大家,不知道用质谱的方法可不可以?另外,国内有哪里用质谱进行DNA序列的研究的吗?我知道用MALDI-TOF-TOF这个进行SNP的研究,但是对于我们这个200-300bp来说量程上就太小了。期待大家的回答~~~~
食品安全国家标准 细菌回复突变试验
求《计量技术》2016年第11期:《居民用户智能电能表电能数据突变的产生和预防》一文,谢谢!

走基线时,基线反向突变,有很多毛刺,是什么原因造成的?是进样器衬管脏了,还是检测器脏了,两个都要清洗吗?还有其他的原因吗?http://ng1.17img.cn/bbsfiles/images/2011/03/201103161838_283283_1621482_3.jpg
迄今为止,虽然在人类中只发生孤立的H5N1禽流感病毒(H5N1 bird flu virus)感染病例,但是这种感染并没有发生广泛的传播,这是因为该病毒不能够在人鼻子中有效地复制。在一项新的研究中,来自英国肯特大学和伦敦帝国理工学院的研究人员发现H5N1禽流感病毒发生较小的基因突变就能够在哺乳动物鼻子中更容易复制。相关研究结果于2013年3月13日在线发表在Journal of General Virology期刊上,论文标题为“Mutations in hemagglutinin that affect receptor binding and pH stability increase replication of a PR8 influenza virus with H5 HA in the upper respiratory tract of ferrets and may contribute to transmissibility”。这一发现支持了2012年发表的一项争议性研究的结论:只需几次基因突变就能够让禽流感在雪貂(常被用作人类流感病毒感染的研究模型)之间传播。
文献:遗传性脊髓小脑型共济失调的CAG三核苷酸突变检测 唐北沙[1] 夏家辉[2] [1]湖南医科大学汀雅医院神经病研究所 [2]中国医学遗传学国家重点实验室 《中华医学遗传学杂志》 1999年第16卷第5期
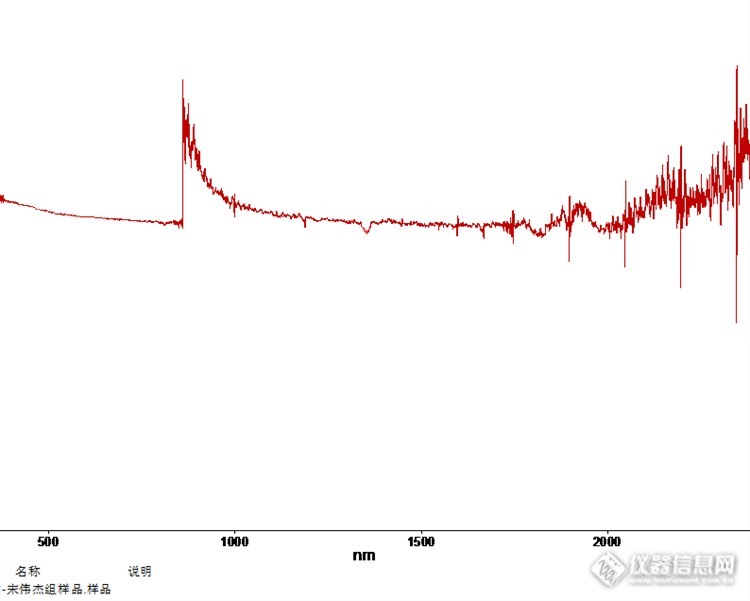
单位有台Lambda950型紫外,测反射率在860nm处切换检测器时有3%左右的突变,不知道是什么原因,请高手指点?另外有哪个单位也有Lambda950的紫外吗?想联系交流一下http://ng1.17img.cn/bbsfiles/images/2015/08/201508211811_562100_3024213_3.png
食品安全国家标准 体外哺乳类细胞HGPRT基因突变试验
请问通常快速筛选的灵敏度和检出限要比定量检测方法高还是低呢?常用的快速筛选方法都有什么呢?
食品安全国家标准 体外哺乳类细胞TK基因突变试验
20世纪30年代臭名昭著的劫匪约翰-狄林杰为了不让警方发现犯罪现场的指纹与他的指纹一样,不惜忍受巨大的痛苦,利用硫酸把指纹烧掉。但是对我们来说,没有指纹是一件非常可怕的事情,它会在边界控制和证明身份时造成很多麻烦。科学家目前已经确定可以导致一些人天生没有指纹的基因。http://www.bioon.com/biology/UploadFiles/201109/2011092211413775.jpg皮纹病患者天生没有指纹这种情况被称作皮纹病(adermatoglyphia)或者“入境延期病(Immigration Delay Disease)”,患有这种疾病的人天生手指肚上没有指纹。以色列特拉维夫大学的艾里-斯普雷彻教授获得的最新发现显示,一种基因突变造成这种与众不同的病变。一名瑞士女性试图穿越边境,进入美国,边境控制人员需要收集她的指纹。然而当这位女子告诉他们,她无法满足他们的要求,因为她根本没有指纹时,这些官员感到非常迷惑。这也促使医学界开始注意皮纹病。研究人员对这位女性以及她的另外9名没有指纹的家庭成员进行遗传分析。特拉维夫大学的科学家将存在这种情况的人的基因与没有这种情况的人的基因进行比对,确定基因变异到底发生在哪里。他们发现,SMARCAD1突变影响了指纹的形成。研究发现,没有指纹的人拥有的与皮肤发育有关的这种基因更少。现在科学家将能进一步研究这种基因是如何调控指纹的发展的。与DNA一样,每个人的指纹也是独一无二的,即使是同卵双胞胎也不例外,因此它们也成为破案和国际旅行管理等的重要鉴定工具。它们之所以会被当作确定身份的工具,是因为它们在受精24周后就会发育健全,而且整个一生都不会发生任何改变。目前全球仅有4个记录在案的家庭存在这种情况。斯普雷彻表示,不仅手指上长有带图案的皮肤,手掌、脚趾和足底也长着被称作皮纹的纹路。然而他说:“胚胎发育阶段导致指纹形成和拥有不同图案的因素目前大部分还不得而知。”除了缺少指纹外,这种情况还导致汗腺减少。指纹异常可能也是出现更严重的疾病的一种预警信号。该研究成果发表在《美国人类基因学期刊》上。2009年,一名中国女性通过整形手术改变她的指纹,想利用这种方法非法进入日本。东京警方表示,以前因签证过期被驱逐出日本的林荣(Lin Rong)花了1.5万美元在中国做了这项整形手术。警方认为,这种欺骗行为可能普遍存在,因为中国经纪人进行了大量指纹修改手术。这项手术涉及到摘除拇指和食指上的指纹,然后把它们嫁接到另一只手的手指上。
如果要推举出过去几年全民参与度最高的话题,那么“转基因”当之无愧。翻看近年的新闻报道,部分转基因农作物遭曝光,转基因是否应该强制标识等也被频频提及,这些都一度引起了广泛的讨论。而转基因食品是否安全,更成为人们关注和争议的焦点。外表能辨认转基因食品吗?外界有一种说法是通过颜色来辨别转基因食品。通过辣椒的黄、红色去分辨,但那些颜色异于常规的未必都是转基因产物。例如彩椒,是一个太空育种的产品,是突变,跟我们转基因的原理是一样的。另一种说法是通过外形来辨别。比如我们的圣女果,就外形而言与普通的番茄有很大差异,实际上却是从以色列引进的一个品种。如此看来,单看颜色或外形,是无法确认是否为转基因食品的。如何能辨认转基因食品呢?通过检测公司可以快速轻松地分辨转基因食品。基于食品的种类、加工方式的不同以及在食品中含有的相应的转基因片段的不同,适用合适的、高效的、精确地检测方法和手段。通过PCR技术,经过罗氏检测仪对待检测的转基因食品的DNA进行适当的扩增,通过其有无特定长度和序列的DNA序列和片段来判定是否为转基因食品。