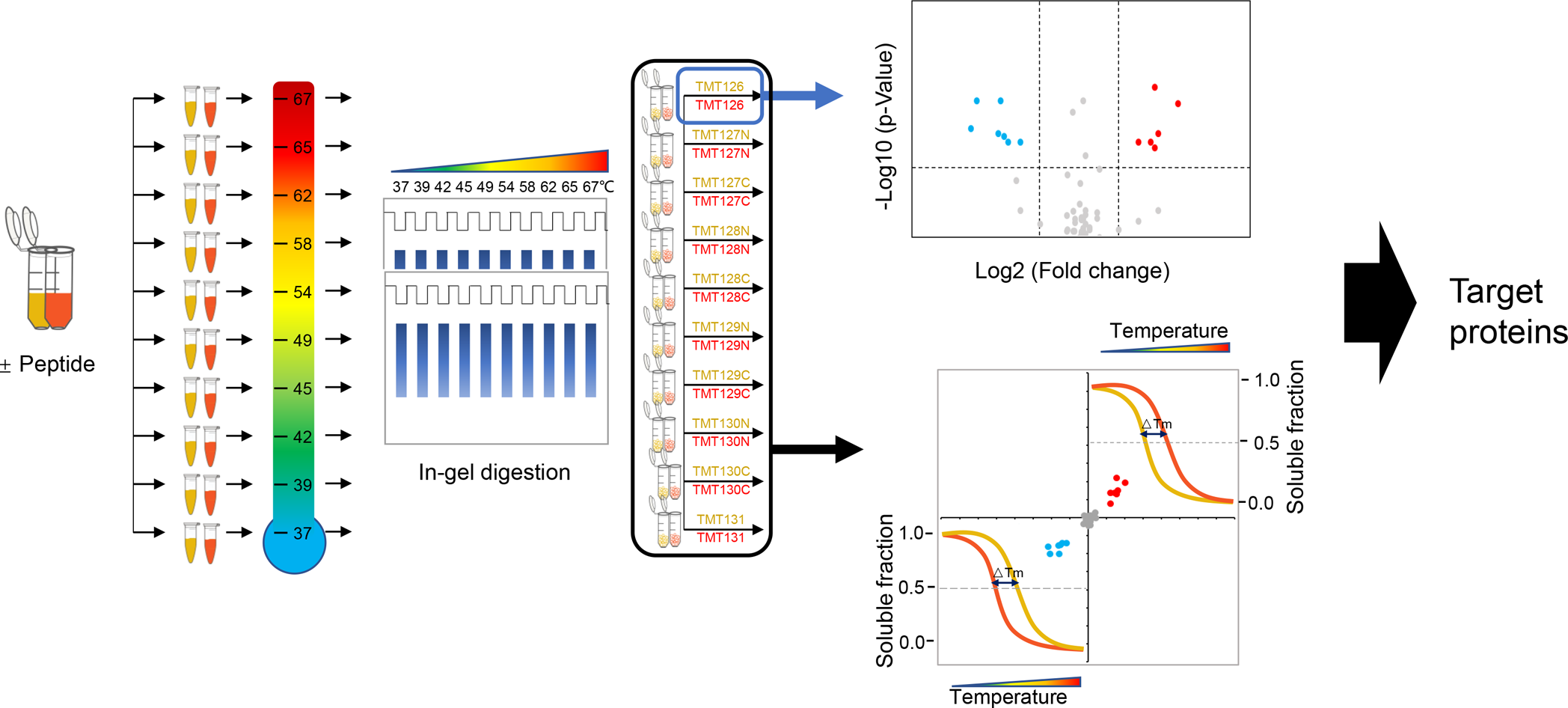
蛋白质测序技术发展漫谈(中)
前文回顾(点击):蛋白质测序技术发展漫谈(上)前面提到,基于质谱的蛋白质测序主要流程为:首先对蛋白质酶解得到肽段,经过LC-MS/MS分析得到相应的质谱数据,再使用测序软件根据质谱数据对肽段测序,最后对测序得到的肽段序列进行拼接。其中根据肽段的二级质谱图进行从头测序是其核心内容。目前已发展的肽段从头测序算法有三十余种,主要可以分为三类:图方法、穷举法和动态规划法,包括PEAKS[ 1]、pNovo系列[2]、Pepnovo[3]、Novor[4]等。 Muth[5]评估了Novor、PEAKS和PepNovo三种测序软件在实验数据集上测序的准确度,这三款软件对肽段的测序准确度最高只有35%。这是由于质谱谱图中存在着噪声和干扰离子,无法有效的区分谱图中可用于肽段测序的碎片离子[6],使得精准解析谱图的难度增加且耗费大量的时间。基于碎片离子的蛋白质组稳定同位素标记定量方法通过在细胞培养或样品处理的过程中引入不同种类的同位素标记,混合后进行LC-MS分析。不同稳定同位素标记的相同序列肽段质量相同或相近,可在质谱中同时碎裂,形成成对的碎片离子[7]。借鉴该方法,可更好的区分并提取用于测序的碎片离子,用于肽段的序列测定。Nie[8]在细胞培养时加入同位素标记的精氨酸和赖氨酸,再利用Lys-N和Arg-C对提取的蛋白质酶解,形成N端为精氨酸、C端为赖氨酸的等重肽段,在二级谱中可形成成对的b离子和成对的y离子,但这种标记方法只能在细胞水平标记,且通过两种蛋白酶酶解后只有少部分肽段质量相等并被鉴定到。Zhang[9]发展了部分等重肽段末端标记方法,使用Lys-C酶解后,肽段的C端为含有氨基的赖氨酸,再通过对两末端使用不同同位素标记,使得相同序列的肽段质量差为2 Da,在二级谱中产生了质量差为4 Da的成对b离子和质量差为6 Da的成对y离子,为使肽段能够碎裂在同一张谱图中,质谱的分离窗口需要被放大到4 m/z甚至更多[10],但放大分离窗口会导致更多的质量相近的肽段发生共碎裂,谱图会变得更加复杂难以解析,增加了从头测序的难度。为此,我们开发了一种基于二甲基化标记和胰蛋白酶催化18O标记的肽段末端准等重标记(Pseudo Isobaric Peptide Termini Labelling,PIPTL)从头测序方法 [11](图1)。经该方法进行同位素标记后,序列相同的肽段质量仅相差0.0166 Da,这些准等重肽段无需扩大质谱分离窗口即可在质谱中同时碎裂,产生成对的b离子和成对的y离子;基于发展的PIPTL-Novo测序算法,根据不同系列碎片离子质量差可快速准确提取并区分b/y离子,再对b/y离子进行测序分析,从而实现对肽段的准确测序。以牛血清白蛋白为研究对象,对肽段从头测序的准确度进行评价,测序准确率为95.5%;最后将此从头测序方法应用于对单克隆抗体赫赛汀重链和轻链的序列测定,对赫赛汀的酶解肽段从头测序准确率为93.6%。图1 基于二甲基化和胰蛋白酶催化18O标记的PIPTL-Novo策略参考文献[1] Ma B, Zhang K, Hendrie C, et al. PEAKS: powerful software for peptide de novo sequencing by tandem mass spectrometry. Rapid Commun Mass Spectrom, 2003, 17(20): 2337-42.[2] Yang H, Chi H, Zhou W-J, et al. Open-pNovo: de novo peptide sequencing with thousands of protein modifications. J Proteome Res, 2017, 16(2): 645-54.[3] Frank A M, Savitski M M, Nielsen M L, et al. De novo peptide sequencing and identification with precision mass spectrometry. J Proteome Res, 2007, 6(1): 114-23.[4] Ma B. Novor: real-time peptide de novo sequencing software. J AmSoc Mass Spectrom, 2015, 26(11): 1885-94.[5] Muth T, Renard B Y. Evaluating de novo sequencing in proteomics: already an accurate alternative to database-driven peptide identification? . Brief Bioinform, 2018, 19(5): 954-70.[6] Lu B, Chen T. A suboptimal algorithm for de novo peptide sequencing via tandem mass spectrometry. Journal of Computational Biology, 2003, 10(1): 1-12.[7] Merrill A E, Coon J J. Quantifying proteomes and their post-translational modifications by stable isotope label-based mass spectrometry. Curr Opin Chem Biol, 2013, 17(5): 779-86.[8] Nie A-Y, Zhang L, Yan G-Q, et al. In vivo termini amino acid labeling for quantitative proteomics. Anal Chem, 2011, 83(15): 6026-33.[9] Zhang S, Shan Y, Zhang S, et al. NIPTL-Novo: Non-isobaric peptide termini labeling assisted peptide de novo sequencing. J Proteomics, 2017, 154(40-8.[10] Hennrich M L, Mohammed S, Altelaar A M, et al. Dimethyl isotope labeling assisted de novo peptide sequencing. J Am Soc Mass Spectrom, 2010, 21(12): 1957-65.[11] 杨超,刘健慧,张玮杰等,基于末端准等重同位素标记的肽段从头测序方法. 分析化学, 2021, 49 (03), 366-376.作者简介:中国科学院大连化学物理研究所 单亦初副研究员1997年于中国科学技术大学获理学学士学位。2002年于中国科学院大连化物所获理学博士学位。2002年10月至2009年5月在德国马普协会马格德堡研究所、美国德克萨斯大学医学院及澳大利亚弗林德斯大学工作。2009年7月应聘到中国科学院大连化物所任副研究员。主持多项研究课题,包括国家重点研发计划子课题、国家自然科学基金面上项目等。已在Analytical Chemistry、Journal of Proteome Research、Journal of Chromatography A等杂志发表论文近80篇。主要研究方向包括蛋白质组鉴定和蛋白质组相对及绝对定量、蛋白质翻译后修饰富集和鉴定、蛋白质组末端肽富集和鉴定、蛋白质相互作用分析、蛋白质全序列从头测定及药物靶蛋白筛选。(本文经授权发布,仅供读者学习参考)专家约稿招募:若您有生命科学相关研究、技术、应用、经验等愿意以约稿形式共享,欢迎邮件投稿或沟通(邮箱:liuld@instrument.com.cn)。中国临床质谱产业化发展论坛(点击报名)仪器信息网联合浙江省先进质谱技术与分子检测重点实验室、宁波大学质谱技术与应用研究院,共同举办“第六届中国质谱产业化发展论坛——临床质谱产业化发展”,在2021年第十五届中国科学仪器发展年会(ACCSI 2021)召开同期,邀请临床质谱业内专家、国内质谱企业、第三方医学实验室、医院专家代表,共同就中国临床质谱技术与产业化发展等话题展开探讨、答疑解惑,为中国临床质谱产业链上中下游三方搭建互动交流平台,助力中国临床质谱产业发展,进一步优化和拓展临床质谱产业市场,共同促进中国质谱产业健康快速发展。