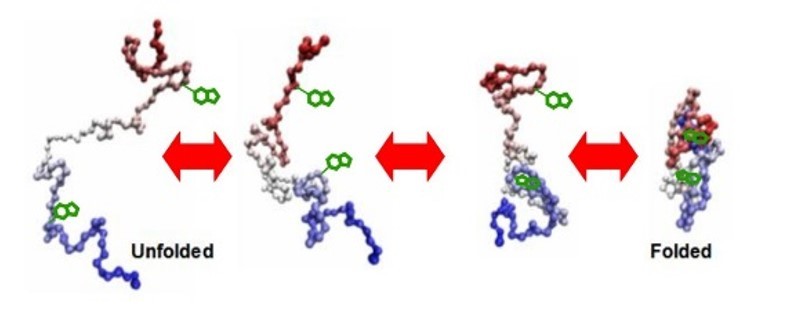
蛋白质测序技术发展漫谈(续)——基于荧光、纳米孔的单分子蛋白质测序
前文回顾(点击查看):蛋白质测序技术发展漫谈(上篇);蛋白质测序技术发展漫谈(中篇);蛋白质测序技术发展漫谈(下篇)前面描述了目前成熟的蛋白质测序方法,并对最流行的基于质谱的蛋白质测序方法进行了综述。非质谱依赖的蛋白质测序手段,除了几十年前发展的基于Edman降解法通过气相或液相色谱测序的方法,最近热门领域的方法主要包括基于荧光或纳米孔的单分子蛋白质测序,代表了未来的发展方向。基于纳米孔单分子蛋白质测序方法纳米孔测序(nanopore sequencing)法是借助电泳驱动力使待测单个分子逐一通过纳米孔,通过检测纳米孔截面的电流变化来实现对序列的测定。纳米孔测序最初在1996年被提出,通过膜通道检测多核苷酸序列,也就是单分子DNA的测序[1]。随着使用纳米孔对单分子DNA测序技术的逐渐成熟[2-5],纳米孔技术也被应用在单分子蛋白质的鉴定上。对于DNA来说,其二级结构和电荷相对比较一致,它的聚合物比较容易处理,而且仅由四种碱基组成,单分子DNA测序比较简单。相比之下,蛋白质分子由20种氨基酸组成,并且蛋白的电荷和疏水性多变,还存在大量的二级和三级结构,因此基于纳米孔技术对蛋白质的鉴定要比DNA困难很多[6]。当前的基于纳米孔对蛋白质分析的主要探索方向是通过寡核苷酸适配子或抗体等亲和分子对纳米孔进行功能化,当蛋白质或肽段分子通过纳米孔时,由于不同氨基酸在纳米孔附近的结合或通过会引起不同幅度的电流变化,基于这些变化就可以确定氨基酸的种类,从而逐个得到所测蛋白质或肽段的序列信息(图1)。图 1 借助纳米孔的横向电流检测单分子蛋白质[2]牛津大学的Hagan Bayley[7]团队将单个α-血溶素蛋白孔插入两侧带有电极的膜中,磷酸化的蛋白质在DNA寡核苷酸的牵引下展开,并穿过纳米孔,通过记录纳米孔的电流变化区分出了202个磷酸化蛋白质的4种不同亚型,但无法鉴定蛋白质的一级结构。Francesco[8]团队将蛋白质或氨基酸吸附在金纳米星上,并施加电等离子体力将粒子推进并约束在金纳米孔内,利用金纳米星与金纳米孔壁之间的单个热点,实现了单分子表面增强拉曼散射(SERS)探测,用于检测氨基酸,并且可以分辨仅含有两个不同氨基酸的单个多肽分子抗利尿激素和催产素。Cao等[9]通过单个定点突变,在具有锥形识别位点的耻垢分枝杆菌孔蛋白A(MspA)的纳米孔内腔中引入了甲硫氨酸,从而将该反应有目的的移植到了MspA纳米孔最尖锐的识别位点,并观测到了相应的单分子反应信号。该纳米孔可以引入更多的离子电流,从而放大检测信号,其狭窄的识别位点则提供了更高的空间分辨率,大大削弱了周围氨基酸的干扰,从而拓宽生物纳米孔的单分子检测功能,有望推进基于孔道的单分子蛋白质测序研究。Ouldali[10]研究团队研发出了一种新型气溶素纳米孔,此纳米孔借助将氨基酸附着在聚阳离子载体上,使氨基酸在纳米孔上停留时间变长,并检测其通过纳米孔时电流的变化,最终可识别出组成蛋白质的15种氨基酸,也能检测到组成蛋白质的其余5种氨基酸的电流变化,但是无法对其进行区分。虽然只是对氨基酸进行识别,但作者设想通过对蛋白或者肽段末端氨基酸逐个降解,利用纳米孔技术鉴定从末端释放出来的氨基酸,从而对蛋白质或肽段序列进行测定。Zhao[11]等将一对金属电极分隔在约2nm的孔洞旁,当氨基酸线性穿过这种纳米孔的时候,每一个氨基酸都会完成一个回路,并反馈出相应的电信号,常见的20种氨基酸在通过纳米孔时都可以产生电信号。有的氨基酸需通过大约50种不同信号特征被鉴定,但绝大多数的氨基酸仅需要不到10个信号特征被鉴别。这种方法不仅能够高可信度的鉴定氨基酸,还能区分翻译后修饰的氨基酸(肌氨酸)及其前体(甘氨酸)、区分同分异构体的亮氨酸与异亮氨酸、区分对应对映异构体的氨基酸镜像分子L-天冬酰胺和D-天冬酰胺。此技术被应用于对两条由四个氨基酸组成的短肽(GGGG 和GGLL)进行测序,单分子短肽穿过纳米孔,孔道两边电极记录每个氨基酸通过时产生的电信号,通过测序算法,识别代表不同氨基酸的特征信号,从而得到短肽的序列。基于纳米孔单分子蛋白测序目前还属于初步发展阶段,除了需要根据电信号准确区分组成蛋白质的氨基酸以外,另一个关键是设计可一次拉动一个蛋白质或氨基酸穿过纳米孔的“马达”。为了让蛋白质或肽段顺利穿过纳米孔,研究者们在蛋白质一端添加了一串带有负电的氨基酸或者一段短DNA,用氨基酸或DNA链拉动蛋白质,可以使一些蛋白质打开折叠并顺利穿过纳米孔,但另一些复杂折叠的蛋白需要更多拉力,于是研究者在引导序列上添加了可以打开折叠的ClpX的识别位点[12]。这个系统能够将简单折叠的目标蛋白牵引过纳米孔,但对于折叠非常紧密的蛋白质仍要使用变性剂来打开折叠。基于纳米孔技术对单分子肽段或蛋白质测序目前还停留在对氨基酸鉴定和对短肽的区分阶段,还不能实际应用于对蛋白质的测序。虽然纳米孔测序具有高通量、对样品需求量少的优点,但是现有的纳米孔过大,失去了对氨基酸的区分能力,同时蛋白质分子通过孔道过快,加大了对信号读取难度;其次由于需要将蛋白的三级和二级结构破坏掉,纳米孔道需要能够耐受非常苛刻的化学和力学条件;第三,由于蛋白带电不均匀,控制其穿孔的速率也非常困难。所以目前的方法还不能准确的测得蛋白质的序列,基于纳米孔的单分子蛋白质测序技术还有很大的发展空间。基于荧光的单分子蛋白质测序方法基于荧光的单分子蛋白质测序同纳米孔测序一样,都可以对极少量蛋白质样品进行检测,其原理是先将蛋白质酶解成肽段,对肽段中特定氨基酸选择性标记不同的荧光基团[13],对不同氨基酸上的荧光进行观察,从而确定肽段部分氨基酸序列,再将这些序列与蛋白质组序列比对,即可确定肽段的来源蛋白(图2)。图 2 基于荧光的单分子蛋白测序流程[14]。Ginkel[15] 和Yao [16]都利用ClpXP蛋白酶辅助对肽段进行选择性荧光标记,可对序列中的赖氨酸和半胱氨酸进行标记,通过Förster共振能量转移依次读出被标记的肽段的氨基酸的信号。Swaminathan[14] 将蛋白质酶解成肽段,再将肽段固载到玻璃片上[17],使用特定荧光基团分别对肽段中的赖氨酸和半胱氨酸选择性标记,通过Edman降解技术对固载的肽段进行降解,每次降解后都使用全内反射荧光(TIPF)显微镜进行观测。如果被标记的赖氨酸和半胱氨酸在Edman降解中从肽段N端释放出来,被标记的以上两种氨基酸的位置就会被检测到。同时还发展了用于监测单个肽荧光强度的图像处理算法,并对误差源进行分类和建模,可以测得序列中部分氨基酸的信息。将测得的部分序列与参考蛋白质组序列比对,即可确定肽段的来源蛋白,通过与蛋白质组序列比对,可以鉴定到在人源蛋白质组中的绝大多数蛋白质。基于荧光单分子蛋白测序技术主要有三方面难点,一方面在于目前仅能对赖氨酸和半胱氨酸等几种氨基酸进行特异性荧光基团的标记,无法对所有氨基酸都进行标记;第二个难点是Edman降解是在强酸或强碱的环境中进行,对这些荧光基团的稳定性要求很高;第三个难点是对后期图像处理有较高的要求,如果序列中每个氨基酸都标记上不同的荧光基团,且发光峰易交叠难分辨,这给荧光处理算法带来了难度。因此,基于荧光的单分子蛋白测序技术虽然可以对极微量蛋白质样品分析,但目前仅能测得部分氨基酸序列,对蛋白质全序列的测定目前尚不能实现。[1] Kasianowicz J J, Brandin E, Branton D, et al. Characterization of individual polynucleotide molecules using a membrane channel [J]. Proceedings of the National Academy of Sciences, 1996, 93(24): 13770-13773.[2] Branton D, Deamer D W, Marziali A, et al. The potential and challenges of nanopore sequencing [J]. Nanoscience and technology: A collection of reviews from Nature Journals, 2010: 261-268.[3] Laver T, Harrison J, O’neill P, et al. Assessing the performance of the oxford nanopore technologies minion [J]. Biomolecular detection and quantification, 2015, 3: 1-8.[4] Karlsson E, Lärkeryd A, Sjödin A, et al. Scaffolding of a bacterial genome using MinION nanopore sequencing [J]. Sci Rep, 2015, 5(1): 1-8.[5] Huang S, Romero-Ruiz M, Castell O K,et al. High-throughput optical sensing of nucleic acids in a nanopore array [J]. Nature nanotechnology, 2015, 10(11): 986-991.[6] Nivala J, Marks D B, Akeson M. Unfoldase-mediated protein translocation through an α-hemolysin nanopore [J]. Nat Biotechnol, 2013, 31(3): 247-250.[7] Rosen C B, Rodriguez-Larrea D, Bayley H. Single-molecule site-specific detection of protein phosphorylation with a nanopore [J]. Nat Biotechnol, 2014, 32(2): 179.[8] Huang J, Mousavi M, Giovannini G, et al. Multiplexed Discrimination of Single Amino Acid Residues in Polypeptides in a Single SERS Hot Spot [J]. Angewandte Chemie 2020, 59(28): 11423-11431.[9] Cao J, Jia W, Zhang J, et al. Giant single molecule chemistry events observed from a tetrachloroaurate (III) embedded Mycobacterium smegmatis porin A nanopore [J]. Nature communications, 2019, 10(1): 1-11.[10] Ouldali H, Sarthak K, Ensslen T, et al. Electrical recognition of the twenty proteinogenic amino acids using an aerolysin nanopore [J]. Nat Biotechnol, 2020, 38(2): 176-181.[11] Zhao Y, Ashcroft B, Zhang P, et al. Single-molecule spectroscopy of amino acids and peptides by recognition tunnelling [J]. Nature nanotechnology, 2014, 9(6): 466-473.[12] Nivala J, Mulroney L, Luan Q, et al. Unfolding and Translocation of Proteins Through an Alpha-Hemolysin Nanopore by ClpXP [M]. Nanopore Technology. Springer. 2021: 145-155.[13] Hernandez E T, Swaminathan J, Marcotte E M, et al. Solution-phase and solid-phase sequential, selective modification of side chains in KDYWEC and KDYWE as models for usage in single-molecule protein sequencing [J]. New J Chem, 2017: 462-469.[14] Swaminathan J, Boulgakov A, Hernandez E, et al. Highly parallel single-molecule identification of proteins in zeptomole-scale mixtures [J]. Nat Biotechnol, 2018, 36(11): 1076-1082.[15] Ginkel J V, Filius M, Szczepaniak M, et al. Single-molecule peptide fingerprinting [J]. Proceedings of the National Academy of Sciences, 2018, 115(13): 3338-3343.[16] Yao Y, Docter M, Ginkel JV, et al. Single-molecule protein sequencing through fingerprinting: computational assessment [J]. Phys Biol, 2015, 12(5): 055033.[17] Howard C, Floyd B, Bardo A, et al. Solid-Phase Peptide Capture and Release for Bulk and Single-Molecule Proteomics [J]. ACS Chem Biol, 2020, 15(6): 1401-1407.作者简介:中国科学院大连化学物理研究所 单亦初副研究员1997年于中国科学技术大学获理学学士学位。2002年于中国科学院大连化物所获理学博士学位。2002年10月至2009年5月在德国马普协会马格德堡研究所、美国德克萨斯大学医学院及澳大利亚弗林德斯大学工作。2009年7月应聘到中国科学院大连化物所任副研究员。主持多项研究课题,包括国家重点研发计划子课题、国家自然科学基金面上项目等。已在Analytical Chemistry、Journal of Proteome Research、Journal of Chromatography A等杂志发表论文近80篇。主要研究方向包括蛋白质组鉴定和蛋白质组相对及绝对定量、蛋白质翻译后修饰富集和鉴定、蛋白质组末端肽富集和鉴定、蛋白质相互作用分析、蛋白质全序列从头测定及药物靶蛋白筛选。(本文经授权发布,仅供读者学习参考)专家约稿招募:若您有生命科学相关研究、技术、应用、经验等愿意以约稿形式共享,欢迎邮件投稿或沟通(邮箱:liuld@instrument.com.cn )。