
方案摘要
方案下载| 应用领域 | 医疗/卫生 |
| 检测样本 | 其他 |
| 检测项目 | 生化检验>蛋白, 定性定量分析 |
| 参考标准 | - |
MALDI Guided SpatialOMx,即基于质谱成像定位的空间多组学分析,完美的将MALDI成像技术和传统LC-MS/MS组学流程结合在了一起,它的特点是利用了MALDI成像技术能在分子原位对生物分子进行定位的特点,在生物组织的内部锁定目标微区(ROI,Region of Interests),在对该目标微区实施激光微切割(LCM,laser capture microdissection)后,进行LC-MS/MS组学分析,从而实现了深度的蛋白组信息挖掘。
荷兰马斯特里赫特大学的Ron M.A. Heeren教授实验室利用空间定位组学工作流程,对来自于乳腺癌患者的肿瘤组织,进行了蛋白质分子特征的深度研究。

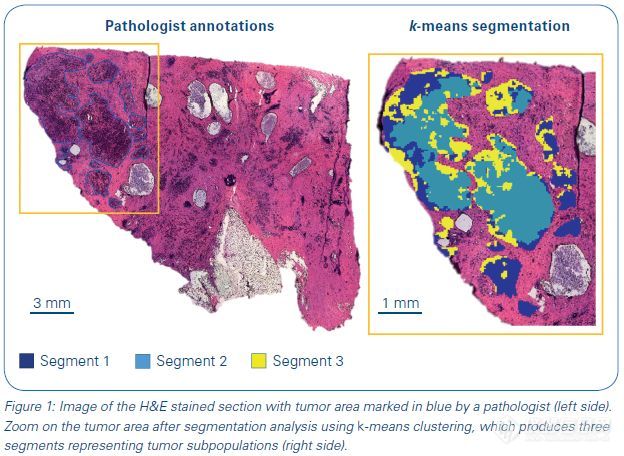
布鲁克timsTOF fleX质谱仪同时具备MALDI成像功能和LC-MS/MS组学分析功能,成功地实现了空间定位组学工作流程中的关键步骤。与传统组学的“整体匀浆方法”相比,空间定位组学充分地考虑到乳腺癌组织中不同的肿瘤亚细胞群之间的差异性,并分别对各个肿瘤亚细胞群体进行了有针对性的、深度的蛋白组分析。以上的众多优点使得空间定位组学为精准地揭示乳腺癌的致病机理奠定了坚实的基础。


4D-脂质组学高通量工作流程
PASEF应用于COVID-19的高通量定量大规模血浆蛋白质组学研究
相关产品
布鲁克neofleX™ MALDI-TOF/TOF 空间成像质谱仪
布鲁克 timsTOF Ultra 2 捕集离子淌度质谱系统
布鲁克 timsTOF HT 捕集离子淌度质谱仪
PepSep™ 色谱柱
布鲁克scimaX™磁共振质谱仪
布鲁克 timsTOF fleX with MALDI-2
布鲁克timsTOF Pro 2 捕集离子淌度质谱
布鲁克 timsTOF fleX 组学和成像质谱系统
布鲁克ultrafleXtreme MALDI TOF/TOF质谱
布鲁克 timsTOF MALDI PharmaPulse
autoflex maX MALDI-TOF(TOF) 质谱
布鲁克solariX磁共振质谱仪
布鲁克 maXis II 飞行时间串联质谱
布鲁克 compact 高分辨飞行时间质谱
布鲁克impact II四极杆飞行时间质谱仪

关注
拨打电话

留言咨询