中科院崔丽研究员最新进展:耐药基因的水平基因转移研究进展,单细胞分析显身手
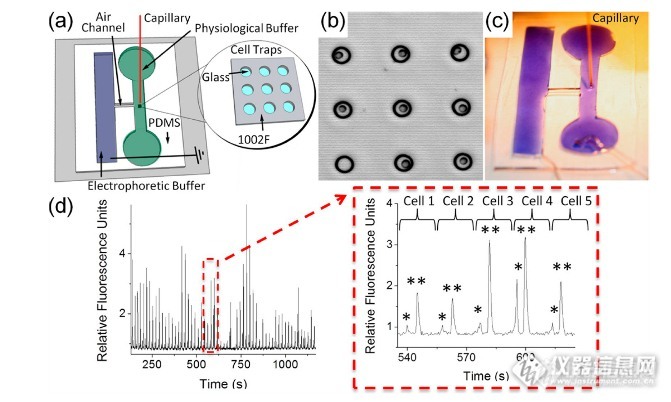
p style=" text-align: justify text-indent: 2em " span style=" text-indent: 2em font-size: 16px " /span /p p style=" text-align: justify text-indent: 2em " span style=" text-indent: 2em text-align: justify " 10月28日,中科院城市环境研究所崔丽研究员团队在期刊《Analytical Chemistry》上发表耐药基因水平基因转移最新研究。文章标题为“ /span i style=" text-indent: 2em text-align: justify " Phenotypic Tracking of Antibiotic Resistance Spread via Transformation from Environment to Clinic by Reverse D2O Single-Cell Raman Probing”。 /i br/ /p p img src=" https://img1.17img.cn/17img/images/202012/uepic/6221444b-ddc8-474b-b2fc-01abdfc691fd.jpg" title=" 企业微信截图_20201216144900.png" alt=" 企业微信截图_20201216144900.png" style=" text-align: center max-width: 100% max-height: 100% " / br/ /p p style=" text-align: justify text-indent: 2em " span style=" font-size: 16px " 原文链接: /span /p p style=" text-align: justify text-indent: 2em " a href=" https://pubs.acs.org/doi/10.1021/acs.analchem.0c03218?ref=pdf" target=" _blank" span style=" text-indent: 2em font-size: 16px text-decoration: underline " https://pubs.acs.org/doi/10.1021/acs.analchem.0c03218?ref=pdf /span /a /p p style=" text-align: justify text-indent: 2em " span style=" color: rgb(0, 0, 0) " strong 【研究背景 span style=" text-indent: 2em " 】 /span /strong /span /p p style=" text-align: justify text-indent: 2em " span style=" font-size: 16px font-family: 楷体, 楷体_GB2312, SimKai " 微生物耐药是一个危害公共卫生安全的全球性问题。耐药基因(antibiotic resistance gene,ARG)在环境或临床中出现并传播扩散。在这一过程中,水平基因转移(Horizontal gene transfer,HGT)现象起着不可忽视的作用。HGT的一个主要途径是转化(transformation)——受体细菌摄取胞外DNA(extracelluar DNA,eDNA)。这些eDNA多是细菌主动分泌或者裂解过程中释放的质粒DNA以及片段化DNA,常常携带着ARG。环境充当着ARG储库的角色,威胁着临床治疗。因此了解ARG从环境到临床转移的过程是必要的。 /span /p p style=" text-align: justify text-indent: 2em " span style=" font-size: 16px font-family: 楷体, 楷体_GB2312, SimKai " 在上述问题的研究中,常规方法依赖耐药菌的选择性培养或是报告基因标记eDNA技术。前者需要已知被筛选微生物的培养条件,且无法筛选出耐药但扩增不活跃的微生物。后者需要对eDNA等进行改造,不能反应实际的HGT情况。 /span /p p style=" text-align: justify text-indent: 2em " span style=" font-size: 16px font-family: 楷体, 楷体_GB2312, SimKai " 近年新兴了一种联合正向D2O标记的单细胞拉曼光谱方法用于微生物抗性区分。微生物利用环境中的D2O合成相关的生物分子,可以通过拉曼光谱的C-D带(2040-2300 cm sup -1 /sup )进行表征。抗性环境中敏感菌与耐药菌会表现出不同D2O摄入,进而表现出不同的C-D峰情况。然而微生物不仅摄入D2O,还会摄入其他含H元素的营养物质,这导致单位时间C-D谱带的增长较低。同时较短时间的孵育也降低了该方法的灵敏度。 span style=" font-size: 16px font-family: 楷体, 楷体_GB2312, SimKai color: rgb(0, 112, 192) " strong 作者团队改进并提出了反向D2O-单细胞-拉曼检测技术(reverse D2O single-cell Raman (Raman-rD2O) probing,Raman-rD2O)。微生物预先在D2O中孵育,后置换为含抗生素的H2O环境孵育并被检测。作者确定了该方法的有效性并利用该方法研究了HGT过程.。 /strong /span /span /p p style=" text-align: justify text-indent: 2em " span style=" font-size: 16px font-family: 楷体, 楷体_GB2312, SimKai " /span /p p style=" text-align: center" img style=" max-width:100% max-height:100% " src=" https://img1.17img.cn/17img/images/202012/noimg/94849f8b-b936-4174-925c-1ed3894541ed.gif" title=" ac0c03218_0007.gif" alt=" ac0c03218_0007.gif" / /p p style=" text-align: justify text-indent: 2em " span style=" color: rgb(0, 0, 0) " strong 【实验设计 span style=" text-indent: 2em " 】 /span /strong /span /p p style=" text-align: justify text-indent: 2em " 这项研究中,为了评价微生物耐药性,作者建立了Raman-rD2O方法,确定了其有效性,以及灵敏度。之后在大肠杆菌体系中确认了水平基因转移的情况。接着评价了与来自于复杂环境中质粒共孵育的大肠杆菌对各抗生素(meropenem,vancomycin,ampicillin,cefradine以及ciprofloxacin)的耐药情况,并确认了该方法比传统培养方法更加准确。最后提出了一种用于反映ARGs传播风险的计算方法。 /p p style=" text-align: justify text-indent: 2em " span style=" color: rgb(0, 0, 0) " strong 【结果讨论 span style=" text-indent: 2em " 】 /span /strong /span /p p style=" text-align: justify text-indent: 2em " span style=" color: rgb(0, 112, 192) " strong Raman-rD2O方法区分耐药菌以及敏感菌的灵敏度 /strong /span /p p style=" text-align: justify text-indent: 2em " 对colistin耐药的FIT10(B. cereus)以及colistin敏感的DH5α(E. coli)菌株进行D2O培养12h。转移至含抗生素colistin的无D2O环境进行培养,取不同时间点进行拉曼图谱采集,结果如图 1。在C-D峰处可以看出,耐药菌FIT10峰强逐渐变低,非耐药菌DH5α无明显变化。 /p p style=" text-align: center" img style=" max-width:100% max-height:100% " src=" https://img1.17img.cn/17img/images/202012/uepic/324d314b-6c4d-41d3-97cd-3f0d1f1ad776.jpg" title=" 002.jpg" alt=" 002.jpg" / /p p style=" text-align: center " strong 图1 /strong /p p style=" text-align: justify text-indent: 2em " 图 1. 预先标记同位素D微生物在含0.125 mg/L colistin的LB培养基中培养0、0.5、1、4、8小时后的单细胞拉曼光谱(a)耐药菌FIT10,(b)敏感菌DH5α。 /p p style=" text-align: justify text-indent: 2em " 采集含colistin培养条件下FIT10以及DH5α的单细胞拉曼图谱并统计C-D比值(CD/(CD + CH)),结果如图 2a-Ⅰ。另外比较了含ampicillin条件下的DH5α-Ampr以及DH5α数据,如图 2a-Ⅲ。可以看出该方法能够区分耐药菌以及敏感菌,且适用不同抗生素。 /p p style=" text-align: justify text-indent: 2em " 从图 2a-Ⅱ/Ⅲ以及2b数据,可以看出正向标记方法4h后才能观察到差异。因此反向标记方法是优于正向标记的。 /p p style=" text-align: center" img style=" max-width: 100% max-height: 100% width: 394px height: 526px " src=" https://img1.17img.cn/17img/images/202012/uepic/3db3bf0c-fe48-4a7e-8d06-4679a9ed238e.jpg" title=" 003.jpg" alt=" 003.jpg" width=" 394" height=" 526" / /p p style=" text-align: center " strong 图2 /strong & nbsp /p p style=" text-align: justify text-indent: 2em " 图 2. (a-Ⅰ,a-Ⅲ) 反向D2O方法下,不同时间点的colistin耐药FIT10、ampicilin耐药DH5α-Ampr以及DH5α的C-D比值统计结果;(a-Ⅲ,a-Ⅳ) 正向D2O方法下结果;(b) 1× MIC抗生素浓度下培养1h,反向D2O、正向D2O的单细胞拉曼图谱C-D区域结果。 /p p style=" text-align: justify text-indent: 2em " span style=" color: rgb(0, 112, 192) " strong span style=" text-indent: 2em " 利用Raman-rD2O方法揭示转化过程中ARGs的水平基因转移 /span /strong /span /p p style=" text-align: justify text-indent: 2em " 作者比较了实验组JM109(E . coli)以及对照组DH5α、DH5α-Ampr 菌C-D比值。JM109预先与携带bla基因(编码抗ampicilin蛋白)的pMD19-T质粒共孵育。从图 3可以看出,DH5α组维持高的C-D比值,DH5α-Ampr 组比值降至6.4%以下。而JM109组分为了高比值群体以及低比值群体。这意味着JM109组中部分微生物获得了bla基因并表达,从而能在含ampicilin环境中进行代谢生长。 /p p style=" text-align: justify text-indent: 2em " span style=" color: rgb(0, 112, 192) " strong HOOKE单细胞分选仪助力耐药基因水平基因转移研究 /strong /span /p p style=" text-align: justify text-indent: 2em " 为了验证这一假设,作者利用 strong 长光辰英科仪(HOOKE instruments)的单细胞分选仪(PRECI SCS) /strong ,对各样品进行了单个细胞分选。对分选的单个微生物进行全基因组扩增后,通过PCR验证bla基因的携带情况。从图 3b可看出,DH5α组无bla基因,DH5α-Ampr 组携带bla基因,部分JM109组携带bla基因。这一结果印证了bla基因转移的假设。 /p p style=" text-align: center" img style=" max-width:100% max-height:100% " src=" https://img1.17img.cn/17img/images/202012/uepic/32f0c549-ecc1-41a1-a8b5-dc7775175666.jpg" title=" 006.jpg" alt=" 006.jpg" / /p p style=" text-align: center " strong 长光辰英科仪(HOOKE instruments)单细胞分选仪HOOKE PRECI SCS /strong /p p style=" text-align: center " a href=" https://www.instrument.com.cn/zc/1835.html" target=" _blank" span style=" text-decoration: underline " (点击进入单细胞分析专场查看更多仪器信息) /span /a /p p style=" text-align: justify text-indent: 2em " 作者统计了不同采集个数下耐药菌个数并计算了转化频率(Transformation Frequency = 转化菌个数/(10× 拉曼采集总数),10为微生物1h时扩增倍数)。采集数目大于40时,转化频率趋近4.33 × 10 sup -2 /sup 。该结果与传统平板培养方法得到的5.07 × 10 sup -2 /sup 一致。 /p p style=" text-align: center" img style=" max-width: 100% max-height: 100% width: 398px height: 795px " src=" https://img1.17img.cn/17img/images/202012/uepic/5d4cd009-861e-4257-864f-d39b0a4467bb.jpg" title=" 004.jpg" alt=" 004.jpg" width=" 398" height=" 795" / /p p style=" text-align: center " strong 图3 /strong /p p style=" text-align: justify text-indent: 2em " 图 3. (a)预标记同位素D的E. coli DH5α(敏感对照),E. coli DH5α-Ampr(抵抗对照)以及转化了含bla基因质粒的接受菌E. coli ,经过含ampicillin的LB培养基孵育1h,采集拉曼光谱并统计C-D比率。每个点对应一个拉曼结果。阈值6.4%为未经过D标记的抗性转化子的C-D比值的平均值+3倍标准偏差;(b)PCR产物(针对质粒上bla基因)的电泳结果,1-3道为高C-D比值的敏感接受菌,4-6为小于10% C-D比值的耐药接受菌,7为不含质粒的E. coli DH5α,8为含bla基因的E. coli DH5α-Ampr;(c)质粒(携带bla基因)的转化频率(线)以及转化频率的标准偏差(柱)。利用单细胞Raman-rD2O方法对不同拉曼图谱采集个数下结果进行了统计。 /p p style=" text-align: justify text-indent: 2em " span style=" color: rgb(0, 112, 192) " strong 通过单细胞Raman-rD2O评价复杂环境中质粒介导的eARGs转化现象 /strong /span /p p style=" text-align: justify text-indent: 2em " 作者提取了土壤中的质粒并与E. coli共孵育。通过单细胞Raman-D2O,分析了不同抗生素下(meropenem,vancomycin,ampicillin,cefradine以及ciprofloxacin)的拉曼C-D比值。从图 4a可以看出,ampicillin,cefradine以及ciprofloxacin组出现了水平基因转移现象。考虑到土壤等环境中质粒对受体微生物增殖能力的影响,不同质粒编码导致同一抗性等问题,作者引入了传播效率(Spread Efficiency = 转化菌个数/拉曼采集总数)来评估传播风险。就图 4b结果来说,传播效率ampicillin(1.5 × 10 sup -1 /sup ) & gt cefradine(8.6 × 10 sup -2 /sup )和ciprofloxacin(6.7 × 10 sup -2 /sup ) & gt meropene(0)m和 vancomycin(0)。 /p p style=" text-align: justify text-indent: 2em " 作者同时计算了Raman rD2O方法的转化效率,ampicillin (1.9 × 10 sup -3 /sup )& gt ciprofloxacin (5.9 × 10 sup -4 /sup ) and cefradine (8.8 × 10 sup -4 /sup ) & gt meropenem and vancomycin (0) 。而传统平皿培养方法下的转化效率数值小于上述80-100倍。传统方法低估了HGT的程度,一方面存在携带抗性基因但在极端环境下不可培养鉴定的微生物(viable but nonculturable,VBNC),另一方面存在接受质粒导致扩增速率大幅降低(1000倍)的微生物。而在传统方法中过夜培养后,这种差异变得更加的明显。 /p p style=" text-align: center" img style=" max-width:100% max-height:100% " src=" https://img1.17img.cn/17img/images/202012/uepic/59c22524-2c5b-489a-8645-ba9dd52fd408.jpg" title=" 005.jpg" alt=" 005.jpg" / /p p style=" text-align: center " strong 图4 /strong /p p style=" text-align: justify text-indent: 2em " 图 4. (a) E. coli DH5α(敏感对照)C-D比值,以及接受菌E. coli 的C-D比值。接受菌经同位素D预标记后,与从土壤提取质粒进行共孵育。后分别在meropenem,vancomycin,ampicillin,cefradine以及ciprofloxacin条件下孵育1h。每一个点对应一个单细胞拉曼图谱。C-D比值小于6.4%的为不带D标记的耐受转化子;(b)携带抗meropenem,vancomycin,ampicillin,cefradine以及ciprofloxacin基因的质粒的传播效率。星号表示比较具有统计学意义(单因素方差分析ANOVA,P& lt 0.001)。 /p p style=" text-align: justify text-indent: 2em " span style=" color: rgb(0, 0, 0) " strong 【结论 span style=" text-indent: 2em " 】 /span /strong /span /p p style=" text-align: justify text-indent: 2em " 利用单细胞拉曼反向D2O检测方法能够可靠且快速的从表型上追踪耐药从环境到临床的扩散。方法具有很高的灵敏度,能够在大量受体菌种识别出那些少量的抗性转化子。单细胞水平的检测避免了繁琐的培养过程,可以直接计算传播效率,能够表征临床相关抗生素的不同风险。该方法后续未来可用于破解影响抗性质粒转移以及持续的因素。通过与基因型研究的联系,能更好的理解环境质体(environmental plastidome)传播风险。也是一种研究HGT机制的新手段。 /p p style=" text-align: justify text-indent: 2em " strong 关于作者 /strong /p p style=" text-align: center" img style=" max-width: 100% max-height: 100% width: 134px height: 170px " src=" https://img1.17img.cn/17img/images/202012/uepic/698827c5-ef1f-459f-935d-7b76ee9361ab.jpg" title=" 459b1093-fe12-4d67-bfb0-17b9f500ce02.jpg" alt=" 459b1093-fe12-4d67-bfb0-17b9f500ce02.jpg" width=" 134" height=" 170" / /p p style=" text-align: justify text-indent: 2em " span style=" font-family: 楷体, 楷体_GB2312, SimKai " 崔丽 (Li Cui),研究员,博士生导师,国家优秀青年科学基金获得者,中科院青年创新促进会会员。2002年获厦门大学化学系学士学位,2008年获厦门大学理学(物理化学专业)博士学位。2008.9-至今在中科院城市环境研究所工作,2010. 10评为副研究员,2019.01评为研究员。英国牛津大学(2015.8-2016.2),兰卡斯特大学(2015.2-2015.7)和瑞士伯尔尼大学访问学者(2009.11-2010.2)。至今已在化学和环境国际主流刊物J. Am. Chem. Soc., Anal. Chem.(7篇), Environ. Sci. Technol., Water Res,J Mem Sci, Soil Biol. Biochem,J Phys Chem等发表论文45篇。先后承担国家自然科学基金项目5项,科技部项目1项,省市基金3项。 strong 研究领域: /strong 环境分析化学(微生物为主),尤其是利用单细胞拉曼/表面增强拉曼光谱,并与稳定同位素标记技术联用,开展环境微生物的研究,如抗生素抗性、氮磷微生物利用过程、生物膜、纳米毒性等。 /span /p p style=" text-align: justify text-indent: 2em " strong 关于长光辰英 /strong /p p style=" text-align: justify text-indent: 2em " 长春长光辰英生物科学仪器有限公司(长光辰英科仪,Hooke Instruments)成立于 2017年,坐落于风景秀美的塞外春城长春,由海外归国团队、长春奥普光电技术股份有限公司(上市公司)、上海合森生物科技有限公司和自然人等股东投资设立的高技术企业,注册资金 3350 万人民币,拥有自主知识产权,主要开展光学、生物学、医学等领域科学仪器的研发、生产、销售及技术服务等工作。 /p p style=" text-align: center" img style=" max-width: 100% max-height: 100% width: 387px height: 272px " src=" https://img1.17img.cn/17img/images/202012/uepic/3d0c783e-ca60-46fb-9bd0-1d6225cdfed8.jpg" title=" hooke instruments 长光辰英-仪器信息网.jpg" alt=" hooke instruments 长光辰英-仪器信息网.jpg" width=" 387" height=" 272" / /p p style=" text-align: justify text-indent: 2em " span style=" text-indent: 2em " 辰英科仪自主研制的单细胞分选仪PRECI SCS具有独特的可视化分选功能,所见即所得,精准实现目标细胞的逐一分离。采用独特的激光与物质相互作用原理,对于复杂生物样本中形态各异的细胞,可实现非标记状态下的精准分离。对于百纳米级的单个微生物细胞也同样适用。 /span /p p style=" text-align: center" img style=" max-width:100% max-height:100% " src=" https://img1.17img.cn/17img/images/202012/uepic/1d280178-fb1f-4040-bbbc-fa069b8e6821.jpg" title=" 006.jpg" alt=" 006.jpg" / /p p style=" text-align: center text-indent: 0em " strong 图5. 单细胞分选仪HOOKE PRECI SCS /strong /p p style=" text-align: center" img style=" max-width: 100% max-height: 100% width: 343px height: 196px " src=" https://img1.17img.cn/17img/images/202012/uepic/571c4847-8a4f-4ce9-a1ca-cf9eeabdab49.jpg" title=" 微信图片编辑_20201216145515.jpg" alt=" 微信图片编辑_20201216145515.jpg" width=" 343" height=" 196" / /p p style=" text-align: center text-indent: 0em " strong 图6. 拉曼单细胞分选仪HOOKE PRECI SCS-R300 /strong /p p style=" text-align: justify text-indent: 2em " PRECI SCS具有可视化、精准、广泛适用等特点。分选过程不依赖标记,可与形态、拉曼、荧光等多种识别方式结合,多种机型可选,满足不同应用需求。搭载潜心研制的HOOKE IntP智能软件,实现单细胞图像智能识别、一键自动分选、全自动细胞获取等。设备操作流程简易,为单细胞测序、未培养微生物开发、工程细胞筛选、细胞图谱绘制等研究提供完美解决方案,助力前沿科学研究。 strong span style=" font-size: 14px color: rgb(165, 165, 165) " (文源:长光辰英) /span /strong /p
 留言咨询
留言咨询
 银牌23年
银牌23年
 400-860-5168转0264
400-860-5168转0264
 留言咨询
留言咨询
 银牌6年
银牌6年
 400-860-5168转4231
400-860-5168转4231
 留言咨询
留言咨询

 400-875-3676
400-875-3676
 留言咨询
留言咨询

 留言咨询
留言咨询
 400-803-5518
400-803-5518
 留言咨询
留言咨询