蛋白质测序技术发展漫谈(上)
导读:本期中国科学院大连化物所单亦初老师将分享蛋白质测序技术的发展与前沿技术,以飨读者。通过蛋白质测序获得的信息有许多用途,包括:蛋白质的鉴定;合成可用作免疫原的肽段;用于治疗的抗体仿制产品的研发等。
本期中国科学院大连化物所单亦初老师将分享蛋白质测序技术的发展,本次分享将以连载形式以飨读者。
蛋白质一级结构是组成蛋白质的氨基酸序列。蛋白氨基酸序列分析是确定蛋白质全部氨基酸序列的过程。通过蛋白质测序获得的信息有许多用途,包括:蛋白质的鉴定;合成可用作免疫原的肽段;用于治疗的抗体仿制产品的研发;以市场上销售的抗体试剂为基础进行抗体药物研发。目前的蛋白质测序方法主要分为三类:基于PCR扩增的蛋白质测序、Edman降解测序以及基于质谱的蛋白质测序。
基于PCR扩增的蛋白质测序是利用细胞中表达的DNA或者RNA进行基因测序,然后再按照氨基酸密码子表转换为蛋白质的氨基酸序列,本质上属于基因测序技术。Edman降解测序是较早发展的蛋白质测序技术,利用化学方法从蛋白质的N端将氨基酸依次降解,再使用高效液相色谱对氨基酸进行鉴定。但是这种方法只能用于鉴定蛋白质和多肽的N-末端氨基酸残基(通常是几个-十几个残基,最多不超过四十个残基),无法对大的蛋白质进行全序列测定。此外,Edman降解法也有一定的局限,例如N末端封闭或有化学修饰的情况下将不能使用Edman降解法对蛋白质序列进行分析。
目前使用最广的蛋白质测序方法是质谱法,较Edman降解法而言,其优点在于,质谱法更敏感,可以更快地裂解肽,可以识别末端封闭或修饰的蛋白质。基于质谱的蛋白质测序策略可分为两大类:自上而下策略(Top-Down)和自下而上(Bottom-Up)策略。自上而下的策略无需对蛋白质进行降解,直接使用LC-MS对完整蛋白质进行分析,根据谱图中碎片离子确定其序列;自下而上策略是先将蛋白质水解成肽段,通过LC-MS对肽段检测,再对肽段从头测序以及序列拼接从而得到完整蛋白质序列。
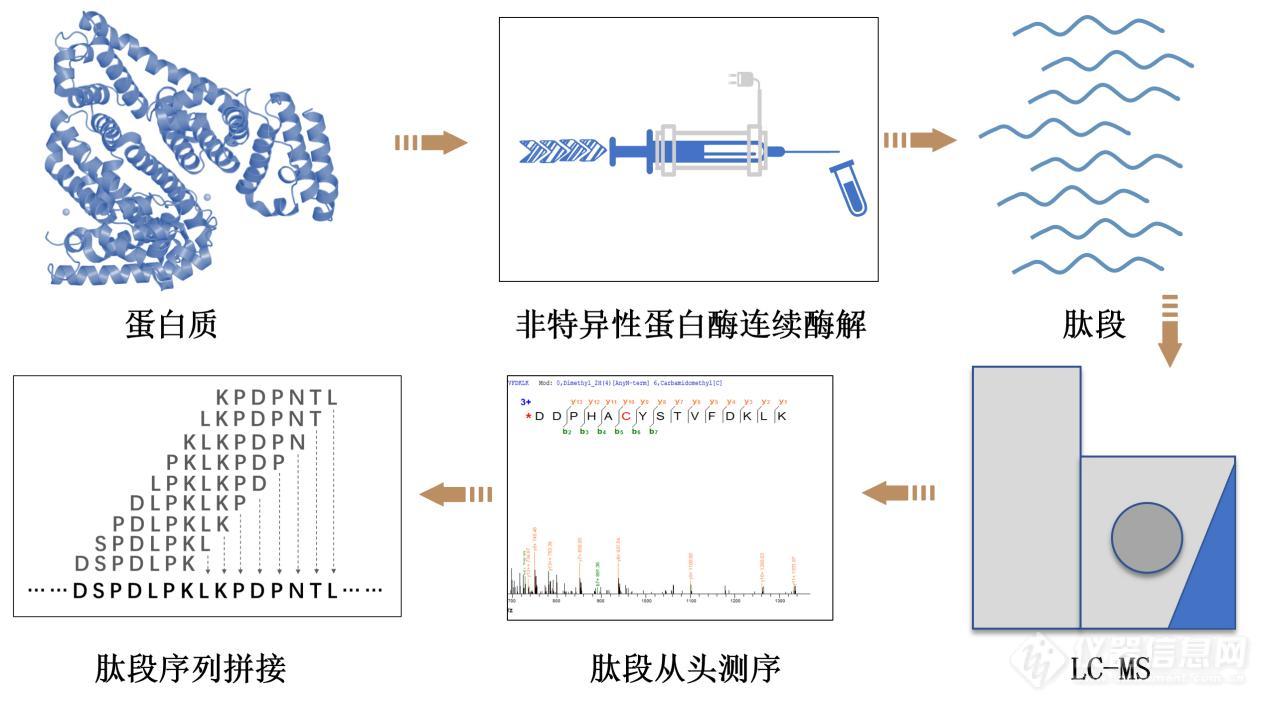
图 :蛋白质序列测定原理
Kira Vyatkina[1]通过自上而下的策略发展了一种Twister测序算法对单克隆抗体测序,虽然不需要使用蛋白酶酶解,减少了蛋白质预处理的步骤,但仅可以鉴定到抗体的序列片段。
Liu[2]结合自上而下和自下而上两种策略发展了TBNovo测序算法对蛋白质进行测序,将自上而下的质谱数据作为抗体序列的骨架,再将胰蛋白酶酶解肽段的质谱数据对骨架的序列进行补充覆盖。由于特异性蛋白酶酶解后肽段种类少、覆盖率低,对抗体的轻链和CAH2区的测序覆盖率为86.9%和75.2%。
Sen[3]发展了一种基于同源数据库搜索与从头测序结合的Supernovo测序算法,通过4种蛋白酶对单克隆抗体分别酶解,该测序方法仅可实现对抗体重链的可变区测序,无法对抗体全序列进行测定。
Savidor[4]发展了一种蛋白质全序列从头测序的方法。将蛋白质在微波辅助下快速酸解,得到了种类丰富的肽段,使用其发展的肽段序列拼接算法——“肽段标签组装”(Peptide Tag Assembler,PTA),对从头测序的肽段进行序列拼接,但由于酸解的消化方式会使谷氨酰胺和天冬酰胺发生脱酰胺化,分别变为谷氨酸和天冬氨酸,降低了对蛋白质序列测定的准确度。
为了解决蛋白质测序覆盖度低、准确度低的问题,我们发展了一种蛋白质全序列测定新方法[5]:该方法使用多种非特异性蛋白酶对蛋白质连续酶解,提高蛋白质酶解肽段种类和重叠度,从而提高蛋白质测序的覆盖度;此外,发展了一种序列拼接算法,根据从头测序得到的肽段序列中每个氨基酸的得分值和出现次数,对蛋白质序列进行组装和拼接,显著提高了蛋白质全序列测定的准确度。利用该测序方法对牛血清白蛋白的多种非特异性蛋白酶酶解后的肽段序列进行测序和拼接,实现了对牛血清白蛋白全序列100%准确度的测定。此外,将该方法应用于对乳腺癌药物单克隆抗体赫赛汀的全序列测定,重链和轻链的测序准确度分别达到99.6%和100%。
参考文献
[1] K V. De Novo Sequencing of Top-Down Tandem Mass Spectra: A Next Step towards Retrieving a Complete Protein Sequence [J]. Proteomes, 2017, 5(1), https://doi.org/10.3390/proteomes5010006
[2] LIU X, DEKKER L J M, WU S, et al. De novo protein sequencing by combining top-down and bottom-up tandem mass spectra [J]. J Proteome Res, 2014, 13(7): 3241-3248.
[3] KI S, WH T, S N, et al. Automated Antibody De Novo Sequencing and Its Utility in Biopharmaceutical Discovery [J]. J Am Soc Mass Spectrom, 2017, 28(5): 803-810.
[4] SAVIDOR A, BARZILAY R, ELINGER D, et al. Database-independent Protein Sequencing (DiPS) Enables Full-length de Novo Protein and Antibody Sequence Determination [J]. Mol Cell Proteomics, 2017, 16(6): 1151-1161.
[5]杨超,单亦初,张玮杰等,基于非特异性蛋白酶连续酶解的蛋白质全序列测定方法,化学学报,修稿中。
作者简介:

中国科学院大连化学物理研究所 单亦初副研究员
1997年于中国科学技术大学获理学学士学位。2002年于中国科学院大连化物所获理学博士学位。2002年10月至2009年5月在德国马普协会马格德堡研究所、美国德克萨斯大学医学院及澳大利亚弗林德斯大学工作。2009年7月应聘到中国科学院大连化物所任副研究员。主持多项研究课题,包括国家重点研发计划子课题、国家自然科学基金面上项目等。已在Analytical Chemistry、Journal of Proteome Research、Journal of Chromatography A等杂志发表论文近80篇。主要研究方向包括蛋白质组鉴定和蛋白质组相对及绝对定量、蛋白质翻译后修饰富集和鉴定、蛋白质组末端肽富集和鉴定、蛋白质相互作用分析、蛋白质全序列从头测定及药物靶蛋白筛选。
(本文经授权发布,仅供读者学习参考)
专家约稿招募:若您有生命科学相关研究、技术、应用、经验等愿意以约稿形式共享,欢迎邮件投稿或沟通(邮箱:liuld@instrument.com.cn)。
来源于:仪器信息网
热门评论
最新资讯
新闻专题













