推荐厂家
暂无
暂无






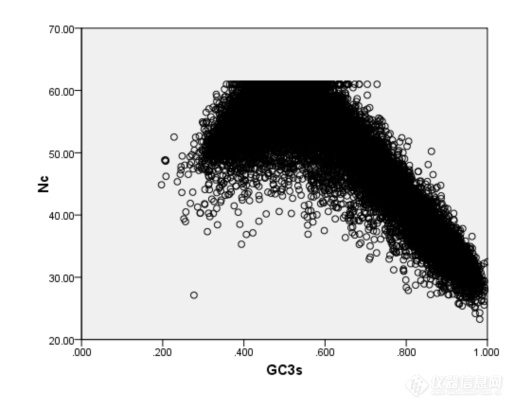
[align=center]短柄草全基因组密码子用法分析分析[/align]摘要:本研究运用CodonW程序分析了短柄草全基因组的密码子使用特性,并且通过对应分析探讨了若干重要因子对短柄草全基因组序列密码子用法的影响。结果表明短柄草基因组存在高[url=https://insevent.instrument.com.cn/t/Mp]gc[/url]含量和低[url=https://insevent.instrument.com.cn/t/Mp]gc[/url]含量的基因,它们在密码子使用上差异较大。Nc-plot曲线表明基因组的密码子组成受到碱基组成的影响;对应分析显示,在DNA水平上发生的核苷酸突变可能是造成短柄草基因组密码子使用偏好的主要因素;同时,基因长度和蛋白质疏水性对密码子的使用也存在一定偏性,但影响程度不大。确定了UUC等27个以G或C碱基结尾的密码子为“最优密码子”,研究结果可为短柄草基因的鉴定、表达、结构、功能等的深入研究提供参考。关键词:同义密码子偏好性,短柄草基因组,对应分析近年来,随着分子生物学的快速发展,许多小基因组的低等生物和高等模式生物的全基因组序列均被测定,为利用生物信息学方法挖掘海量基因组数据提供了便利。密码子是生物体内遗传信息传递的基本环节,是核酸携带信息和蛋白质携带信息间对应的基本规则。在长期进化过程中,任一物种的基因都会逐渐适应宿主的基因组环境,而形成特定的且符合宿主基因组的密码子用法,因此不同生物具有不同的密码子使用模式。以生物基因组数据为基础,研究其密码子使用模式,为深入研究基因的结构、功能和基因组进化,以及指导基因转化等具有重要意义。密码子具有简并性,生物在同义密码子的使用上并不是完全随机的,而是具有一定的偏向性,对有的密码子使用频率高,有的使用频率低,甚至避免使用,这种不均衡使用密码子的现象普遍存在于原核和真核生物中。早在20世纪70年代,人们在研究基因的异源表达时,就已经意识到密码子偏性的重要性[1],随着不同生物基因组数据的获得和各种数据库的构建,更多的研究者对密码子偏性的研究产生了浓厚的兴趣,尤其在分子进化,翻译调控等研究领域,通过对不同物种的密码子使用偏性的大量研究[2~4],发现不同物种的基因在密码子使用上存在着明显的偏性。 短柄草是一种广泛分布于温带地区的禾本科植物,与小麦,大麦和燕麦同属早熟禾亚科,原产于非洲北部,欧洲南部和亚洲中部,包含约10个亚种。该植物为一年生,自花授粉,植株高度15~20cm,生育期70~80d,柄草植株较小,适应性强,不象种植水稻那样需要严格的生长条件。生育期短,籽粒产量较高,一年可以繁殖4~5代,繁殖系数达140左右。未成熟胚和成熟胚愈伤组织诱导率高,农杆菌介导和基因枪介导的转化体系已经建立,胚性愈伤组织分化率90%以上,转化效率最高可达55%左右。基因组小,染色体少,DNA重复序列低,获得突变体容易,突变性状容易显现,具备了模式植物的所有基本特征。加之短柄草基因组序列与黑草麦,小麦,大麦等早熟禾亚科植物高度相似,很多重要农艺性状与温带禾草类植物相似,如株型,穗型,粒型,抗逆性,生长习性和病原菌等,其中麦类作物白粉病菌,条锈病菌和稻类作物瘟病菌都可侵染短柄草植株,引起相应症状[7]。其籽粒不含高分子量麦谷蛋白亚基,低分子量麦谷蛋白亚基也很少,并与小麦一样具有二倍体,四倍体和六倍体,因此短柄草是小麦等基因组庞大的重要农作物理想的模式植物,借此来获得目前小麦等早熟禾类植物中尚缺少的遗传信息和基因共线区,进而对小麦等重要植物进行基因定位,克隆,突变,测序和功能等方面的研究[8]。 目前,在短柄草的生物学、细胞学和遗传学特性方面开展了大量研究,并且其全基因组测序也基本完成[9],为深入研究其密码子用法提供了便利。因此本研究将以短柄草全基因组序列为基础,分析其基因的密码子用法特性和影响密码子使用的因素等,其研究结果将对指导转基因及对基因进行特定分子改造,提高其在短柄草中的表达效率和完善基因预测软件,提高基因预测和基因组注释准确性等均具有重要的参考价值,同时也为深入开展基因结构和功能,分子进化等研究提供理论基础。1.实验材料与方法1.1材料 短柄草全基因组DNA序列来源于短柄草官方数据库(http://www.brachypodium.org/node/8),根据基因组序列的注释信息,获得蛋白编码基因序列,为了减少长度较短的基因变异带来的样本误差,根据国际惯例,去除小于300bp的基因,去除中间不表达的密码子,终止密码子。编写程序提取剩下的蛋白编码基因的CDS(coding sequence)序列。1.2方法用codonw软件计算短柄草全基因组的密码子用法相关参数,主要包括有效密码子数(Effective Number of Codon,ENC)、基因的G+C含量([url=https://insevent.instrument.com.cn/t/Mp]gc[/url]%)、[url=https://insevent.instrument.com.cn/t/Mp]gc[/url]3s%、相对同义密码子使用度(relative synonymous codon usage,RSCU)、氨基酸组分指数(平均亲水性值(gravy))、基因长度即氨基酸数(L_aa)。其中,有效密码子数(Effective Number of Codon,ENC)描述密码子使用偏离随机选择的程度,能反映密码子家族中同义密码子的非均衡性的偏好;其取值范围在20到61之间,即如果每种氨基酸只使用一种密码子则有效密码子数为20,如果各种同义密码子的使用机会完全均等,则有效密码子数为61,数值越小偏性越强。此值是以描述密码子使用偏离随机选择的程度,能反映密码子家族中同义密码子的非均衡性的偏好。基因密码子偏爱程度越大,ENC值越小。RSCU是指对于某种特定的密码子在编码对应氨基酸的同义密码子间的相对频率;[url=https://insevent.instrument.com.cn/t/Mp]gc[/url]3s%表示同义密码子第三位碱基的G+C的含量。为进一步了解该家族基因密码子使用特征和影响密码子使用的因素,对7个基因的相对同义密码子使用度进行了对应性分析(correspondence of analysis,COA)。2 结果与分析2.1 基因的碱基组成对密码子使用的影响图一 短柄草基因NC值散点图[img=,515,409]https://ng1.17img.cn/bbsfiles/images/2019/10/201910311236371230_3093_3295053_3.png!w515x409.jpg[/img]2.2短柄草基因密码子使用特性的对应性分析[img=,690,535]https://ng1.17img.cn/bbsfiles/images/2019/10/201910311237226440_1452_3295053_3.png!w690x535.jpg[/img][img=,690,534]https://ng1.17img.cn/bbsfiles/images/2019/10/201910311237233450_935_3295053_3.png!w690x534.jpg[/img]2.3 确定最优密码子Phe UUU 0.05 (323) 1.23 (19733) Ser UCU 0.22 (990) 1.60 (23834) UUC* 1.95 (13527) 0.77 (12294) UCC* 2.55 (11715) 0.64 (9499) Leu UUA 0.02 ( 93) 0.83 (11755) UCA 0.14 (629) 1.52 (22651) UUG 0.16 (1003) 1.37 (19558) UCG* 1.53 (7023) 0.35 (5159) CUU 0.14 (847) 1.55 (21987) Pro CCU 0.22 (1306) 1.57 (17584) CUC* 3.38 (20676) 0.61 (8661) CCC* 1.35 (7940) 0.47 (5299) CUA 0.07 (452) 0.70 (9983) CCA 0.20 (1184) 1.62 (18078) CUG* 2.23 (13637) 0.94 (13401) CCG* 2.22 (13058) 0.34 (3792) Ile AUU 0.12 (398) 1.41 (21216) Thr ACU 0.10 (401) 1.46 (16515) AUC* 2.76 (9124) 0.70 (10557) ACC* 1.75 (7291) 0.66 (7397) AUA 0.12 (380) 0.89 (13461) ACA 0.12 (509) 1.56 (17636) Met AUG 1.00 (8512) 1.00 (20892) ACG* 2.03 (8478) 0.32 (3563) Val GUU 0.10 (693) 1.67 (23852) Ala [url=https://insevent.instrument.com.cn/t/Mp]gc[/url]U 0.14 (1914) 1.65 (26184) GUC* 1.71 (12491) 0.63 (9025) [url=https://insevent.instrument.com.cn/t/Mp]gc[/url]C* 1.98 (27398) 0.58 (9131) GUA 0.05 (349) 0.75 (10713) [url=https://insevent.instrument.com.cn/t/Mp]gc[/url]A 0.13 (1802) 1.48 (23459) GUG* 2.14 (15605) 0.95 (13562) [url=https://insevent.instrument.com.cn/t/Mp]gc[/url]G* 1.75 (24170) 0.29 (4678) Tyr UAU 0.05 (229) 1.28 (14480) Cys UGU 0.06 (194) 1.10 (9360) UAC* 1.95 (8126) 0.72 (8075) U[url=https://insevent.instrument.com.cn/t/Mp]gc[/url]* 1.94 (6645) 0.90 (7595) TER UAA 0.42 (172) 0.82 (335) TER UGA 1.63 (665) 1.30 (530) UAG 0.94 (384) 0.87 (356) Trp UGG 1.00 (4992) 1.00 (10053) His CAU 0.15 (598) 1.42 (16785) Arg CGU 0.16 (750) 0.85 (6945) CAC* 1.85 (7568) 0.58 (6825) C[url=https://insevent.instrument.com.cn/t/Mp]gc[/url]* 2.75 (12565) 0.49 (4043) Gln CAA 0.15 (627) 1.05 (20215) CGA 0.11 (500) 0.64 (5273) CAG* 1.85 (7975) 0.95 (18259) CGG* 1.92 (8761) 0.55 (4527) Asn AAU 0.12 (465) 1.31 (26650) Ser AGU 0.05 (235) 1.13 (16754) AAC* 1.88 (7141) 0.69 (13985) A[url=https://insevent.instrument.com.cn/t/Mp]gc[/url]* 1.52 (7002) 0.77 (11441) Lys AAA 0.11 (552) 0.98 (27077) Arg AGA 0.10 (445) 1.94 (15854) AAG* 1.89 (9406) 1.02 (28423) AGG 0.96 (4387) 1.53 (12516) Asp GAU 0.15 (1344) 1.44 (39136) Gly GGU 0.11 (882) 1.34 (18423) GAC* 1.85 (16539) 0.56 (15322) G[url=https://insevent.instrument.com.cn/t/Mp]gc[/url]* 2.53 (20795) 0.71 (9826) Glu GAA 0.17 (1437) 1.13 (36292) GGA 0.19 (1522) 1.26 (17423) GAG* 1.83 (15812) 0.87 (27746) GGG* 1.18 (9700) 0.69 (9476) 注:Number of codons in high bias dataset 372333 Number of codons in low bias dataset 915109标注*的密码子是(p 0.01)3 讨论密码子使用偏好是突变偏好、自然选择和遗传漂变等共同作用的结果,与碱基组成、翻译选择压力、基因表达水平、基因长度、蛋白质氨基酸组成、碱基突变频率和模式、mRNA二级结构稳定性等很多因素有关[17]。张晓峰[18]等研究表明,单子叶植物基因组的[url=https://insevent.instrument.com.cn/t/Mp]gc[/url]含量在同义密码子使用偏性的产生过程中起着决定性的作用,同义密码子使用偏性强烈的基因往往偏爱使用C或G结尾的密码子,且第三位密码子突变往往是密码子偏好性发生变化的决定原因。短柄草基因密码子使用模式的调查表明其中有高含量的[url=https://insevent.instrument.com.cn/t/Mp]gc[/url],并且[url=https://insevent.instrument.com.cn/t/Mp]gc[/url]3的含量高于[url=https://insevent.instrument.com.cn/t/Mp]gc[/url]1和[url=https://insevent.instrument.com.cn/t/Mp]gc[/url]2。这表明相对于以A和T结尾的密码子而言,这些基因偏好于使用以G或C结尾的密码子。从原核生物到真核生物的基因中,密码子使用偏好是一个被广泛研究的重要进化现象。研究发现,许多因素,比如碱基组成,基因表达水平,蛋白质疏水性等影响着密码子的使用。为了解释密码子使用偏好的起因,也有许多假设被提了出来。其中被广为接受理论是“选择——突变——漂移”模型。该模型认为在对偏好密码子的选择和通过突变-漂移对非偏好密码子的保留之间,同义密码子的使用偏性存在一种平衡。本文的研究结果显示,[url=https://insevent.instrument.com.cn/t/Mp]gc[/url]3s值与ENC值密切相关,并且基因也位于第一轴线,揭示了碱基组成是影响短柄草基因组中的密码子使用偏好的主要因素。碱基组成是影响短柄草基因密码子使用的主要因素,基因长度和蛋白质的疏水性在短柄草基因密码子使用中也起到了一定的作用,相似的结果在水稻、小麦中被发现[15,19]。本研究发现,在基因长度和[url=https://insevent.instrument.com.cn/t/Mp]gc[/url]之间存在很强的负相关性。这表明,高[url=https://insevent.instrument.com.cn/t/Mp]gc[/url]含量的基因越短,密码子偏好就越大。可能的原因是富含AT基因的翻译效率比富含[url=https://insevent.instrument.com.cn/t/Mp]gc[/url]基因的翻译效率更高,这种效率的差异对长的基因更为重要。通常,全基因组的基因表达值在许多多细胞真核生物中并不能得到,特别是基因表达水平在不同的组织和不同发育阶段不一样时。因此,要定量相当困难。在短柄草基因组中,目前还缺少相当数量的基因表达的准确数据。另外,我们发现[url=https://insevent.instrument.com.cn/t/Mp]gc[/url]含量特别是在第三个碱基位置的[url=https://insevent.instrument.com.cn/t/Mp]gc[/url]含量较大的影响着密码子的偏好时,暗示着碱基突变可能是重要因素,同时,碱基突变又受控于翻译选择。所以,尽管基因表达水平影响着密码子的使用,但这影响还是远远小于核苷酸组成对密码子使用的影响。因此,我们没有进一步分析基因表达的影响。通过优化密码子,提高外源基因在微生物、植物、动物中的表达已有不少成功报道,而确定最优密码子可为合理有效进行密码子改造提供可靠信息。本文确定了UUC等27个密码子为短柄草全基因组的最优密码子。分析结果可为指导转基因及对基因进行特定分子改造,提高其在短柄草中的表达效率和完善基因预测软件,提高基因预测和基因组注释准确性等提供重要的参考价值。参考文献[1] Stanley D,Farnden K J F, MacRae E A. Plant a-amylases:Func-tions and roles in carbohydrate metabolism[J]. Biologia,Bratislava,2005.60(suppl l6):65-71[2] Smith AM. Zeeman SC, Smith S M. Starch degradation[J]. Annu Rev Plant Biol,2005,56(25):73-98[3] Asatsuma S, Sawada C, Itoh K et al. Involvement of α-amylase I-1 in starch degradation in rice chloroplasts[J]. Plant Cell Physiol,2005,4:858-869[4] Kaplan F, Guy C L. β-amylase induction and the protective role of maltose during temperature shock[J]. Plant Physiol, 2004, 1:1674-1684 [5] Kaplan F,Guy C L. RNA interference of Arabidopsis beta-amylase 8 prevents maitose accumulation upon cold shock and increases sensitivity of PSII photochem-ical efficiency to freezing stress[J]. Plant J.2005,44(13):730-743[6] Joho Mundy, Anders Brandt. Messenger RNAs from the Scutellum and Aleurone of Germinating Barley Encode (lm3,14)--D-Glucanase, a-Amylase and Carboxypeptidase[J]. Plant Physiol, 1985,79(5):867-871 [7] 言普,李桂双.高压对水稻种子细胞膜透性和淀粉酶活性的影响[J]. 浙江大学学报(农业与生命科学版),2007,33(5):174-179[8] Monica M, Sanwo and Darleen A. DeMason. Characteristics of a-Amylase during Germination of Two High-Sugar Sweet Corn Cultivars of Zea mays L[J]. Plant Physiol, 1992,99(8):1184-1192[9] Goldman N , Yang Z. A codon based model of nucleotide substitution for protein coding DNA sequences[J]. Molecular Biology and Evolution,1994,11(9):725-736[10] Schmidt W. Phylogeny reconstruction for protein sequences based on amino acid properties[J]. Mol Evol,1995,41(8) :522-530[11] 时成波, 吕安国.改造稀有密码子提高SEA蛋白表达量[J]. 生物工程学报,2002,18(4):477-480[12] Ghosh T C , Gupta S K, Majumdar S. Studies on codon usage in Entamoeba histolytica[J]. Int J Parasitol,2000,30(6): 715-722[13] Musto H, Cruveiller S. Translational selection on codon usage in Xenopus laevis[J].Molecular Biology and Evolution,2001,18(9):1703-1707[14] 廖登群,张洪亮等. 水稻(Oryza sativa L.)a-淀粉酶基因的进化及组织表达模式[J]. 中国农业大学学报,2009,14(5):1-11[15]刘汉梅,何瑞. 玉米密码子用法分析[J]. 核农学报,2008,22(2):141-147[16] Jia M, Luo L. The relation between Mrna folding and protein structure[J]. Biophys Res Commum, 2006,343(4):177-182[17] 赵耀,刘汉梅. 玉米waxy基因密码子偏好性分析[J]. 玉米科学,2008,16(2):16-21 [18] Wang H C,Hickey D A. Rapid divergence of codon usage patterns within the rice genome[J].BMC Evol Biol,2007,15(8):347-356
霍华休斯医学研究所,Baylor医学研究所的科学家们近期在PloS One上发表最新研究性文章,文章标题为:Big Genomes Facilitate the Comparative Identification of Regulatory Elements,该文章解析了基因组大小对基因组学的研究带来的影响。基因组越大则更容易找出控制基因活性的DNA区域。在小基因组上,功能性元件紧紧地结合在一起。而在大基因组上,功能性元件分得比较散,于是也更容易找到控制基因活性的区域。 基因组分为结构基因和调控基因,要从基因组上找到功能元件并不难,难的是找到调控基因表达的机制,因此,对小的基因组来说,紧凑的结构给寻找调控区域带领更多的困难,而相对来说大基因组却容易多了。功能元件散落在基因组上,更便于寻找调控区域。大的基因组更便于研究非编码DNA和RNA,对研究基因调控也更为有利。而目前,研究生命的遗传物质DNA的科学家一直觉得,基因组越小越受欢迎,因为操作简单,可以节省大量的时间和精力,尤其在金钱方面也能更节约成本,测序的费用更低。甚至有科学家说,基因组小则基因排列更紧凑,垃圾DNA也越少。 [img]http://www.instrument.com.cn/bbs/images/affix.gif[/img][url=http://www.instrument.com.cn/bbs/download.asp?ID=137848]Big Genomes Facilitate the Comparative Identification of Regulatory Elements[/url]
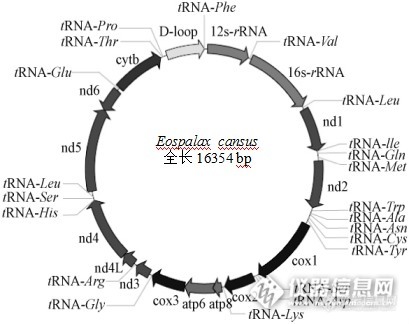
[b][/b][align=center]鼢鼠([i]Eospalax[/i])线粒体基因组测定及注释分析[/align][align=center]西安国联质量检测技术股份有限公司[/align][align=center]安平中心:李瑞[/align][b]摘要【[/b]目的】获得鼢鼠线粒体基因组全序列,为线粒体基因组功能标记及进化生物学等研究提供基础资料。【方法】参考鼹型鼠等动物的线粒体基因组序列,设计出可覆盖鼢鼠线粒体基因组的16对引物,采用[url=https://insevent.instrument.com.cn/t/jp][color=#3333ff]PCR[/color][/url]产物直接测序法测得甘肃鼢鼠线粒体基因组全序列,分析其基因组的特点和基因结构。并结合GenBank中发表的啮齿类动物基因组全序列,探讨啮齿类动物的系统进化关系。【结果】鼢鼠线粒体基因组全长16354bp,其中包括22个tRNA基因、13个蛋白质编码基因、2个rRNA基因和2个D-loop区。碱基组成为33.5%A、24.2 %C、12.3 %G、30.0 %T。【结论】鼢鼠线粒体基因组结构及其信息和其他啮齿类动物的结构一致,线粒体变异保守。研究结果为鼢鼠的低氧适应、系统发育关系等提供了基础资料。[b]关键词 [/b]鼢鼠;线粒体基因组;序列分析 鼢鼠([i]Eospalax[/i])是分布于我国的主要啮齿类动物之一,其体型较小,栖息于洞穴内有挖掘活动,扩散能力强,数量波动大,是生态系统中重要的初级消费者,处于生态系统中的中心位置,草原生态系统中其能流比重很大[sup][/sup]。动物线粒体([color=#333333]Mitochondrion[/color])基因组为双链闭合环状分子[sup][/sup],少数也有线性的,它们具有分子量相对较小、结构简单、缺少重组、母性遗传和进化速率快等特点,已成为动物系统发育与进化、群体遗传学、分子生态学以及疾病机理研究等领域的理想材料[sup][/sup]。甘肃鼢鼠是仅分布于我国西北部的土著物种,其外形似中华鼢鼠,主要分布于甘肃临潭县及其附近地区。目前对线粒体DNA的研究主要在动物分子遗传学、分子生态学、种群遗传结构分析、遗传多样性、物种和品系鉴定、保护遗传学等方面得到了广泛应用[sup][4[/sup][sup],[/sup][sup]5][/sup]1. [b]实验材料和方法[/b]1.1 实验材料鼢鼠:采集于天祝(经度102.84、纬度 37.2)1个群体;鼢鼠解剖采集肝脏及肌肉组织样品,-20℃保存备用。1.2 线粒体DNA的提取用剪刀将肝脏及肌肉材料剪成小块,取0.1cm左右的小块肝脏及肌肉材料,采用常规的SDS/蛋白酶K裂解,酚氯仿提取DNA[sup][/sup],使用琼脂糖凝胶电泳检测其完整性。1.3 引物设计和[url=https://insevent.instrument.com.cn/t/jp][color=#3333ff]PCR[/color][/url]扩增通过Clustal X1.83比对,寻找相对应保守区域位置,用Primer Premier5.0引物设计软件设计引物,并对每条引物进行评价和修改,最终确定16对引物。以所提取的DNA为模板,用16对引物扩增覆盖整个线粒体基因组。利用引物进行[url=https://insevent.instrument.com.cn/t/jp][color=#3333ff]PCR[/color][/url]扩增,反应体系总体积为50μL,其中含有6μL [url=https://insevent.instrument.com.cn/t/jp][color=#3333ff]PCR[/color][/url] buffer、3μL MgCl[sub]2[/sub](1.5mmol)、MgCl[sub]2[/sub],2μL dNTPs (100μL mol)、上下游引物各2μL (0.25μL mol)、Taq DNA聚合酶2μL (1U)、总DNA约为2μL (25ng)、去离子水31μL。反应程序为:94℃预变性4 min,94℃变性50s,48-45℃退1min,72℃延伸1 min 30s,循环30次,之后72℃延伸10min,并根据不同引物的退火温度和扩增反应的实际效果进行优化。取 5 μL [url=https://insevent.instrument.com.cn/t/jp][color=#3333ff]PCR[/color][/url]扩增产物,和2 μL DNA marker 2000,进行1.0%琼脂糖凝胶(1×TBE)5V/电泳,用紫外观察[url=https://insevent.instrument.com.cn/t/jp][color=#3333ff]PCR[/color][/url]产物扩增情况,凝胶成像仪扫描记录结果。1.4 纯化、测序和序列拼接 在[url=https://insevent.instrument.com.cn/t/jp][color=#3333ff]PCR[/color][/url]产物中加入5 U SAP和2 U ExoⅠ,震荡混匀,37℃保温1 h,然后75℃保温15 min以灭活SAP和ExoⅠ酶,纯化好的模板可以在4℃保存24 h或-20℃长期保存。将纯化后的引物送往上海生工生物技术服务有限公司用ABI-3730序列自动分析仪进行双向测序。利用DNASTAR和测序峰图结果分析软件Chromas 2.22校对测序图,DNAMAN拼接序列。得到甘肃鼢鼠线粒体全基因组全序列。2. [b]结果[/b]2.1 鼢鼠线粒体基因组基因定位2.2.1 鼢鼠线粒体2个rRNA的分析哺乳动物线粒体的rRNA具有高度的保守性,它们的位置固定,12S rRNA位于tRNA-phe 和tRNA-Val之间,16S rRNA位于tRNA-Val和 tRNA-Leu之间,12S rRNA起始位置为68,终止位置为1019,长度为952bp,16S rRNA起始位置为1086,终止位置为2651,长度为1566。同时我们比对了鼢鼠和中华鼢鼠的rRNA基因和蛋白质基因,12S rRNA和16S rRNA的相似性分别为91.0%和87.3%,高于蛋白质编码基因之间的相似性。2.2.3 鼢鼠线粒体基因组结构 除NADH脱氢酶亚基6外均在H链上,虽然鼢鼠染色体数目少、染色体大,但与其它哺乳动物线粒体全基因组相比,它的线粒体基因组的结构与其它哺乳动物是十分相似的。甘肃鼢鼠线粒体基因组结构见图1。[align=center][img=,409,324]http://ng1.17img.cn/bbsfiles/images/2017/09/201709081454_02_2904018_3.png[/img][/align]注:ND: NADH脱氢酶亚基(NADH dehydrogenase subunit)、Cox:细胞色素氧化酶亚基(cytochrome oxidase subunit)、Atp:ATP合成酶亚基(ATP synthase F0 subunit)、Cyt b:1个细胞色素b编码基因(cytochrome b)。[align=center][b]图1[/b] 甘肃鼢鼠线粒体基因组结构简图[/align][align=center]Fig.1 The gene organization of [i]Eospalax cansus[/i] mitochondrial genome[/align]3. [b] 讨论[/b] 甘肃鼢鼠线粒体基因组的D-loop区,长度为933bp,比中国地鼠D-loop区(867bp)长。D-loop区对目的基因是不可缺少的,虽然D-loop区不能编码蛋白质但对于遗传信息表达是不可缺少的,在它上面有调控遗传信息表达的核苷酸序列,具有遗传效应的,比如RNA聚合酶结合位点是具有遗传效应的。8只甘肃鼢鼠中有5个单倍型:3只临潭群体共享1个单倍型,2只天祝群体独享单倍型;其余个体均独享单倍型,表明了甘肃鼢鼠线粒体DNA D-loop区碱基变异快、进化快的特性,符合啮齿动物线粒体变异大的现象。随着研究的深入,以线粒体DNA中完整的基因序列或多个基因序列协同而获得遗传信息来探讨物种的系统进化关系,将是以后研究发展的主要方向[sup][/sup]。目前,线粒体DNA已经在许多哺乳类动物的起源进化的研究中取的了重大进展,而对甘肃鼢鼠的起源进化的研究却很少,并且存在着甘肃鼢鼠属于[url=http://baike.baidu.com/view/113192.htm][color=#000000]瞎鼠科[/color][/url]和仓鼠二者之争,因此,为了更好的阐明甘肃鼢鼠的起源,还需要做更多、更深入的研究。

 400-860-5168转4819
400-860-5168转4819
 留言咨询
留言咨询

 400-807-5250
400-807-5250
 留言咨询
留言咨询
 400-801-8117
400-801-8117
 留言咨询
留言咨询