中文摘要: 目的 实验室规模通过近红外光谱分析技术对人血白蛋白醇沉过程进行监测,为提高组分I+II+III上清液乙醇沉淀环节收率提供支持。 方法 实验室条件下模拟8批正常过程和3批异常过程。定性模型以6个正常批次作为校正集建立主成分分析模型,2个正常批次和3个异常批次作为验证集考察模型过程监测和错误诊断能力。定量模型以醇沉过程中人血白蛋白含量和总蛋白含量作为建模指标,6个正常批次样品作为校正集,2个正常批次样品作为验证集,建立了可用于人血白蛋白和总蛋白含量快速准确测定的偏最小二乘回归模型。 结果 定性的主成分分析模型和定量偏最小二乘回归模型可实现醇沉过程的监测和错误判断。 结论 利用近红外光谱分析技术结合化学计量学对人血白蛋白生产过程中的FI+II+III上清液醇沉过程进行监测的方法可行。
关键词:近红外光谱分析技术;组分I+II+III;化学计量学;醇沉淀
人血白蛋白(Human Albumin,HA)是最早从人血浆中提取并应用于临床上的血液制品。组分(Fraction, F)I+II+III上清液醇沉过程是HA生产过程中的重要一步,其目的是去除各种杂蛋白,得到FIV上清液,为下一步醋酸缓冲液沉淀过程做准备。目前人血白蛋白组分I+II+III上清液醇沉过程生产过程的控制模式大都采用离线的方式,即当乙醇加入到含量为40%时醇沉过程结束,然后取样进行实验室化验,其结果严重滞后于生产过程,无法实时的监测指导生产过程的进行。在制药领域,NIRS作为一种重要的PAT工具,已成功用于药物的原辅料质量评价、关键过程的监测和控制、成品的快速放行和质量检测等各个环节,为保证产品质量、降低生产成本、革新生产过程发挥了重要的作用。本研究中利用NIRS结合化学计量学对HA生产过程中的FI+II+III上清液醇沉过程进行监测,以实现醇沉环节的过程控制。
1 材料
1.1 试剂
FI+II+III压滤后上清液(山东泰邦生物制品有限公司);95%乙醇(分析纯,国药集团化学试剂有限公司);去离子水。
1.2 仪器和软件
Antaris II傅里叶变换近红外(Fourier Transform NearInfrared, FT-NIR)光谱仪(美国Thermo Fisher Scientific公司),液体透射采样模块,内径为4×50 mm的玻璃小管(德国Kimble Chase公司);低温反应仪(郑州长城科工贸有限公司);BF300恒流泵(保定齐力恒流泵有限公司);Alpha 1-2 LD实验室型冻干机(德国Christ公司);高速离心机(Thermo Fisher Scientific公司);MATLAB 2010a(美国Mathworks公司);PLS_Toolbox工具箱(美国Eigenvector Research公司)。
2 方法
2.1 醇沉过程
正常醇沉过程:醇沉过程在低温反应仪中进行,温度设定和实际生产反应温度一致。每个批次取100 mL FI+II+III压滤后上清液置于250 mL的圆底烧瓶中,恒流泵加入95%乙醇的速度为1 mL/min。醇沉开始前以及醇沉过程中每隔3 min取样1 mL用于光谱的采集及蛋白含量的测定。异常醇沉过程:本研究中为考察定性模型对异常醇沉过程的诊断能力,每个批次样品量均为100 mL。8个正常的醇沉过程批次和3个异常的醇沉过程批次信息如表1中所示。
醇沉结束后将醇沉过程中取得的样品在12000 r/min条件下离心15 min,以去除沉淀颗粒对光谱采集及一级数据采集的影响。
表1 不同批次醇沉过程信息
批次 |
过程条件 |
醇沉过程时间 (min) |
正常批次1-8 |
正常的醇沉条件 | 45 |
异常批次 1 |
10 min时停止乙醇的加入
25 min时重新开始加入乙醇 |
异常批次 2 |
15 min将乙醇的加入速度提高为2 mL/min
25 min时乙醇的加入速度恢复至1 mL/min |
异常批次 3 |
10 min时停止搅拌
27 min时重新打开搅拌 |
2.2 样品光谱的采集
将样品装载于内径为4 mm的玻璃小管中,采用Antaris II FT-NIR光谱仪的透射模块进行原始近红外光谱的采集。光谱的扫描范围为10000-4000 cm-1,分辨率为8 cm-1,扫描次数为32次,以空气作为参比,每小时采集一次背景。
2.3 白蛋白和总蛋白含量的测定
HA和总蛋白含量测定方法分别为溴甲酚绿(Bromocresol Green, BCG)比色法[8]和双缩脲法[9],所有样品均委托金域医学检验中心进行相关含量的测定,使用的仪器为贝克曼AU 5800型全自动生化分析仪。
由于HA和总蛋白的含量测定均需要在特定的pH条件下进行,pH的变化对含量测定有较大的影响。不同样品中乙醇的含量有较大的差别,对测定过程中的pH可能会有影响,本研究中对采集得到的样品进行冻干24 h以上然后用去离子水复溶的处理,以去除不同乙醇含量对结果的影响。
2.4 光谱数据的处理和模型的建立
本研究中通过MATLAB 2010a 数学软件以及基于该软件的PLS_Toolbox工具箱对光谱数据进行处理,建立定性分析和定量测定的模型。首先,对光谱数据进行预处理,以消除无关信息的干扰。然后,建立定性监测醇沉生产过程及异常批次判断的PCA模型,以主成分得分对生产过程进行定性监测。最后,建立测定HA含量和总蛋白含量的PLSR定量分析模型,以实现醇沉过程中HA和总蛋白含量的实时、定量测定,有效的对醇沉过程进行监测。
3 实验结果
3.1 样品的原始光谱
图1为11个批次176个样品的原始近红外光谱图,由图中显示样品光谱间相似性很高,波段6600-5300 cm-1间存在较大的差别。同时由于样品中主要是以水和乙醇组成的溶液环境,样品光谱中有很强的O-H吸收峰,可见在O-H的一级倍频光谱吸收区(7050-6650 cm-1)和组合频光谱吸收区(5200-4500 cm-1)出现了吸收饱和的现象。
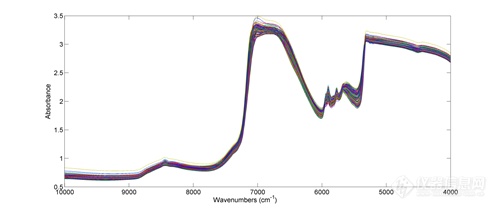
图1 醇沉过程样品原始光谱图
3.2 白蛋白和总蛋白含量测定结果
正常批次醇沉过程HA的含量变化范围为9.5-16.9 g/L,总蛋白含量变化范围为10.3-19.4 g/L。同时3个异常批次的蛋白含量变化和正常批次有较为明显的不同,这为PCA定性分析模型的建立提供了较好的依据。
图2显示了8批正常醇沉过程中杂蛋白的比例(除掉HA含量后的蛋白含量占总蛋白含量的比例)变化图。由图中显示随着醇沉过程的不断进行,杂蛋白的比例总体呈下降趋势,表明醇沉过程中杂蛋白被不断的去除。图中红色的直线代表杂蛋白的比例为0.1时的阈值线,由图中可知不同批次中杂蛋白的比例小于0.1时的乙醇含量不同,变化范围为36.0-43.2%(40%左右),因此过程监测HA和总蛋白的含量对最佳醇沉终点的判断有重要的借鉴作用,对保证最终产品的质量和提高产品的收率有重要的意义。
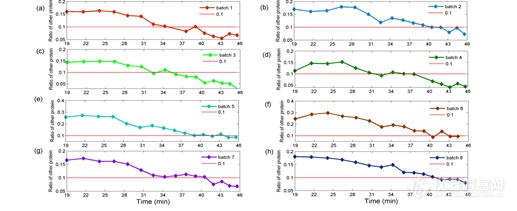
图2 醇沉过程中正常批次杂蛋白比例变化
3.3 光谱的预处理
在本研究中,为去除噪声等无关信息对光谱的影响,有效提取表征特征物质的光谱信息,对原始光谱进行的预处理为一阶导数SG 15点平滑和mean center。
3.4 PCA模型的建立
将预处理后的光谱进行PCA,建立用于醇沉过程监测的PCA定性分析模型。本研究中以正常醇沉过程的批次1至6作为建立模型的校正集,以正常批次7至8和3个异常批次作为验证集。由图1可知光谱区间7093-6576 cm-1和5334-4000 cm-1吸收达到饱和,作为异常光谱区间去除,然后建立定性分析模型。
3.4.1 主成分贡献率
光谱进行PCA后原来的光谱变量点线性组合为少数几个新变量,这些新变量称之为主成分。图3为得到的主成分贡献图。
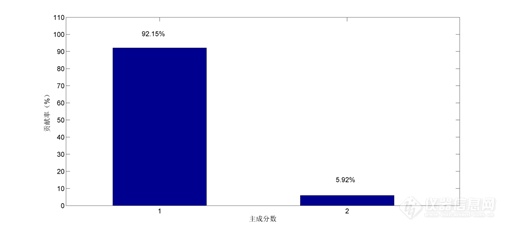
图 3 主成分贡献率
3.4.2 主成分得分控制图
由图3可知主成分1(PrincipalComponent 1, PC1)解释了光谱92.15%的变异,代表了大部分的光谱信息,并且得分代表了醇沉过程中体系的变化趋势,因此建立了基于PC1得分的主成分得分控制图。本研究中首先计算6个正常批次醇沉过程PC1得分的平均值,然后计算平均值±3倍的标准偏差作为控制图的上下控制线。图4为醇沉过程中校正集PC1得分图,图中红色线表示由平均值±3倍标准偏差得到的上下控制线。由图中显示,随着醇沉过程的进行,PC1得分不断下降,并且6批校正集样品醇沉过程的PC1得分的变化趋势基本一致,说明醇沉过程批次间的差异很小。
图5为验证集2个正常批次和3个异常批次醇沉过程PC1得分图,图中红色线为±3倍标准偏差的控制线。图5 (a) 为正常批次7和8醇沉过程PC1得分图,由图中显示批次7和批次8醇沉过程PC1得分完全落在±3倍的标准偏差的控制线内,证明了模型对正常醇沉过程监测的有效性。图5 (b) 为异常批次1醇沉过程PC1得分图,异常批次1为模拟恒流泵加醇出现故障的情况,由图中显示前4个取样点的得分变化逐渐下降,从第4个取样点处得分开始出现拐点,得分下降趋势不明显,当醇沉过程进行到大约15 min左右时,异常批次1的得分超出±3倍的标准偏差的控制线,证实了模型对恒流泵出现异常的诊断能力。图5 (c) 为异常批次2的过程得分图,异常批次2模拟了加醇速度出现异常的情况,由图中显示从第6个点开始,PC1得分逐渐接近-3倍的标准偏差的控制线,在30 min时,PC1的得分超出±3倍的标准偏差的控制线,表明PC1得分能够一定程度的判断加醇出现异常的情况。图5(d)为异常批次3的过程控制图,该批次在10-27 min停止搅拌以模拟搅拌出现故障的情况,由图中显示在12 min时PC1得分出现了明显的拐点,在15 min时PC1得分开始脱离±3倍的标准偏差的控制线,第27 min复开搅拌时PC1得分开始出现急剧的下降过程,第30 min时PC1得分重新回到±3倍的标准偏差的控制线内,直至醇沉终点到达,说明如果在生产过程中及时发现搅拌失败的故障并能迅速的处理,醇沉过程可恢复到正常状态中。由此可知PC1得分能够有效的判断搅拌出现异常的情况,可以实现醇沉过程的故障监测和诊断。
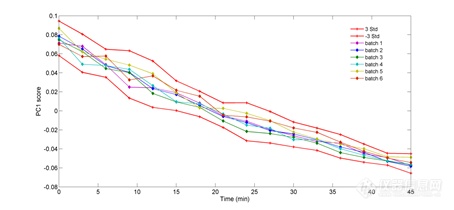
图4 醇沉过程校正集PC1得分控制图

图5 醇沉过程验证集PC1得分控制图
(a) 正常批次7和8 (b) 异常批次1 (c) 异常批次2 (d) 异常批次3
3.5 PLSR模型的建立
本研究中以PCA模型建立中的6个正常批次作为校正集,2个正常批次作为验证集,建立用于醇沉过程HA和总蛋白含量测定的PLSR定量分析模型。表2为样品集划分信息表。在模型的建立中选择Venetian Blinds交互验证方法选择最佳的潜在变量数(Latent Variables, LVs),同时以RMSEP值和RPD值作为模型的评价标准,以确定最佳的建模光谱区间。其中RMSEP值越小,RPD值越大说明模型的预测能力越高,模型结果越好。
表2 样品集划分信息表
| 指标 | 样品集 | 批次数 | 最大值(g/L) | 最小值(g/L) | 平均值(g/L) | SD(g/L) |
| HA | 校正集 | 6 | 16.9 | 9.5 | 13.3 | 1.87 |
| 验证集 | 2 | 15.7 | 10.0 | 12.9 | 1.78 |
| 总蛋白 | 校正集 | 6 | 19.4 | 10.3 | 14.9 | 2.59 |
| 验证集 | 2 | 19.1 | 10.7 | 14.8 | 2.61 |
3.5.1 Loading值分析
图6为LV1和LV2的载荷图,由图中显示光谱的变异主要集中在7100-6950 cm-1和6121-5361 cm-1这两个区间。其中区间7100-6950 cm-1主要是O-H基团(存在于水和乙醇中)的一级倍频吸收,光谱的变异主要是由于水和乙醇含量的变化引起的。此外,从原始光谱图可知该波段区域达到了吸收饱和,因此不将该波段选择用于建模。区间6121-5361 cm-1主要代表C-H和S-H基团(主要存在于蛋白质分子中)的一级倍频吸收,其光谱的变异可能是由于样品体系中蛋白质的含量发生变化引起的,因此该波段区域可能对最终模型的建立有重要的作用。
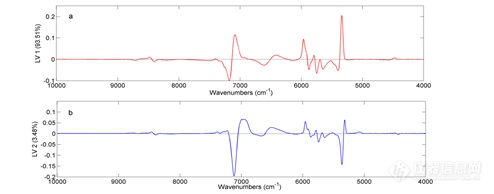
图6载荷图(a: LV1 b: LV2)
3.5.2 手动波段选择及模型的建立
将8个正常醇沉过程中同一时间点的样品光谱进行平均,图7为得到的平均后的16张不同醇沉时间点的样品光谱。由光谱图可知吸光度值在6121-5361 cm-1这一波段区间存在明显的差异。对其进行局部放大,图中红色的箭头代表随着醇沉过程进行吸光度的变化情况。可知在四个明显的波峰和波谷区域,吸光度值随着醇沉过程的进行存在着有规律的变化。本研究中对该区域的5个波段进行了考察,并与全光谱建模的结果进行比较。表3为不同的波段建模得到的模型结果,结果显示该区域5个波段建模得到的RMSEP值均小于全波段建模得到的RMSEP值,而RPD值则大于全波段建模得到的RPD值,说明模型的预测能力得到了提高。对5个波段区间建模得到的RMSEP和RPD值进行比较,结果显示用于HA和总蛋白含量测定的最佳的建模光谱区间均为5951-5361 cm-1。图8为区间5951-5361 cm-1建模得到的模型结果图,图中灰色圆圈代表校正集的样品点,红色菱形代表验证集的样品点,可见样品点较均匀的分布于拟合线的两侧。最终得到用于测定HA和总蛋白含量的模型的Rp2、RMSECV、RMSEP、RPD值分别为0.969、0.530 g/L、0.341 g/L、5.47和0.975、0.501 g/L、0.465 g/L、5.57。
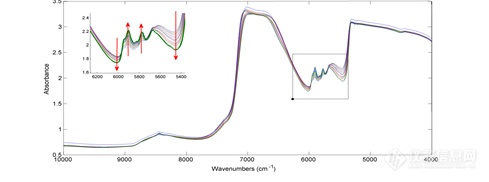
图7 近红外原始光谱及光谱的局部放大图
表3 不同光谱区间建模结果
| 区间 | Rp2 | RMSECV (g/L) | RMSEP (g/L) | RPD |
| HA | 总蛋白 | HA | 总蛋白 | HA | 总蛋白 | HA | 总蛋白 | |
| 全波段 | 0.968 | 0.970 | 0.515 | 0.489 | 0.384 | 0.678 | 4.86 | 3.82 | |
| 6121-5361 cm-1 | 0.967 | 0.968 | 0.539 | 0.496 | 0.350 | 0.525 | 5.33 | 4.93 | |
| 6121-5751 cm-1 | 0.963 | 0.969 | 0.527 | 0.497 | 0.363 | 0.504 | 5.14 | 5.14 | |
| 5951-5361 cm-1 | 0.969 | 0.975 | 0.530 | 0.501 | 0.341 | 0.465 | 5.47 | 5.57 | |
| 5951-5751 cm-1 | 0.962 | 0.971 | 0.519 | 0.481 | 0.369 | 0.506 | 5.06 | 5.12 | |
| 5581-5361 cm-1 | 0.957 | 0.957 | 0.495 | 0.491 | 0.371 | 0.592 | 5.03 | 4.37 | |
| | | | | | | | | | | |

图8 最终模型结果图 (a: HA建模结果 b: 总蛋白建模结果)
3.5.3 白蛋白和总蛋白含量的相关性分析
将参比方法得到的HA和总蛋白含量进行相关性分析,图9为得到的相关性分析结果图,可知两种含量的决定系数(R2)为0.97,说明两种含量之间存在很高的相关性。并且本研究中分别测定了HA和总蛋白含量,蛋白质分子间有相同的化学结构,存在相同的吸收峰位置,因此用于测定HA和总蛋白含量的最佳建模光谱波段均为区间5951-5361 cm-1。
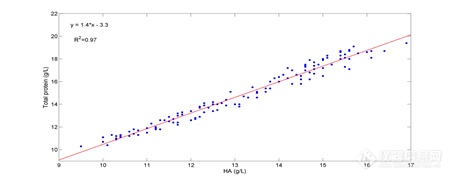
图 9HA和总蛋白含量相关性分析
3.5.4 参考值与预测值比较
将验证集中参考值和预测值进行比较,对建立的PLSR定量分析模型进行评价。图10为参比方法和建立的PLSR定量分析模型方法测得的HA和总蛋白含量的对比图。由图中显示PLSR模型测定的HA和总蛋白的含量值与参比方法得到的值变化趋势一致且非常接近。对验证集样品参考值和预测值间的偏差进行分析,结果见表1-4所示。HA和总蛋白含量NIRS得到的预测值和参考值间的平均偏差为0.259 g/L和0.384 g/L(计算方法见式8),相对平均偏差为2.1%和2.6%(计算方法见式9),说明两种方法结果相差较小,证实了NIRS用于测定醇沉过程中HA和总蛋白含量变化的准确性和有效性。
平均偏差= (8)
相对平均偏差= ×100% (9)
式中yi,actual为参考方法得到的参考值,yi, predicted为NIRS得到的预测值,n为验证集样品数量。
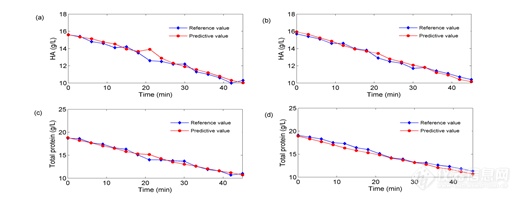
图10 验证集中参考值和预测值对比图 (a: batch 7 HA b: batch 8 HA c: batch 7总蛋白 d: batch 8总蛋白)
表4 验证集样品预测结果评价表
| 指标 | 参考值均值(g/L) | 预测值均值(g/L) | 平均偏差(g/L) | 相对平均偏差(%) |
| HA | 12.9 | 13.0 | 0.26 | 2.1 |
| 总蛋白 | 14.8 | 14.6 | 0.38 | 2.6 |
3.6 重复性考察
选择批次8(验证集)中的2、8、14号样品(分别代表高、中、低的浓度)对模型进行重复性验证,每个样品在10 min内采集10张光谱,然后利用近红外模型计算HA和总蛋白含量值,得到10个预测值的平均值、SD、相对标准偏差(Relative StandardDeviation, RSD)值,结果见表5所示。由表中可知,采用所建立的模型测定的HA和总蛋白的含量值差别较小,RSD值均在1.5%以下,证明了模型有很好的重复性,可以用于醇沉过程中HA和总蛋白含量的测定。
表5 模型重复性考察结果
| 编号 | HA (g/L) | 总蛋白 (g/L) |
| 2号 | 8号 | 14号 | 2号 | 8号 | 14号 |
| 1 | 15.3 | 12.7 | 10.3 | 17.6 | 14.0 | 11.1 |
| 2 | 15.5 | 13.2 | 10.7 | 18.0 | 14.5 | 11.5 |
| 3 | 15.2 | 12.8 | 10.4 | 17.7 | 14.2 | 11.2 |
| 4 | 15.3 | 12.9 | 10.5 | 17.8 | 14.2 | 11.3 |
| 5 | 15.3 | 13.0 | 10.6 | 17.8 | 14.3 | 11.4 |
| 6 | 15.3 | 13.0 | 10.6 | 17.9 | 14.4 | 11.4 |
| 7 | 15.3 | 13.2 | 10.7 | 17.9 | 14.5 | 11.4 |
| 8 | 15.3 | 13.2 | 10.7 | 17.9 | 14.5 | 11.5 |
| 9 | 15.4 | 13.2 | 10.7 | 18.0 | 14.5 | 11.5 |
| 10 | 15.4 | 13.1 | 10.7 | 18.0 | 14.5 | 11.5 |
| 平均值 | 15.3 | 13.0 | 10.6 | 17.9 | 14.4 | 11.4 |
| SD | 0.10 | 0.18 | 0.15 | 0.13 | 0.19 | 0.15 |
| RSD (%) | 0.66 | 1.4 | 1.5 | 0.74 | 1.3 | 1.3 |
| | | | | | | | | |
4讨论和结论
本研究中利用NIRS结合化学计量学对HA生产过程中的FI+II+III上清液醇沉过程进行监测,以实现醇沉环节的过程控制。首先,实验室条件下模拟11批醇沉过程,其中包括8批正常条件的醇沉过程和3批异常条件的醇沉过程。然后,以一阶导数SG15点平滑和mean center的预处理方法对原始近红外光谱进行处理,以提高光谱的质量。本研究中建立了醇沉过程监测的定性模型和含量测定的定量模型:(1)以6个正常批次作为校正集,建立用于过程监测的PCA定性模型, PC1得分图作为生产过程控制图,以2个正常批次和3个异常批次作为验证集对模型的过程监测能力和错误诊断能力进行评价,结果显示建立的模型能够有效的对过程进行监测,实现醇沉过程中的错误判断;(2)以6个正常批次作为校正集,5951-5361 cm-1作为建模光谱区间,建立醇沉过程中HA和总蛋白含量测定的定量分析模型。以2个正常批次的样品作为验证集,对模型的预测能力进行评价,最终得到用于测定HA和总蛋白的模型的Rp2、RMSECV、RMSEP、RPD值分别为0.969、0.530 g/L、0.341 g/L、5.47和0.975、0.501 g/L、0.465 g/L、5.57。结果显示模型有较好的预测能力,能够实现醇沉过程中的HA和总蛋白含量的快速测定。
在定性模型的建立中,为了考察所建立模型的过程监测和错误诊断能力,实验中进行了3个异常批次的醇沉研究,分别模拟了恒流泵停止工作、加醇速度出现异常、搅拌出现异常的情况。另外,在模型的建立中由于区间7093-6576 cm-1和5334-4000 cm-1光谱吸收达到饱和,作为异常光谱区间去除。结果显示建立的定性模型可有效的对过程进行监测和错误进行诊断。
在定量模型的建立中,为有效的去除无关信息变量的干扰并同时简化模型,首先对loading值进行分析,结果显示区间波段6100-5350 cm-1变异程度较大,可能包含更多的光谱信息。对该区间进行进一步的分析发现,用于测定HA和总蛋白的最佳光谱区间均为5951-5361 cm-1,通过一级数据的相关性分析发现R2达到0.97,有着很高的相关性,很好的解释了有着相同最佳建模区间的原因。
同时,本实验还存在一定的局限性。所有的样品的取得均在实验室条件下进行模拟得到,和大生产中的条件会存在一定的差别。所以本研究对生产中的过程控制提供了重要的解决思路,尚不能直接用于过程的在线监测和控制中。为实现醇沉过程的NIRS在线监测和HA以及总蛋白的含量的过程测定,促进生产过程的革新,最大程度的提高产品收率和产品质量,有效的样品收集和预处理系统、近红外光谱采集系统、模型的更新和维护等,还需要进行进一步的研究和探索。
参考文献
倪道明. 血液制品 (第三版) . 北京: 人民卫生出版社, 2013.
Kistler P, Nitschmann H. Large scale production of human plasmafractions. Eight years experience with the alcohol fractionation procedure ofNitschmann, Kistler and Lergier .Vox Sang, 1962, 7: 414-424.
刘欣晏. 人血白蛋白连续流压滤工艺研究. 山东大学, 2008.
杨海龙, 臧恒昌, 胡甜, 等. 近红外漫反射光谱法对不同产地山楂的定性鉴别和定量分析. 药物分析杂志, 2014, 34(3): 396-401.
Li L, Ding B, Yang Q, et al. The relevance study of effectiveinformation between near infrared spectroscopy and chondroitin sulfate inethanol precipitation process . JInnov Opt Heal Sci, 2014, 07(06): 1450022.
Wang P, Zhang H, Yang H, et al. Rapid determination of majorbioactive isoflavonoid compounds during the extraction process of kudzu(Pueraria lobata) by near-infrared transmission spectroscopy . Spectrochim Acta A Mol BiomolSpectrosc, 2015, 137: 1403-1408.
Zhang XB, Feng YC, Hu CQ. Feasibility and extension ofuniversal quantitative models for moisture content determination in beta-lactampowder injections by near-infrared spectroscopy . Anal Chim Acta, 2008, 630(2): 131-140.
Rodkey FL. Direct Spectrophotometric Determination of Albuminin Human Serum . Clin Chem, 1965,11: 478-87.
Goa J. A micro biuret method for protein determination;determination of total protein in cerebrospinal fluid . Scand J Clin Lab Invest, 1953, 5(3): 218-222.











